
StanとRでベイズ統計モデリングをPyMC Ver.5で写経~第8章「8.3 非線形モデルの階層モデル」
第8章「階層モデル」
書籍の著者 松浦健太郎 先生
この記事は、テキスト第8章「階層モデル」の8.3節「非線形モデルの階層モデル」の PyMC5写経 を取り扱います。
予測分布の95%信用区間が観測値を包み込む感じがいいですね🍓
はじめに
StanとRでベイズ統計モデリングの紹介
この記事は書籍「StanとRでベイズ統計モデリング」(共立出版、「テキスト」と呼びます)のベイズモデルを用いて、PyMC Ver.5で「実験的」に写経する翻訳的ドキュメンタリーです。
テキストは、2016年10月に発売され、ベイズモデリングのモデル式とプログラミングに関する丁寧な解説とモデリングの改善ポイントを網羅するチュートリアル「実践解説書」です。もちろん素晴らしいです!
「アヒル本」の愛称で多くのベイジアンに愛されてきた書籍です!
テキストに従ってStanとRで実践する予定でしたが、RのStan環境を整えることができませんでした(泣)
そこでこのシリーズは、テキストのベイズモデルをPyMC Ver.5に書き換えて実践します。
引用表記
この記事は、出典に記載の書籍に掲載された文章及びコードを引用し、適宜、掲載文章とコードを改変して書いています。
【出典】
「StanとRでベイズ統計モデリング」初版第13刷、著者 松浦健太郎、共立出版
記事中のイラストは、「かわいいフリー素材集いらすとや」さんのイラストをお借りしています。
ありがとうございます!
PyMC環境の準備
Anacondaを用いる環境構築とGoogle ColaboratoryでPyMCを動かす方法について、次の記事にまとめています。
「PyMCを動かすまでの準備」章をご覧ください。
8.3 非線形モデルの階層モデル
インポート
### インポート
# 数値・確率計算
import pandas as pd
import numpy as np
# PyMC
import pymc as pm
import pytensor.tensor as pt
import arviz as az
# 描画
import matplotlib.pyplot as plt
import seaborn as sns
plt.rcParams['font.family'] = 'Meiryo'
# ワーニング表示の抑制
import warnings
warnings.simplefilter('ignore')データの読み込み
サンプルコードのデータを読み込みます。
### データの読み込み ◆データファイル8.3 data-conc-2.txt
# PersonID:患者ID,
# 薬の血中濃度Y[mg/mL]:Time1:投与から1時間後のY,・・・, Time24:投与から24時間後のY
data = pd.read_csv('./data/data-conc-2.txt')
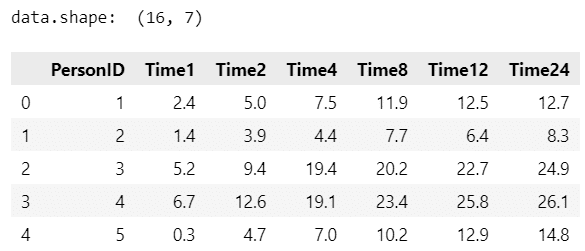
print('data.shape: ', data.shape)
display(data.head())【実行結果】

データの基本統計量を確認します。
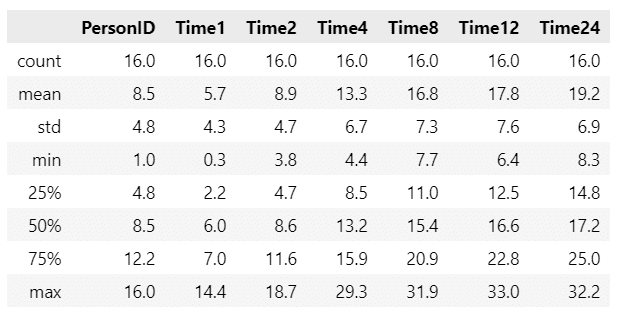
### 基本統計量の表示
data.describe().round(1)【実行結果】
患者別の折れ線グラフを描画してみましょう。
### 折れ線グラフの描画
# カラーマップの取得(線の色分けに利用)
cmap = plt.get_cmap('tab20')
# 描画領域の設定
plt.figure(figsize=(10, 5))
ax = plt.subplot()
# 16人分描画を繰り返し処理
for i in range(len(data)):
# 折れ線グラフの描画
ax.plot([1, 2, 4, 8, 12, 24], data.iloc[i, 1:], color=cmap(i/20),
label=data.iloc[i, 0])
# 修飾
ax.set(xticks=[1, 2, 4, 8, 12, 24], xlabel='Time[hour]',
ylabel='Y : 血中濃度[mg/mL]')
ax.grid(lw=0.5)
ax.legend(bbox_to_anchor=(1, 1), title='PersonID');【実行結果】


8.3.1 解析の目的とデータの分布の確認
時系列折れ線グラフとヒストグラムで可視化します。
テキスト図8.7の左右のグラフに相当します。
まず左側の「患者別の経過時間TimeとYの関係」です。
### 患者ID別折れ線グラフの描画 ◆図8.7左
# 描画領域の設定
fig, ax = plt.subplots(4, 4, figsize=(10, 8), sharex=True, sharey=True)
# 16人分の描画を繰り返し処理
for i in range(len(data)):
# axesの値を算出
pos = divmod(i, 4)
# 折れ線グラフの描画
ax[pos].plot([1, 2, 4, 8, 12, 24], data.iloc[i, 1:], '-o')
# 修飾
ax[pos].set(title=f'ID:{data.iloc[i, 0]}', xticks=[1, 2, 4, 8, 12, 24])
ax[pos].grid(lw=0.5)
# 全体修飾
fig.supxlabel('Time[hour]')
fig.supylabel('Y : 血中濃度[mg/mL]')
plt.tight_layout();【実行結果】

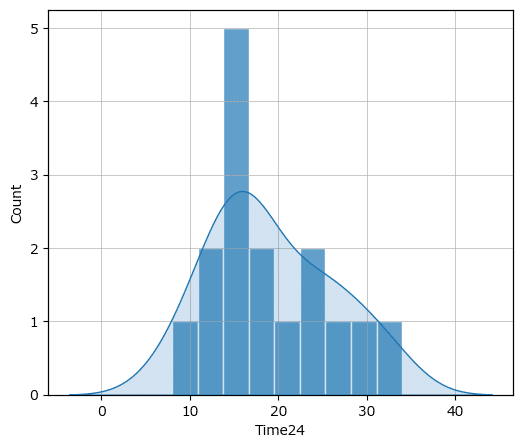
続いて図8.7右の「最終時点におけるYのヒストグラム」です。
### 最終時点のYのヒストグラムの描画 ◆図8.7右
# binsの設定
bins = np.linspace(8, 34, 10)
# 描画領域の設定
fig, ax = plt.subplots(figsize=(6, 5))
# twinxの設定(KDEプロットの軸用)
twinax = ax.twinx()
# ヒストグラムの描画
sns.histplot(data['Time24'], bins=bins, ec='white', alpha=0.7, ax=ax)
# KDE曲線の描画
sns.kdeplot(data['Time24'], fill=True, alpha=0.2, ax=twinax)
# twinxのaxesの縦軸をテキストに寄せた上で表示消去
twinax.set(ylim=[0, 0.1], ylabel='', yticks=[])
ax.grid(lw=0.5);【実行結果】

8.3.4 Stanで実装
PyMCでモデル式8-7を実装します。
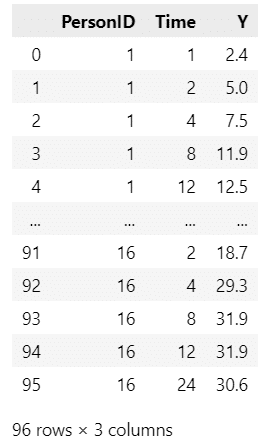
モデルに用いるデータの縦持ち化です。
### データ前処理 縦持ちに変換
data2 = data.copy()
data2.columns = ['PersonID', 1, 2, 4, 8, 12, 24]
data2 = data2.melt(id_vars=['PersonID'], value_vars=[1, 2, 4, 8, 12, 24],
var_name='Time', value_name='Y')
data2 = data2.sort_values(['PersonID', 'Time']).reset_index(drop=True)
data2['Time'] = data2['Time'].astype(int)
display(data2)【実行結果】
予測分布の算出に用いるTime_newを作成します。
### 予測分布の算出に用いるTime_newの作成
# 時間間隔の設定
T_new = 60
# 時間の値の設定:0~24を60分割
Time_new = np.tile(np.linspace(0, 24, T_new), len(data))
print('Time_new.shape:', Time_new.shape)
print(Time_new[:120])
# 患者IDの値の設定
person_idx_new = np.repeat(np.arange(0, 16), T_new)
print('\nperson_idx_new.shape:', person_idx_new.shape)
print(person_idx_new[:120])【実行結果】

続いてモデルの定義です。
mu に曲線$${a\{1-\exp(-bt)\}}$$を含んでいます。
### モデルの定義 ◆モデル式8-7 model8-7.stan
with pm.Model() as model:
### データ関連定義
## coordの定義
model.add_coord('data', values=data2.index, mutable=True)
model.add_coord('person', values=data['PersonID'].sort_values(),
mutable=True)
model.add_coord('dataNew', values=range(len(Time_new)), mutable=True)
## dataの定義
# 目的変数 Y
Y = pm.ConstantData('Y', value=data2['Y'].values, dims='data')
# 説明変数 time
time = pm.ConstantData('time', value=data2['Time'].values, dims='data')
# 患者IDインデックス personIdx 0始まり
personIdx = pm.ConstantData('personIdx', value=data2['PersonID'].values - 1,
dims='data')
# 予測用説明変数 timeNew
timeNew = pm.ConstantData('timeNew', value=Time_new, dims='dataNew')
# 予測用患者IDインデックス personIdxNew 0始まり
personIdxNew = pm.ConstantData('personIdxNew', value=person_idx_new,
dims='dataNew')
### 事前分布
a0 = pm.Uniform('a0', lower=-10, upper=10)
b0 = pm.Uniform('b0', lower=-10, upper=10)
sigmaA = pm.Uniform('sigmaA', lower=0, upper=10)
sigmaB = pm.Uniform('sigmaB', lower=0, upper=10)
sigmaY = pm.Uniform('sigmaY', lower=0, upper=10)
logA = pm.Normal('logA', mu=a0, sigma=sigmaA, dims='person')
logB = pm.Normal('logB', mu=b0, sigma=sigmaB, dims='person')
### mu
a = pm.Deterministic('a', pt.exp(logA), dims='person')
b = pm.Deterministic('b', pt.exp(logB), dims='person')
mu = pm.Deterministic('mu', a[personIdx]*(1 - pt.exp(-b[personIdx]*time)),
dims='data')
### 尤度関数
obs = pm.Normal('obs', mu=mu, sigma=sigmaY, observed=Y, dims='data')
### 計算値
muNew = pm.Deterministic(
'muNew',
a[personIdxNew]*(1 - pt.exp(-b[personIdxNew]*timeNew)),
dims='dataNew')
yNew = pm.Normal('yNew', mu=muNew, sigma=sigmaY, dims='dataNew')モデルの定義内容を見ます。
### モデルの表示
model【実行結果】

### モデルの可視化
pm.model_to_graphviz(model)【実行結果】


MCMCを実行します。
### 事後分布からのサンプリング 2分
with model:
idata = pm.sample(draws=1000, tune=1000, chains=4, target_accept=0.9,
nuts_sampler='numpyro', random_seed=1234)【実行結果】省略
Pythonで事後分布からのサンプリングデータの確認を行います。
Rhatの確認から。
テキストの収束条件は「chainを3以上にして$${\hat{R}<1.1}$$のとき」です。
### r_hat>1.1の確認
# 設定
idata_in = idata # idata名
threshold = 1.03 # しきい値
# しきい値を超えるR_hatの個数を表示
print((az.rhat(idata_in) > threshold).sum())【実行結果】
収束条件を満たしています。

事後統計量を表示します。
### 推論データの要約統計情報の表示
var_names = ['a0', 'b0', 'sigmaA', 'sigmaB', 'sigmaY', 'logA', 'logB']
pm.summary(idata, hdi_prob=0.95, var_names=var_names, round_to=3)【実行結果】
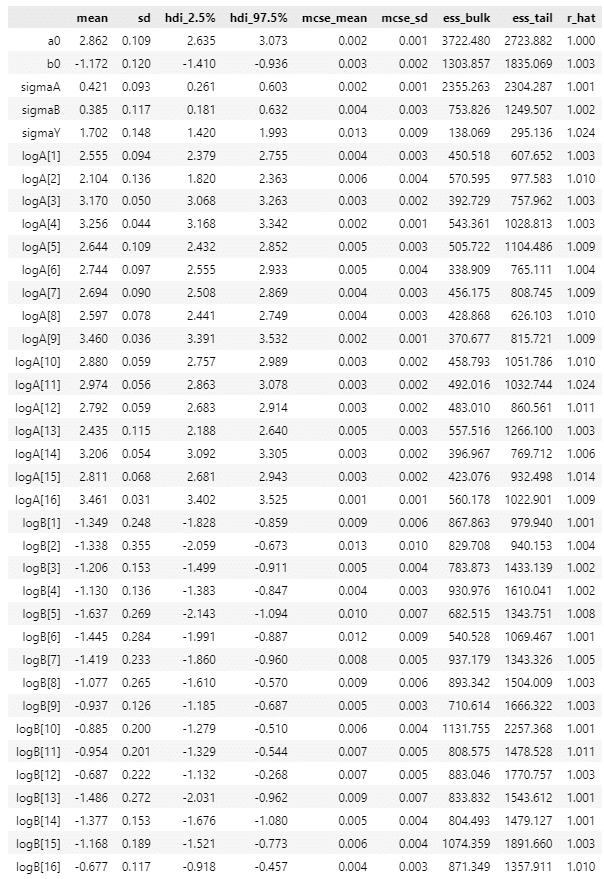
### 推論データの要約統計情報の表示
var_names = ['a', 'b', 'mu']
pm.summary(idata, hdi_prob=0.95, var_names=var_names, round_to=3)【実行結果】

トレースプロットを描画します。
### トレースプロットの表示
var_names = ['a0', 'b0', 'sigmaA', 'sigmaB', 'sigmaY', 'logA', 'logB',
'a', 'b', 'mu']
pm.plot_trace(idata, compact=True, var_names=var_names)
plt.tight_layout();【実行結果】


8.3.5 推定結果の解釈
事後分布の要約統計量を算出します。
算出関数を定義します。
### mean,sd,2.5%,25%,50%,75%,97.5%パーセンタイル点をデータフレーム化する関数の定義
def make_stats_df(y):
probs = [2.5, 25, 50, 75, 97.5]
columns = ['mean', 'sd'] + [str(s) + '%' for s in probs]
quantiles = pd.DataFrame(np.percentile(y, probs, axis=0).T, index=y.columns)
tmp_df = pd.concat([y.mean(axis=0), y.std(axis=0), quantiles], axis=1)
tmp_df.columns=columns
return tmp_df要約統計量を算出します。
### パラメータの要約統計量を確認
vars = ['a0', 'b0', 'sigmaA', 'sigmaB', 'sigmaY']
param_samples = idata.posterior[vars].to_dataframe().reset_index(drop=True)
display(make_stats_df(param_samples).round(2))【実行結果】

患者ごとの予測分布を描画します。
テキスト図8.8に相当します。
### 予測分布の描画 ◆図8.8
# 予測分布データの取り出し
y_new_samples = (idata.posterior.yNew.stack(sample=('chain', 'draw'))
.data.reshape(16, 60, 4000))
# 描画領域の設定
fig, ax = plt.subplots(4, 4, figsize=(10, 8), sharex=True, sharey=True)
# 16人分の描画を繰り返し処理
for i in range(len(y_new_samples)):
# 予測分布の中央値と95%CIを算出
y_new_median = np.median(y_new_samples[i], axis=1)
y_new_95ci = np.quantile(y_new_samples[i], q=[0.025, 0.975], axis=1)
# axesの値を算出
pos = divmod(i, 4)
# x軸の値の設定
xval = np.linspace(0, 24, 60)
# Yの観測値の描画
ax[pos].plot([1, 2, 4, 8, 12, 24], data.iloc[i, 1:], 'o', color='tab:blue')
# Yの予測分布・中央値の折れ線グラフの描画
ax[pos].plot(xval, y_new_median)
# Yの予測分布・95%CIの折れ線グラフの描画
ax[pos].fill_between(xval, y_new_95ci[0], y_new_95ci[1], color='tab:blue',
alpha=0.2)
# 修飾
ax[pos].set(title=f'ID:{data.iloc[i, 0]}', xticks=[1, 2, 4, 8, 12, 24])
ax[pos].grid(lw=0.5)
# 全体修飾
fig.supxlabel('Time[hour]')
fig.supylabel('Y : 血中濃度[mg/mL]')
plt.tight_layout();【実行結果】
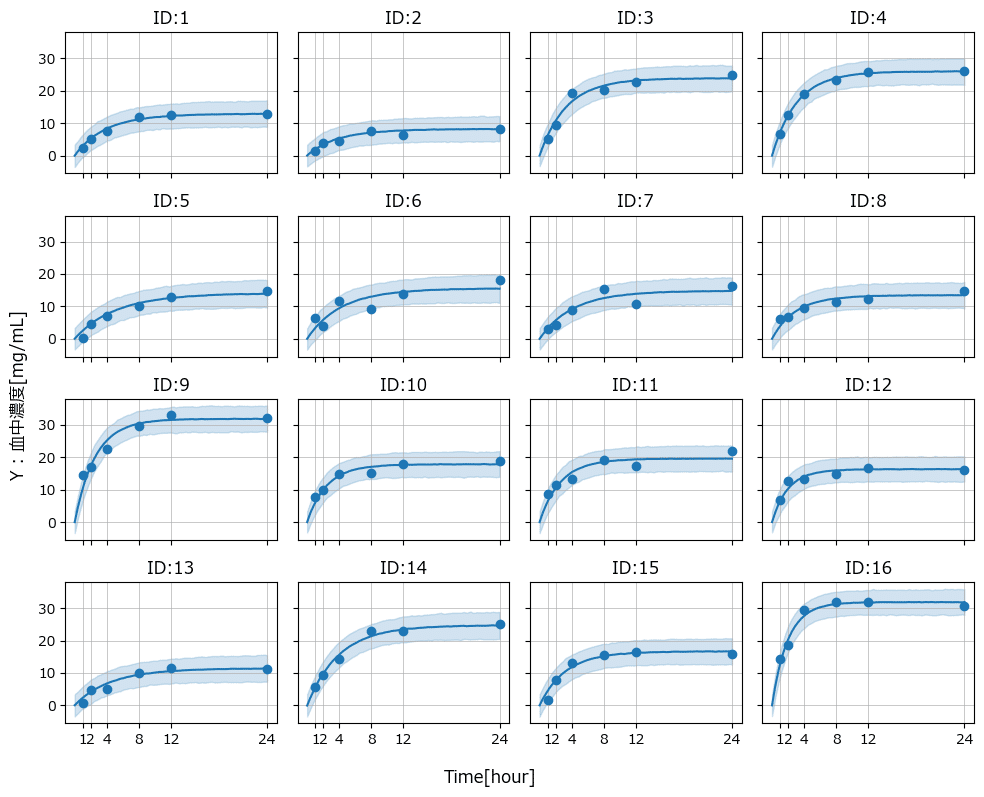
8.3 節は以上です。

シリーズの記事
次の記事
前の記事
目次
ブログの紹介
note で7つのシリーズ記事を書いています。
ぜひ覗いていってくださいね!
1.のんびり統計
統計検定2級の問題集を手がかりにして、確率・統計をざっくり掘り下げるブログです。
雑談感覚で大丈夫です。ぜひ覗いていってくださいね。
統計検定2級公式問題集CBT対応版に対応しています。
Python、EXCELのサンプルコードの配布もあります。
2.実験!たのしいベイズモデリング1&2をPyMC Ver.5で
書籍「たのしいベイズモデリング」・「たのしいベイズモデリング2」の心理学研究に用いられたベイズモデルを PyMC Ver.5で描いて分析します。
この書籍をはじめ、多くのベイズモデルは R言語+Stanで書かれています。
PyMCの可能性を探り出し、手軽にベイズモデリングを実践できるように努めます。
身近なテーマ、イメージしやすいテーマですので、ぜひぜひPyMCで動かして、一緒に楽しみましょう!
3.実験!岩波データサイエンス1のベイズモデリングをPyMC Ver.5で
書籍「実験!岩波データサイエンスvol.1」の4人のベイジアンによるベイズモデルを PyMC Ver.5で描いて分析します。
この書籍はベイズプログラミングのイロハをざっくりと学ぶことができる良書です。
楽しくPyMCモデルを動かして、ベイズと仲良しになれた気がします。
みなさんもぜひぜひPyMCで動かして、一緒に遊んで学びましょう!
4.楽しい写経 ベイズ・Python等
ベイズ、Python、その他の「書籍の写経活動」の成果をブログにします。
主にPythonへの翻訳に取り組んでいます。
写経に取り組むお仲間さんのサンプルコードになれば幸いです🍀
5.RとStanではじめる心理学のための時系列分析入門 を PythonとPyMC Ver.5 で
書籍「RとStanではじめる心理学のための時系列分析入門」の時系列分析をPythonとPyMC Ver.5 で実践します。
この書籍には時系列分析のテーマが盛りだくさん!
時系列分析の懐の深さを実感いたしました。
大好きなPythonで楽しく時系列分析を学びます。
6.データサイエンスっぽいことを綴る
統計、データ分析、AI、機械学習、Pythonのコラムを不定期に綴っています。
統計・データサイエンス書籍にまつわる記事が多いです。
「統計」「Python」「数学とPython」「R」のシリーズが生まれています。
7.Python機械学習プログラミング実践記
書籍「Python機械学習プログラミング PyTorch & scikit-learn編」を学んだときのさまざまな思いを記事にしました。
この書籍は、scikit-learnとPyTorchの教科書です。
よかったらぜひ、お試しくださいませ。
最後までお読みいただきまして、ありがとうございました。
この記事が参加している募集
この記事が気に入ったらサポートをしてみませんか?
