
Rの使い方:医療統計入門
R初心者に簡単に説明
統計ソフトって高いですよね。
さらにJMPなどでは有料なのにできない解析があります。
RやEZRなら無料で使用することができますし、望んだ解析のほとんどをすることができます。
Rはとても優れた統計ソフトですが、コードを理解する必要がありとっつきにくいのも事実です。
私もRに慣れるまで苦労しました。
特に私は医療系の研究をしているので、きれいなデータを使えることは少ないです。
図書館の解説本では「○○分布とは?」、「○○分布の作り方」などから解説が始まり、正直そこじゃない感が強かったです。
そこで、医療系の研究に特化したRの解説をしようと思いました。
なぜなら私の周りでRを使っている人が少なく、仲間が欲しかったからです。
ここでは、数か月前から使い始めた私が引っ掛かったポイントなどを踏まえ、なるべくわかりやすくRについて解説していきます。
Working Directoryの設定
まず、Working Directoryの設定をします。
方法は2つあります。
①クリックでの設定
RStudio右側の中段にある『Files』をクリックします。
使いたいデータが入っているファイルを開きます。
『More▼』をクリックします。
『Set As Working Directory』をクリックすれば完了です。

②コードを書く
setwd(" ")でWorking Directoryを設定します。
setwd("C:\Users\ユーザー名\ファイル名\ファイル名")使いたいデータが入っているフォルダを開きます。

赤い矢印で示したフォルダマークをクリックすると、赤い四角で示したアドレスバーにフォルダのパスが表示されます。
フォルダのパスをコピーして、setwd("ここ ")に貼り付けます。
その際『¥』を『\』に変更し実行します。
これでWorking Directoryの設定は完了です。
ここではWorking Directoryの設定の方法を2つ紹介しました。
無事スタートラインに立てましたね!!
データの読み込み
Rではcsvファイルを用意する必要があります。
csvファイルの作り方は、エクセルを保存する際にWindowsならば『csv(コンマ区切り)』を選択するだけです。

ここでは一つ例としてcsvファイルを共有します。
csvファイルの読み込みは『read.csv("ファイル名.csv")』で行います。
先ほどのcsvファイルを読み込むには
read.csv("Rexample.csv")と入力し実行します。
このあと使いやすいように『df』の中にcsvの入れておきます。
このdfは任意で決めるので、abcでもrenshuでもなんでもよいです。
df <- read.csv("Rexample.csv")無事に読み込まれたかを確認します。
View(df)を実行します。なお、Vは大文字です。
View(df)
RStudio上でもデータを確認することができました。
いよいよ解析のスタートです!!
2群比較
まず、『tableone』パッケージをインストールしましょう。
パッケージのインストール方法は2つあります。
①ツールを使う
RStudio上部のバーに表示されている『Tools』をクリックし、『Install Packages』を選択します。
Packagesにtableoneを入力し、Installをクリックすれば完了です。
②コードを使う
install.packages("tableone")を実行します。
install.packages("tableone")これでtableoneパッケージを使用することができるようになりました。
使用前にtableoneライブラリを読み込んでおきます。
library(tableone)CreateTableoneという機能で群間比較を行うことができます。
Createtableone(vars = 比較したいもの, strata = 比較するグループ, data = データ名, factorVars = varsに含まれる名義変数)
今回はMaleかFemaleかで、BMI, HT, DMに差があるか解析します。
varsにあたる比較したいものをbaseline(任意の文字列)に入れ込んでおきます。
baseline <- c("BMI", "HT", "DM")varsに含まれる名義変数をbaseline_factor(任意の文字列)に入れ込んでおきます。
baseline_factor <- c("HT", "DM")CreateTableOneを使用します。
table1 <- CreateTableOne(vars = baseline,
strata = "Male",
data =df,
factorVars = baseline_factor)
table1上記を実行すると、患者背景の表が完成します。
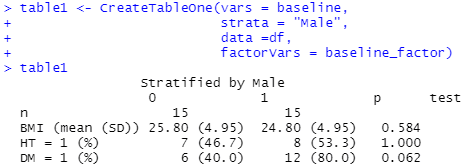
3群比較も同様の方法でできます。
相関係数
相関係数はcor.test()を使用します。
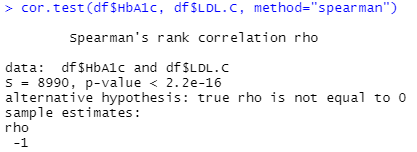
ここでは例としてHbA1cとLDL-Cの相関係数を求めます。
cor.test(df$HbA1c, df$LDL.C, method="spearman")rhoが相関係数です。
相関係数 -1、p値<0.05でHbA1cとLDL-Cに負の相関が有意に認められました。
相関係数を表した図を作成します。
libraryの"ggsci"と"tidyverse"を使用します。
libraryの使い方については、"tableone"の使用については上記を参考にしてください。
library(ggsci)
library(tidyverse)
plt <- ggplot(df, aes(x=HbA1c, y=LDL.C))
plt <- plt + geom_point()
plt <- plt + geom_smooth(method = "lm", se=FALSE)+theme_bw()+xlim(4,9)+ylim(20,200)+labs(x="HbA1c (%)", y="LDL-C (mg/dL)")上記コードではggplotで図を作成しています。

x軸をHbA1c, y軸をLDL-Cとしています。
xlim, ylimで表示する範囲を指定できます。
labsで表示する軸のタイトルを変更できます。
わからなければコピペして自分が使いたいデータに当てはめることをお勧めします。
箱ひげ図
練習用のデータのうち、MaleとBMIの関係を示した箱ひげ図を作成します。
libraryの"tidyverse"を使用します。
library(tidyverse)
g <- ggplot(df,
aes(x = factor(Male), y = BMI))+
labs(x="Male", y="BMI")+
geom_boxplot(width=0.75, fill = c("#4682B4", "#C71585"))+
theme_classic()
plot(g)x軸がMale、y軸がBMIです。
labsで軸のタイトルを変更できます。
geom.boxplotのwidthで箱の横幅、fillで色の指定ができます。

ロジスティック回帰
練習データのEventsに対するMale、HT、DMのリスクを評価します。
glmを使用します。
glm_rslt <- glm (formula = Events ~ Male + HT + DM,
family = binomial(link = "logit"), data = df)上記で代入したglm_rsltを用いて、オッズ比、95%信頼区間、p値を求めます。
coefでオッズ比、confintで95%信頼区間、summaryでp値を求めます。
coef(glm_rslt) #オッズ比 confint(glm_rslt) #95%信頼区間
summary(glm_rslt) #p値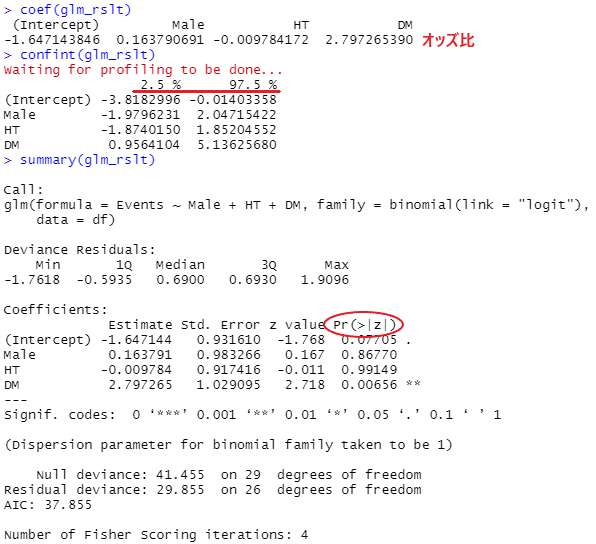
やや発展的ですが、下記コードを使用するときれいにまとまります。
pre <- exp(cbind(OR = coef(glm_rslt), confint(glm_rslt)))#OR and 95%CI
p <- summary(glm_rslt)#p value
p <- p$coefficients
rslt <- cbind(pre, p)
rslt
result <- rslt[-1,c(-4,-5,-6)]
result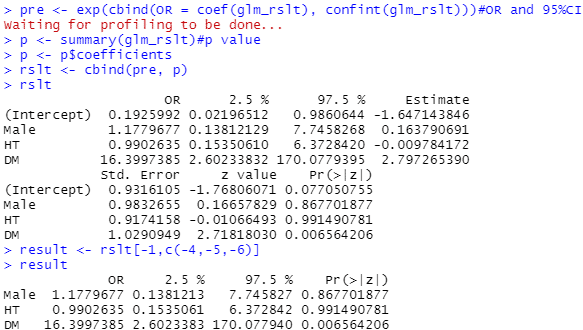
重回帰
重回帰はロジスティック回帰とほぼ同様です。
lin <- glm(Events ~ BMI + SBP + HbA1c, data=df, family = gaussian(link = "identity"))
summary (lin) #p値 coef(lin) #オッズ比 confint(lin) #95%信頼区間
ROC曲線
libraryの"tidyverse"と"pROC"を使用します。
ます、EventsとBMIの関係について評価します。
ROC <- roc(Events ~ BMI, data = df, ci =TRUE)
ROC
r <- ggroc(ROC, size = 1, legacy.axes = TRUE)+
geom_abline(color = "dark grey", size = 0.5)+
theme_bw()+
scale_y_continuous(expand = c( 0, 0 ))+scale_x_continuous(expand = c( 0, 0 ) )
plot(r)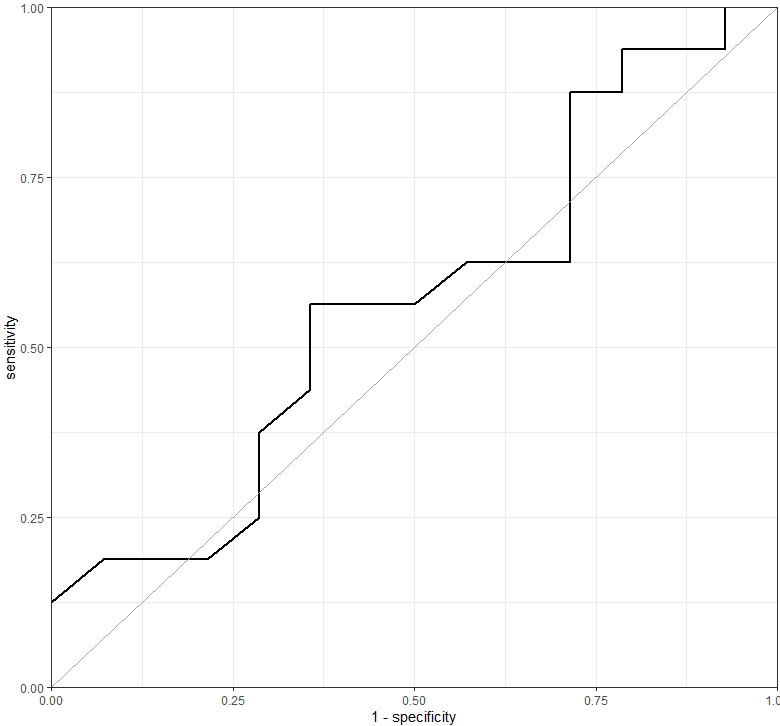
複数のROC曲線を一つの図にまとめることもできます。
ROC <- roc(Events ~ BMI + LDL.C, data = df, ci =TRUE)
ROC
r <- ggroc(ROC, size = 1, legacy.axes = TRUE)+
geom_abline(color = "dark grey", size = 0.5)+
theme_bw()+
scale_y_continuous(expand = c( 0, 0 ))+scale_x_continuous(expand = c( 0, 0 ) )
plot(r)
ここからはROC曲線のパラメーター求め方です。
AUC、感度、特異度、Best cutoff、Youden indexを求めます。
まずはAUCです。
ROC1 <- roc(Events ~ BMI, data = df, ci = TRUE)
ROC1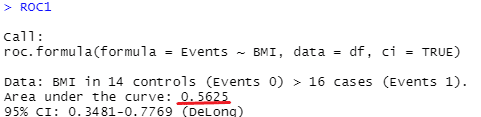
次に感度、特異度、Best cutoff、Youden indexです。
#Youden index
plot(ROC1, identity = TRUE, print.thres = "best", print.thres.best.method = "youden", legacy.axes = TRUE)
#Best cutoff
plot(ROC1, identity = TRUE, print.thres = "best", print.thres.best.method = "closest.topleft", legacy.axes = TRUE)上記を入力するごとに数値が表示されます。

2つのROC曲線のAUCを比較することもできます。
ROC1 <- roc(Events ~ BMI, data = df, ci = TRUE)
ROC2 <- roc(Events ~ LDL.C, data = df, ci = TRUE)
roc.test(ROC1, ROC2)
Eventsに対するBMIとLDL-CのROC曲線をAUCで比較し、p値は0.8772です。
Rで作成した表のExcel (csv)での出力方法
表はExcelデータとして必要な時があります。
R上で作成した表をcsvファイルとして出力することができます。
上記の2群間比較で使用した表を使用します。
library(tableone)
baseline <- c("BMI", "HT", "DM")
baseline_factor <- c("HT", "DM")
table1 <- CreateTableOne(vars = baseline,
strata = "Male",
data =df,
factorVars = baseline_factor)
table1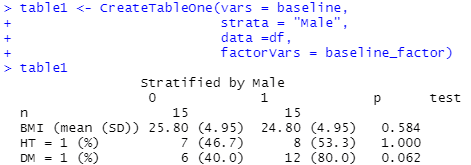
write.csv(table1, "Working Directoryで指定したファイル/表の名前.csv")でcsvファイルに出力されます。
出力先はWorking Directoryで指定したファイルです。
Rでtableの標準偏差を±に表示にする
統計の世界では賛否あるようですが、医学論文では標準偏差を±で表現することがあります。
tableoneではmean (SD)で表示されるため書き換える必要があります。
これを当初の私はcsvに出力してから手作業しておりましたが、手間で仕方がありませんでした。
以下のコードで解決しました。
library(tableone)
baseline <- c("BMI", "HbA1c", "HT", "DM")
baseline_factor <- c("HT", "DM")
table1 <- CreateTableOne(vars = baseline,
strata = "Male",
data =df,
factorVars = baseline_factor)
table1
tab1 <- print(table1)
tab1 <- gsub(" ", "", tab1) #スペースを消す
tab1 <- gsub("\\(", " \\(", tab1) #±に変更する部分にのみスペースを空ける
mean_sd <- c("BMI (mean (SD))", "HbA1c (mean (SD))")
tab1[mean_sd,] <- gsub("\\(", "\\± ", tab1[mean_sd,])
tab1[mean_sd,] <- gsub("\\)", "", tab1[mean_sd,])
table1上記のコードを12行目から解説します。
ここではBMIとHbA1cについて標準偏差を±で表示したいとします。
tableoneで作成した表には余計なスペースがあるので消します。
tab1 <- gsub(" ", "", tab1) #スペースを消す±に変更する部分にのみスペースを空けます。
tab1 <- gsub("\\(", " \\(", tab1) #±に変更する部分にのみスペースを空ける±に変更したい項目をmean_sdに代入します。
mean_sd <- c("BMI (mean (SD))", "HbA1c (mean (SD))")"(" を "±" に変えます。
tab1[mean_sd,] <- gsub("\\(", "\\± ", tab1[mean_sd,])")" を消します。
tab1[mean_sd,] <- gsub("\\)", "", tab1[mean_sd,])これで完成です。
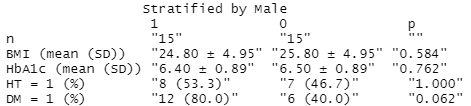
これをcsvで出力すれば手作業不要です。
Rの表を四分位で表示する
医学論文ではしばしば標準偏差での記載が望ましくないデータに出会います。
その際は四分位範囲で記載する必要があります。
ここではSBP, DBPを四分位範囲で表示します。
library(tableone)
baseline <- c("BMI", "HbA1c", "SBP", "DBP","HT", "DM")
baseline_factor <- c("HT", "DM")
baseline_nonnormal <- c("SBP", "DBP") #四分位範囲で記載したいもの
table1 <- CreateTableOne(vars = baseline,
strata = "Male",
data =df,
factorVars = baseline_factor)
table1 <- print(table1, nonnormal = baseline_nonnormal)
table1四分位範囲で記載したい項目をbaseline_nonnormalに代入します。
baseline_nonnormal <- c("SBP", "DBP") #四分位範囲で記載したいものnonnormalにこちらを指定して出力します。
table1 <- print(table1, nonnormal = baseline_nonnormal)
Rで作成した図をパワーポイントで出力する【神機能】
このサイトを参考にしました。
パワーポイントでの編集もできるので、神機能だと思います。
gtsummaryの使い方
libraryのgtsummaryを使用すると一発でtableを出力することができます。
英語の解説が多いので、練習データでgtsummaryを使ってみます。
library(gtsummary)
library(tidyverse)
BASELINE <- df %>%
select(BMI, Male, HT, DM, HbA1c, LDL.C, SBP, DBP, Medication, Events) %>%
mutate(Male = factor(Male, labels = c("Male", "Female"))) %>%
tbl_summary(by = Male, missing = "no",
type = list(c(BMI, HbA1c, LDL.C, SBP, DBP) ~ "continuous",
c(HT, DM, Medication, Events) ~ "dichotomous"),
statistic = list(BMI ~ "{mean} ± {sd}",
HbA1c ~ "{mean} ± {sd}"),
digits = list(c(BMI) ~ 1,
c(HbA1c) ~ 2,
all_dichotomous() ~ c(0,1)),
label = list(BMI ~ "Body mass index",
HT ~ "HT, n (%)",
DM ~ "DM, n (%)",
HbA1c ~ "HbA1c, %",
LDL.C ~ "LDL-C, mg/dL",
SBP ~ "SBP, mmHg",
DBP ~ "DBP, mmHg",
Medication ~ "Medication, n (%)",
Events ~ "Events, n (%)"))%>%
add_p(pvalue_fun = ~style_pvalue(.x, digits = 3)) %>%
bold_p(t = 0.05) %>%
modify_header(label = "**Baseline characteristics**", stat_by = "**{level}**<br>n = {N}") %>%
add_overall(col_label = "**Overall**<br>n = {N}") %>%
modify_spanning_header(c("stat_1", "stat_2") ~ "**性別**") %>%
modify_footnote(update = everything() ~ NA)
BASELINEこれでこうです。
↓
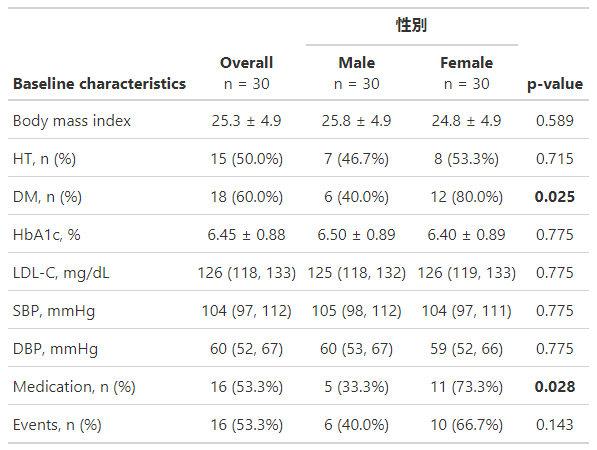
便利すぎます。
以下、使い方です。
selectに使いたい項目を入力します。
select(BMI, Male, HT, DM, HbA1c, LDL.C, SBP, DBP, Medication, Events)data flameではMaleを0か1で表現しているので、mutateでMaleとFemaleに表現し直しています。
mutate(Male = factor(Male, labels = c("Male", "Female")))
ここでは性別による違いを評価しているため、byにMaleを入力します。
typeでは連続変数か名義変数かを指定します。
statisticでは「平均±標準偏差」で記載したい項目を指定します。
digitsでは小数点第何位まで記載するかを指定します。
labelでは表での記載を変更できます。
tbl_summary(by = Male, missing = "no",
type = list(c(BMI, HbA1c, LDL.C, SBP, DBP) ~ "continuous",
c(HT, DM, Medication, Events) ~ "dichotomous"),
statistic = list(BMI ~ "{mean} ± {sd}",
HbA1c ~ "{mean} ± {sd}"),
digits = list(c(BMI) ~ 1,
c(HbA1c) ~ 2,
all_dichotomous() ~ c(0,1)),
label = list(BMI ~ "Body mass index",
HT ~ "HT, n (%)",
DM ~ "DM, n (%)",
HbA1c ~ "HbA1c, %",
LDL.C ~ "LDL-C, mg/dL",
SBP ~ "SBP, mmHg",
DBP ~ "DBP, mmHg",
Medication ~ "Medication, n (%)",
Events ~ "Events, n (%)")) add_pのdigitsでp値の小数点を指定、bold_pで太字にするp値の値を指定します。
add_p(pvalue_fun = ~style_pvalue(.x, digits = 3)) %>%
bold_p(t = 0.05) ヘッダーの修正については**で挟むと太字になります**。
<br>で改行されます。
add_overallでoverallを追加でき、col_labelで表での記載を変更できます。
library(gtsummary)
library(tidyverse)
BASELINE <- df %>%
select(BMI, Male, HT, DM, HbA1c, LDL.C, SBP, DBP, Medication, Events) %>%
mutate(Male = factor(Male, labels = c("Male", "Female"))) %>%
tbl_summary(by = Male, missing = "no",
type = list(c(BMI, HbA1c, LDL.C, SBP, DBP) ~ "continuous",
c(HT, DM, Medication, Events) ~ "dichotomous"),
statistic = list(BMI ~ "{mean} ± {sd}",
HbA1c ~ "{mean} ± {sd}"),
digits = list(c(BMI) ~ 1,
c(HbA1c) ~ 2,
all_dichotomous() ~ c(0,1)),
label = list(BMI ~ "Body mass index",
HT ~ "HT, n (%)",
DM ~ "DM, n (%)",
HbA1c ~ "HbA1c, %",
LDL.C ~ "LDL-C, mg/dL",
SBP ~ "SBP, mmHg",
DBP ~ "DBP, mmHg",
Medication ~ "Medication, n (%)",
Events ~ "Events, n (%)"))%>%
add_p(pvalue_fun = ~style_pvalue(.x, digits = 3)) %>%
bold_p(t = 0.05) %>%
modify_header(label = "**Baseline characteristics**", stat_by = "**{level}**<br>n = {N}") %>%
add_overall(col_label = "**Overall**<br>n = {N}") %>%
modify_spanning_header(c("stat_1", "stat_2") ~ "**性別**") %>%
modify_footnote(update = everything() ~ NA)
BASELINE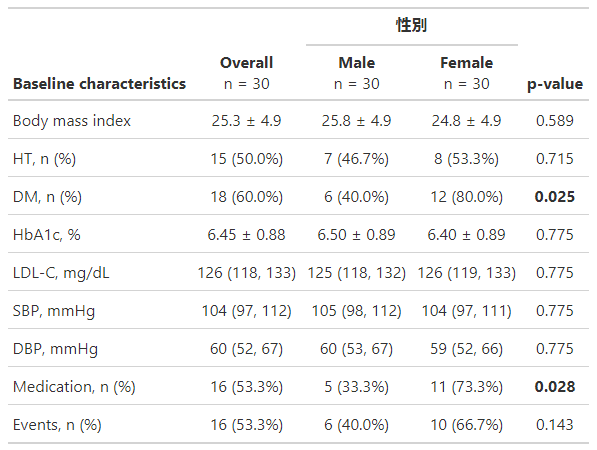
この記事が参加している募集
最後までお読み頂きありがとうございます! よろしければスキ、フォローお願いします!
