
ディープラーニングを治験業務に活用する方法を検討してみました
こんにちは、kudoです。今回は、ディープラーニングを治験業務に活用する方法を検討してみました。結論としては、治験業務ですぐに活用できる使い方まではたどり着きませんでしたが、患者数を予測するためのモデルは作成できそうでした。
きっかけ
ディープラーニングの活用方法を検討することにしたきっかけは2つあります。
1つ目のきっかけ
CRAの頃、施設立ち上げ時期のフィージビリティ調査や施設選定を通じて、一定の候補患者数が見込まれる医療機関を立ち上げても、思った通りに症例が集まらないことが何度かありました。
これは、自分の担当施設だけでなく、同じプロジェクトの他施設、さらには他のプロジェクトでも発生しており、業界内でも議論の対象テーマになることがあるため、重要な課題の1つと考えられます。
このようなことが発生する理由はいくつかありますが、理由の1つに、施設立ち上げ時の候補患者数の予測が現実と乖離していることがあげられます。
結果としてエントリー期間の延長や施設追加などの対応が必要となり、最終的には治験期間延長や治験コストの増加につながることがあるため、候補患者数の予測精度を上げることができると、少しは改善されるかもしれないと思っていました。
2つ目のきっかけ
機械学習やディープラーニングを中心にAIの活用事例が様々な業界において広がっていると感じていました。
機械学習やディープラーニングの長所の1つとして"予測"があげられますが、候補患者数の予測に活用できないものかと考えていました。
この2つのきっかけから、機械学習やディープラーニングを活用して候補患者数の予測を実際に試してみることにしました。なお、機械学習・ディープラーニングについては、まだまだ勉強中ですので、改善点やお気づきの点がありましたら、コメント頂ければと思います。
ゴールの設定
候補患者数の予測で期待されるゴールとしては、以下のように時期により異なると思いますが、特に②は高精度の見積りが要求されますし、①も一定の精度は求められると思います。
①フィージビリティ調査・施設選定時:治験対象疾患の患者数はどれくらいか?
②スクリーニング時:治験の基準に合致する患者数(実施可能症例数)はどれくらいか?
今回は①をターゲットとしますが、公開されているデータに限定すると、医療機関ごとにデータが集計されているDPCデータが活用できそうと考えました。
しかし、公開されているDPCデータを見てみると、今回の取り組みに適したデータセットが少ない状況でした。また、DPCデータ以外の公開データで今回の取り組みに適しているデータを探しましたが、ほとんどない状況でした。
それらのデータを使用することを検討しましたが、前処理に結構な時間が必要となりそうでしたし、今回はお試し程度の感覚でやってみたいという気持ちであったため、他の公開データを探しながら、ゴールを検討することにしました。
その結果、今回のゴールは
・医療機関ごとの治験対象疾患の患者数予測ではなく、都道府県別のがん患者数(がん検診にて発見されたがん患者数)の予測を行うこと
・予測精度を求める前に、そもそも予測モデルの作成が可能か確認すること
としました。
期待されるゴールとは異なりますが、もし今回の試しで予測モデルの作成ができそうであれば、同じ要領で治験対象疾患の患者数予測もできるかもしれないと考えました(もちろん、使用するデータによって異なると思うので一概には言えないですが)。
使用データ
今回使用したデータは、厚生労働省の地域保健・健康増進事業報告に含まれるがん検診のデータになります。具体的なデータはe-Stat(政府統計のポータルサイト)で公開されています。
がん検診のデータでは、がん検診受診者数、要精密検査者数、がんと判断された人数などのデータが市区町村、都道府県、年齢階級別に毎年集計されています。以下の画像が例示になります。
がん種は5種類(胃がん、肺がん、大腸がん、子宮頸がん、乳がん)集計されていますが、今回は胃がんに関するデータを使用しました(選定理由は特にないです)。
今回は、がんと判断される人数を前年・前々年のデータを用いて都道府県別に予測することにしました。なお、具体的な作業を実施するにあたっては、以下の記事・ソースコード、書籍を参考にしました。
現場のAIエンジニアから学ぶ「時系列データの予測モデルの作り方」
Copyright (c) 2019 sakamotomasaki (The MIT License)
詳解 ディープラーニング ~TensorFlow・Kerasによる時系列データ処理~
データの前処理
データの前処理は以下の流れで実施しました。
1. データ整形
2. ライブラリ・データ読込
3. ラグデータ化
4. データスケーリング
5. 学習用データ化
6. データの事前確認
1. データ整形
がん検診のデータは男女別に集計・ファイル化されており、加えて2008年のデータは"個別"と"集団"ごとにそれぞれ集計されています。今回は、総数の予測を実施することしましたので、総数データに加工しました。年齢階級別の内訳データも含まれていますが今回は使用しませんでした。
なお、2016年~2018年のデータは、集計不明なデータが複数存在している影響によりデータ精度が低下していそうであったため、対象データから除外しました。そのため、対象データは2001年~2015年としています。
また、データは市区町村・都道府県別に集計されていますが、市区町村別のデータは欠損値・外れ値が複数存在し処理に時間を要しそうであったこと、都道府県別のデータは欠損値・外れ値がほぼない状態であったことから、今回は都道府県別のデータのみを抽出しました。
データカラムとしては、以下に記載している通り、8つありましたが、がんの疑いのある者、がん以外の疾患であった者、未把握、未受診者のデータは、学習の過程でノイズとなり予測精度が低下する可能性があると思ったため除外しました。

2. ライブラリ・データ読込
必要となるライブラリの読み込みを行います。datetimeは日付操作、numpyは行列計算、pandasはデータ解析、matplotlibはグラフ描画、warningsは警告制御に使用します。また、sklearnからデータの前処理に使用するメソッド(StandardScaler)をインポートします。
%matplotlib inline
import datetime
import numpy as np
import pandas as pd
from sklearn.preprocessing import StandardScaler
import matplotlib.pyplot as plt
import matplotlib.dates as mdates
import matplotlib as mpl
mpl.rcParams['font.family'] = 'IPAexGothic'
plt.style.use('ggplot')
import warnings
warnings.filterwarnings('ignore')その後、整形したデータを読み込みます。
# データファイルの読込
df_all = pd.read_csv('gastric cancer screening_2001-2015_all only.csv', encoding = 'SHIFT_JIS', parse_dates = ['datetime'])
df_all.head()
3. ラグデータ化
ここでは、目的変数と説明変数のデータを作成します。今回は、前年・前々年の全データを説明変数として使用し、今年のがん患者数(目的変数)を予測するようにします。例えば、2015年のがん患者数の予測には、2013年と2014年の全データが使用されています。
# ラグデータ化
df_list1 = []
df_list2 = []
for prefecture in df_all['prefecture'].unique():
df_tmp = df_all[df_all['prefecture'] == prefecture]
df_tmp1 = df_tmp.shift(1)
df_list1.append(df_tmp1)
df_tmp2 = df_tmp.shift(2)
df_list2.append(df_tmp2)
df_all_lag1 = pd.concat(df_list1)
df_all_lag1.rename(columns=lambda x: x + '_lag1', inplace=True)
df_all_lag = pd.concat([df_all.iloc[:, 0:2], df_all.iloc[:, 5], df_all_lag1.iloc[:,2:6]], axis=1)
df_all_lag2 = pd.concat(df_list2)
df_all_lag2.rename(columns=lambda x: x + '_lag2', inplace=True)
df_all_lag = pd.concat([df_all_lag, df_all_lag2.iloc[:,2:6]], axis=1)
df_all_lag.dropna(inplace=True)
df_all_lag.reset_index(drop=True, inplace=True)
df_all_lag.head(5)
4. データスケーリング
続いて、説明変数データをスケーリング(標準化)します。具体的には、説明変数データのスケールの平均を0、分散を1となるよう処理します。これによって、学習時に桁あふれ・桁落ちなどの問題が発生することを予防しています。
# データスケーリング
df_all_list = list()
df_all_list.append(df_all_lag['datetime'])
df_all_list.append(df_all_lag['prefecture'])
df_all_list.append(df_all_lag['patient_all'])
for i in range(3, df_all_lag.shape[1]):
sc = StandardScaler()
a = df_all_lag.iloc[:, i]
a = np.array(a, dtype = 'float32').reshape(-1, 1)
sc.partial_fit(a)
a = sc.transform(a)
a = pd.DataFrame(a, columns = [df_all_lag.columns[i]])
a = df_all_list.append(a)
df_all_sc = pd.concat(df_all_list, 1)
df_all_sc.head()
5. 学習用データ化
ここでは、データを訓練用と検証用に分離し、データの形式をTensorFlowの入力形式に合わせます。今回は、2002年-20012年のデータを訓練用データ、2013年-20015年のデータを検証用データとしています。
# 学習用データ化
dt_train_start = datetime.datetime.strptime('2002-01-01', '%Y-%m-%d')
dt_train_end = datetime.datetime.strptime('2012-01-01', '%Y-%m-%d')
dt_validation_start = datetime.datetime.strptime('2013-01-01', '%Y-%m-%d')
dt_validation_end = datetime.datetime.strptime('2015-01-01', '%Y-%m-%d')
df_train = df_all_sc[((df_all_sc['datetime'] >= dt_train_start) & (df_all_sc['datetime'] <= dt_train_end))]
df_train_rand = df_train.reindex(np.random.permutation(df_train.index)).reset_index(drop = True)
train_x = np.array(df_train_rand.iloc[:, 3:].as_matrix(), dtype='float32')
train_y = np.array(df_train_rand.iloc[:, 2].as_matrix(), dtype='float32')
df_validation = df_all_sc[((df_all_sc['datetime'] >= dt_validation_start) & (df_all_sc['datetime'] <= dt_validation_end))]
df_validation_rand = df_validation.reindex(np.random.permutation(df_validation.index)).reset_index(drop = True)
validation_x = np.array(df_validation_rand.iloc[:, 3:].as_matrix(), dtype='float32')
validation_y = np.array(df_validation_rand.iloc[:, 2].as_matrix(), dtype='float32')6. データの事前確認
モデル作成前に前処理データを確認します。
# 事前のデータ確認(サンプル)
prefecture = '埼玉県'
df_tmp = df_all_sc[df_all_sc['prefecture'] == prefecture]
x = df_tmp['datetime']
y = df_tmp['patient_all']
fig = plt.figure(figsize = (15,3.5))
ax = fig.add_subplot(111)
ax.plot(x, y)
ax.xaxis.set_major_formatter(mdates.DateFormatter('%Y'))
plt.setp(ax.get_xticklabels(), rotation = 90)
ax.set_title(prefecture + ' 目的変数', fontsize = 10)
plt.legend(['patient_all'], bbox_to_anchor = (1, 1), loc = 'upper left')
plt.show()
df_tmp.set_index('datetime', inplace = True)
df_tmp = df_tmp.iloc[:, 2:]
fig = plt.figure(figsize = (15,3.5))
ax = fig.add_subplot(111)
df_tmp.plot(ax = ax)
plt.setp(ax.get_xticklabels(), rotation = 90)
ax.set_title(prefecture + ' 説明変数', fontsize = 10)
plt.legend(bbox_to_anchor = (1, 1), loc = 'upper left')
plt.show()例として埼玉県におけるがん患者数(目的変数)の推移を表示します。

以下は、埼玉県における前年・前々年の全データ(説明変数)の推移です。「_lag1」と記載されているデータが前年のデータ、「_lag2」と記載されているデータが前々年のデータとなります。

モデル作成
前処理が完了したためモデルの作成に進みます。
ディープラーニング実施前のインポート設定
kerasとsklearnから学習に使用するメソッドをディープラーニング実施前にインポートします。
from keras.models import Sequential
from keras.layers.core import Dense, Activation
from keras.layers.normalization import BatchNormalization
from keras.optimizers import Adam
from keras.callbacks import EarlyStopping
from sklearn import datasets
from sklearn.model_selection import train_test_splitディープラーニングのための設定
ディープラーニングを実行するための設定をします。今回はノードを200個、隠れ層を2層としており、活性化関数にはReLUを使用します。評価指標には、平均二乗誤差(Mean Squared Error)を使用します。
# DL設定
n_in = df_all_sc.shape[1] -3
n_hiddens = [200, 200]
n_out = 1
activation = 'relu'
def weight_variable(shape, dtype=None, name=None):
return np.sqrt(2.0 / shape[0]) * np.random.normal(size=shape)
early_stopping = EarlyStopping(monitor='val_loss', patience=10, verbose=2)
model = Sequential()
for i, input_dim in enumerate(([n_in] + n_hiddens)[:-1]):
model.add(Dense(n_hiddens[i], input_dim=input_dim,
kernel_initializer=weight_variable))
model.add(BatchNormalization())
model.add(Activation(activation))
model.add(Dense(n_out, kernel_initializer=weight_variable))
model.add(Activation('relu'))
model.compile(loss='mean_squared_error',
optimizer=Adam(lr=0.001, beta_1=0.9, beta_2=0.999),
metrics=['mae'])ディープラーニング実行
ディープラーニングを実行します。epochsとbatch_sizeは評価指標の動きを見ながら何度か調整し、決定します。
# DL実行
epochs = 100
batch_size = 5
hist = model.fit(train_x, train_y, epochs=epochs,
batch_size=batch_size,
validation_data=(validation_x, validation_y),
callbacks=[early_stopping])学習過程の確認
学習過程を確認します。
# 学習過程の確認
val_loss = hist.history['val_loss']
fig = plt.figure()
plt.plot(range(len(val_loss)), val_loss, label='loss')
plt.xlabel('epochs')
plt.show()
print(model.evaluate(validation_x, validation_y))
モデルを用いた予測データの作成
ここまでの過程で予測モデルが作成されたので、そのモデルを用いて予測データを作成します。
# モデルを用いた予測データの作成
train_x = np.array(df_train.iloc[:, 3:].as_matrix(), dtype='float32')
validation_x = np.array(df_validation.iloc[:, 3:].as_matrix(), dtype='float32')
pred_train = model.predict_proba(train_x)
pred_validation = model.predict_proba(validation_x)
pred = np.concatenate([pred_train, pred_validation])
df_pred = pd.DataFrame(pred, columns=['patient_all_pred'])
df_pred = pd.concat([df_all_sc.iloc[:, 0:3], df_pred], axis=1)
df_pred.head()
予測データの確認(可視化)
作成したモデルから生成された予測データを可視化し、確認します。都道府県別に予測結果を確認するようにします。
# 予測データの確認(可視化)
import matplotlib as mpl
mpl.rcParams['font.family'] = 'IPAexGothic'
for prefecture in df_pred['prefecture'].unique():
df_tmp = df_pred[df_pred['prefecture'] == prefecture]
x = df_tmp['datetime']
y1 = df_tmp['patient_all']
y2 = df_tmp['patient_all_pred']
fig = plt.figure(figsize=(15,3.5))
ax = fig.add_subplot(111)
ax.plot(x, y1)
ax.plot(x, y2)
plt.ylim(0, max(y1.max(), y2.max()) + 50)
ax.xaxis.set_major_formatter(mdates.DateFormatter('%Y'))
plt.setp(ax.get_xticklabels(), rotation=90)
ax.set_title(prefecture + ' : Fact(patient_all), Pred(patient_all_pred)', fontsize=10)
plt.legend(['patient_all', 'patient_all_pred'], bbox_to_anchor=(1, 1), loc='upper left')
plt.show()以下は、山梨県の予測結果です。赤線が実際のがん患者数(patient_all)であり、青線が作成したモデルによるがん患者数予測結果(patient_all_pred')です。青線が赤線と似たような推移をしていることから、今回作成したモデルで山梨県のがん患者数予測はできるように見えます。

しかしながら、他の都道府県の結果を見てみると、予測結果が実際のがん患者数に比べて低く推移しているケースも見られます。以下は東京都の予測結果です。
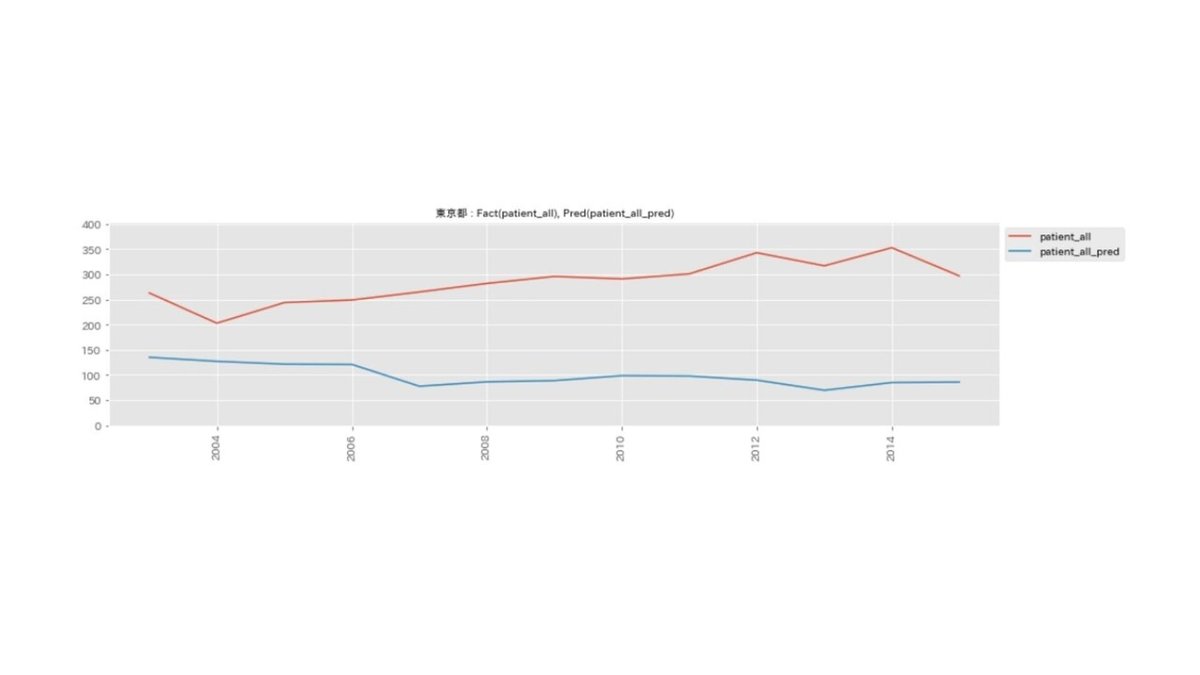
このようなケースが発生している理由としては、データの中に外れ値となるデータが含まれている(今回のケースでは東京都や大阪府など)ためであり、外れ値の予測は難しいことが問題としてあげられます。
そのため、今回のモデルはすべての都道府県に対応して予測が可能とは言えないモデルでした。
外れ値の予測が難しい問題に対しては、東京都だけのデータで学習させ、東京都のみのモデルを作成するなどといった対応方法もありますが、今回は学習用のデータ量が極端に少なくなってしまうことにより、学習が不十分になってしまうことから、実施せずにここまでで終了しました。
考察
今回の取り組みで、がん患者数の予測モデルを作成することは出来ましたが、すべての都道府県の予測が可能とは言えないモデルとなりました。
医療機関ごとの治験対象疾患の患者数を予測するケースでも、医療機関ごとに学習用データの値に大差がある場合(=外れ値が含まれる場合)は、今回と同じように予測精度が低い医療機関が出てくる可能性があるため、データを見ながら既述のような対応方法を検討する必要があるかもしれません。
今回は、公開データ範囲内での取り組みでしたが、DPCデータ・有料データベース・組織で保有するデータなどを組み合わせて今回のような要領で進め、外れ値には既述の対応を実施することで、医療機関ごとの治験対象疾患の患者数予測モデルを作成できる可能性は十分にあると思いました。
なお、今回は実施していませんが、予測モデルの作成が確認できた場合には、次のステップとして予測精度の目標値を定めてチューニングを行うことになると思います。
今回使用したデータに関して言うと、がん検診はがん発見経緯の一部分であるため、一般的に見かける都道府県別のがん患者数はもっと大きくなると思います。
実施環境
以下の環境で実施しました。
OS
Windows10 バージョン 21H1 (OSビルド 19043.1320)
Python
Python 3.7.4 (anaconda 4.10.3)
Python Packages
jupyter 1.0.0
numpy 1.16.5
pandas 0.25.1
scikit-learn 0.21.3
tensorflow 1.14.0
keras 2.3.1
matplotlib 3.1.1
まとめ
・都道府県別のがん患者数予測モデルの作成が可能か試してみた
・予測モデルは作成できたが、都道府県別で予測精度にばらつきがあった
最後に
ディープラーニングを活用するためには学習用データが必要となりますが、公開データのみでは出来ることに限界があるなと感じました。次トライするのであれば、色々なデータを組み合わせて、トライしてみようと思います。
ご質問等がありましたら以下までお問い合わせください
advance10101@gmail.com
この記事が気に入ったらサポートをしてみませんか?
