ファクターXの論文翻訳
SARS-CoV-2に交差反応する機能的に優れた細胞傷害性T細胞のTCRレパートリーの特定
概要
SARS-CoV-2に特異的なCD8+T細胞は少ないが、非被爆者の健康なドナー(UHD)では検出される。しかし、既存のヒトコロナウイルス(HCoV)特異的CD8+ T細胞が、SARS-CoV-2に交差反応する機能的なT細胞に変換されるかどうかは不明である。ここでは、HLA-A24+ UHDの季節性コロナウイルス特異的CD8+ T細胞が認識できる、SARS-CoV-2スパイク領域のHLA-A24高結合の免疫優勢エピトープを同定した。通常、免疫抑制されている血液悪性腫瘍患者では、交差反応性CD8+T細胞がUHDに比べて明らかに減少していた。さらに、HLA-A24+ドナーにおいて、選択されたドミナントエピトープに反応するCD8+T細胞は、多機能性とHCOV間の交差機能性を示すことが示された。さらに、HLA-A24+ドナーから分離したT細胞受容体の交差反応性は、単一細胞レベルでの選択的多様性を示した。以上のことから、免疫優勢エピトープでよく刺激されると、高い機能的アビディティを持つ選択的な既存のCD8+T細胞は、SARS-CoV-2に対して交差反応性を示す可能性がある。
はじめに
SARS-CoV-2(Severe acute respiratory syndrome coronavirus 2)は,COVID-19パンデミックの原因ウイルスである。近年,SARS-CoV-2のスパイク(S)タンパク質を発現させたmRNAベースのワクチンやアデノウイルスベクターを用いたワクチンなど,いくつかのCOVID-19ワクチンが世界的な予防接種プログラムで投与されている1,2,3。SARS-CoV-2ワクチンによって生体を保護するためには、中和抗体(Abs)を作製する必要がある。さらに、SARS-CoV-2をより効率的に抑制するためには、CD4+ T細胞とCD8+ T細胞がSARS-CoV-24対策に重要な役割を果たしている。ワクチンによって誘導される体液性免疫に関する最近の研究では、中和抗体反応の持続時間に関する証拠が示されている5。さらに、SARS-CoV-24の抑制には、中和抗体に加えてCD4+ T細胞やCD8+ T細胞が重要な役割を果たすことが知られている。抗ウイルスCD8+細胞傷害性T細胞は、HIV、CMV、EBV、HSVなどのウイルスが潜伏中であっても、ウイルスに感染した細胞を排除する可能性を持っている6,7,8。抗ウイルスCD8+ T細胞免疫を理解することは、COVID-19の重症度を評価したり、治療戦略を開発するためのバイオマーカーを特定するのに役立つかもしれません。
SARS-CoV-2のORF1にコードされているSタンパク質、膜タンパク質、ヌクレオキャプシドタンパク質などの構造タンパク質や、NSP7やNSP13などの非構造タンパク質の免疫優勢エピトープが予測され、感染者や回復者から検出されている9,10,11,12,13,14,15,16。CD8+T細胞の反応は、病気の重症度と関連している可能性がある17。最近の報告では、SARS-CoV-2に特異的なメモリーCD8+T細胞が、症状発症後6カ月以上経過した回復期患者の50-70%で検出され、その応答エピトープはS、膜、ヌクレオカプシド、ORF3aに存在することが示された18。SARS-CoV-2に特異的なCD8+T細胞を追跡した感染者や回復者を対象とした数多くの研究では、アクティブなエフェクターまたはメモリーCD8+T細胞の重要性が示されている9,10,11,12,13,14,15,16,18。
SARS-CoV-2と交差反応する季節性コロナウイルスに対する既存のCD8+T細胞は、非被爆者である健康なドナー(UHD)の約20%にしか検出されないが、生物学的にはまだ存在する可能性がある16。UHDでは、免疫優勢エピトープの大半が非S領域、ORF1a、ヌクレオカプシドに由来しており、Sタンパク質領域には由来していない11,19。Sタンパク質に対するCD8+T細胞の交差反応性については、十分に検討されていない。UHDのSタンパク質に含まれる交差反応性の免疫優勢エピトープを同定することは、CD8+ T細胞によるワクチン開発の重要な課題の一つです。また、HLA制限下でのTCRの交差反応性は十分に研究されていないため、CD8+T細胞の交差反応性の分子的解析は、その役割を理解するために必要である。
血液悪性腫瘍(HM)患者は、SARS-CoV-2感染症をはじめとする感染症にかかりやすく、固形腫瘍患者よりも高い死亡率を示している20,21,22。HM患者に見られる感染症のリスクの高さだけでなく転帰の悪さも、基礎疾患である血液悪性腫瘍と全身化学療法によって重度の免疫不全に陥っていることが原因であると考えられる。対照的に、B細胞や抗体の産生が極めて低いにもかかわらず、十分なCD8+T細胞数があれば、HM患者のCOVID-19関連死亡率を低下させることができる17。したがって、ワクチンは、CD8+ T細胞や抗体など、免疫系のできるだけ多くの側面に関与する必要があります。ここでは、HLA-A24制限下でSARS-CoV-2交差反応性CD8+T細胞を生成するための、Sタンパク質上の免疫優勢エピトープを探索した。このエピトープを用いて、UHDにおけるエピトープ特異的CD8+T細胞の交差機能性を明らかにした。季節性コロナウイルスに対する交差反応性のメカニズムを単一細胞のTCRレベルで検討した。さらに、HM患者では、このSARS-CoV-2交差反応性CD8+T細胞がUHDのそれよりも減少していることを発見した。
結果
HLA-A24+ UHDにおけるSARS-CoV-2 Sの免疫優勢エピトープの同定
HLA対立遺伝子とCOVID-19との関係は最近報告されている23。そこで我々は、主要HLAの一つであるHLA-A*24:02と、SARS-CoV-2に交差反応するCD8+ T細胞との相互作用を評価した。SARS-CoV-2スパイクタンパク質(SARS-CoV-2 S)のCD8+ T免疫優勢エピトープの標的候補は、NetMHC 4.0を用いて予測した。これらのエピトープの候補となるペプチドを、Sタンパク質の全配列から6つ、対照としてヌクレオカプシドタンパク質から1つ選択して合成した(図1a)9。
図1:HLA-A24陽性の非被爆者健康ドナー(UHD)におけるSARS-CoV-2特異的CD8+T細胞反応。

今回の研究では、2020年初頭(2020年の2月から4月)および2004年から2010年(パンデミック前の時代)に採取した健康なボランティアのPBMCをUHDとして使用しました(補足表1)。また、いずれのボランティアもCOVID-19に罹患しておらず、COVID-19患者と密接に接触したこともありませんでした。また、すべてのUHDは、SARS-CoV-1やMERSの感染歴がありませんでした。さらに、血漿中の抗体産生量を、先行研究でよく使用されている2種類の市販キットを用いて2回測定しました16,19,24。すべてのUHDはSARS-CoV-2に対する抗体が陰性であった。SARS-CoV-2 CD8+ T細胞応答に最適なペプチドを最初にスクリーニングするために、5人のHLA-A24+ UHDを試験した。以前の報告11,13,15に記載されている24時間活性化誘導マーカーアッセイのような短期アッセイは、エフェクターCD8+ T細胞の検出に有用であるが、in-vitro expansionアッセイは、癌に対する既存のCD8+ T細胞やSARS-CoV-2に対する交差反応性CD8+ T細胞を低レベルでも検出するために使用できる14,25,26,27,28。各ペプチドの存在下でPBMCを培養し、21日目にエピトープ特異的IFN-γ産生CD8+T細胞の頻度を評価した。Sタンパク質由来のこれら6つのペプチドは、HLA-A*24:02に高親和性で結合すると予想されたが、これらのうち5つはT細胞応答を誘導せず、Pep#3(QYI)特異的CD8+T細胞数のみが、すべてのUHD由来の細胞で上昇した(図1bおよび補足図1a、b)。陽性反応として、コントロールと比較してT細胞の反応が1.5倍に増加した場合を定義した(補足図1c)。興味深いことに,Pep#3(QYI)は,4種類の季節性コロナウイルスを含む他のコロナウイルスと高い配列相同性を示した(図1c)。Sタンパク質の6つのペプチドはすべてHLA- A*24:02に高い親和性を示すことが予測されたが、5つのペプチドは他の季節性コロナウイルスとは相同性がなかった(補足図1d)。これらの結果から、CD8+T細胞が交差反応を起こしている可能性があり、既存のメモリーCD8+T細胞が免疫反応に関与している可能性が示唆された。
そこで私たちは、UHDのPep#3(QYI)特異的CD8+T細胞に注目しました。このアッセイでは、PBMCからCD8+T細胞を分離し、Pep#3(QYI)をパルス照射した自家PBMCと共培養し、14日目にエピトープ特異的IFN-γ産生CD8+T細胞の頻度を評価しました(図1d)。その結果、UHD参加者18名中15名(83.3%)に抗原特異的CD8+T細胞が存在することがわかりました(図1e, f, g)。HLA-A*24:02+のUHDにおけるPep#3(QYI)特異的T細胞の反応率は、過去の研究11でUHDの20%に見られたものよりも高かった。たとえ頻度が低くても、応答者のほとんどはIFN-γとTNF-αを産生するT細胞を保有していた(図1e, f)。
UHDのPep#3(QYI)特異的CD8+T細胞の多機能性の評価
Pep#3(QYI)特異的CD8+T細胞の多機能性を、サイトカイン産生能、細胞傷害活性、脱顆粒マーカーで評価した。まず、半数のT細胞集団が2種類以上のサイトカイン(IFN-γ、TNF-α、IL-2)を産生していることがわかった(図2a)。一方、IL-10は検出できなかった(補足図2)。次に、Pep#3(QYI)特異的CD8+T細胞株の細胞障害性を評価した(図2b)。Pep#3(QYI)をパルスしたA24/CIRに対しては細胞毒性が検出されたが、パルスしていないA24/CIRに対しては細胞毒性が検出されなかった(図2b)。最後に、細胞毒性分子、すなわちパーフォリンとグランザイムを分泌する細胞の可能性を測定するために使用されるCD107a脱顆粒アッセイを行った。その結果、IFN-γ+ CD8+ T細胞の約80%がCD107aを発現していることがわかった(図2c)。
図2:UHD由来のSARS-CoV-2特異的CD8+T細胞の機能。

Pep#3(QYI)周辺の高親和性領域の検出
次に、Pep#3(QYI)エピトープ周辺のSタンパク質のS2領域(1200-1226)をもとに、11merオーバーラップした15merのペプチドを4種類合成し、この領域の他の免疫優勢エピトープを調べた(図3a)。関連するエピトープはHCOV全体で比較的保存されており、VOC-202012/01、501Y.V2、501Y.V3などのSARS-CoV-2変種株では完全に保存されているため、ペプチドセットはSARS-CoV-2変種株にも利用可能である(補足図3)。15merのペプチドミックスの存在下でUHDのPBMCを培養し、14日目に抗原特異的CD8+T細胞の反応を評価した(図3b)。驚くべきことに、ペプチドを投与したすべてのUHDで、SARS-CoV-2特異的CD8+T細胞が検出された(図3c)。さらに、CD8+T細胞は主に15merのpep#2と#3に反応したが、一部のケースでは15merのpep#1と#4にも反応した(図3d-f)。これらの結果から、HLA-A24+の人にとって15merペプチドミックスはPep#3(QYI)に加えて他の免疫優勢なエピトープを含んでおり、HLA-A24+の人の既存のCD8+ T細胞を検出または拡大するのにより有用であることが示された。
図3:15merのペプチドミックスに反応するCD8+T細胞のデザイン。

SARS-CoV-2 Sの複数のエピトープはHLA-A24+UHDで高い免疫原性を示す
次に、15merペプチドミックスの4種類のエピトープのうち、CD8+ T細胞の活性化に関与する部分を確認するために、Sタンパク質の配列を用いて連続した9merペプチドライブラリを合成した(図4a)。15merペプチドミックスでプライミングした後、各9merペプチドで再刺激し、ペプチド特異的T細胞応答を評価した(図4b)。その結果、9merペプチド#1204〜#1218、特に#1208(Pep#3(QYI))は、15merのpep#2〜#3と同等のCD8+T細胞特異的反応を誘導することがわかった(図4c、d)。そこで、IEBDとNetMHC4.0を用いて、SARS-CoV-2と季節性コロナウイルスのこの領域のインシリコスクリーニングを行い(図4e)、HLA-A24制限下でこれらのCD8+ T細胞に認識される9merペプチドを調べた。その結果、季節性コロナウイルスだけでなく、SARS-CoV-2にも、IEBDやNetMHCのスコアが高い9merエピトープが6種類と8種類発現しており、HLA-A24に結合していることが示唆された(図4e)。このように,in-vitro実験で同定された9merペプチドの一部は,HLA-A24制限下でin-silicoで予測された高親和性ペプチドであることがわかった。しかし、他のペプチドは他のHLAに結合する可能性がある。
図4:15merのペプチドミックスを使ったエピトープのマッピング

HM患者におけるSARS-CoV-2特異的CD8+T細胞の解析
我々は、病気の進行や化学療法に伴う免疫細胞の機能低下によりCOVID-19の影響を受けやすいハイリスクグループであるHLA-A24+のHM患者(補足表2)において、Pep#3(QYI)がエピトープ特異的CD8+T細胞を誘導できるかどうかを調べた。また,本研究で募集したHM患者は全員,SARS-CoV-2に特異的なIgGが陰性であることを確認した(補足表2)。Pep#3(QYI)特異的CD8+T細胞は、HM患者の14.8%(27人中4人)に認められ、Pep#3(QYI)特異的CD8+T細胞の誘導率は、HM患者の方がUHDよりも有意に低かった(p < 0.01; Fisher's/Chi-square test)(図5a-c)。これは、季節性コロナウイルスに対するSARS-CoV-2交差反応CD8+T細胞のレベルが、HM患者ではUHDよりも低いことを示していた。
図5:HLA-A*24:02陽性の血液悪性腫瘍(HM)患者におけるSARS-CoV-2特異的CD8+T細胞反応。
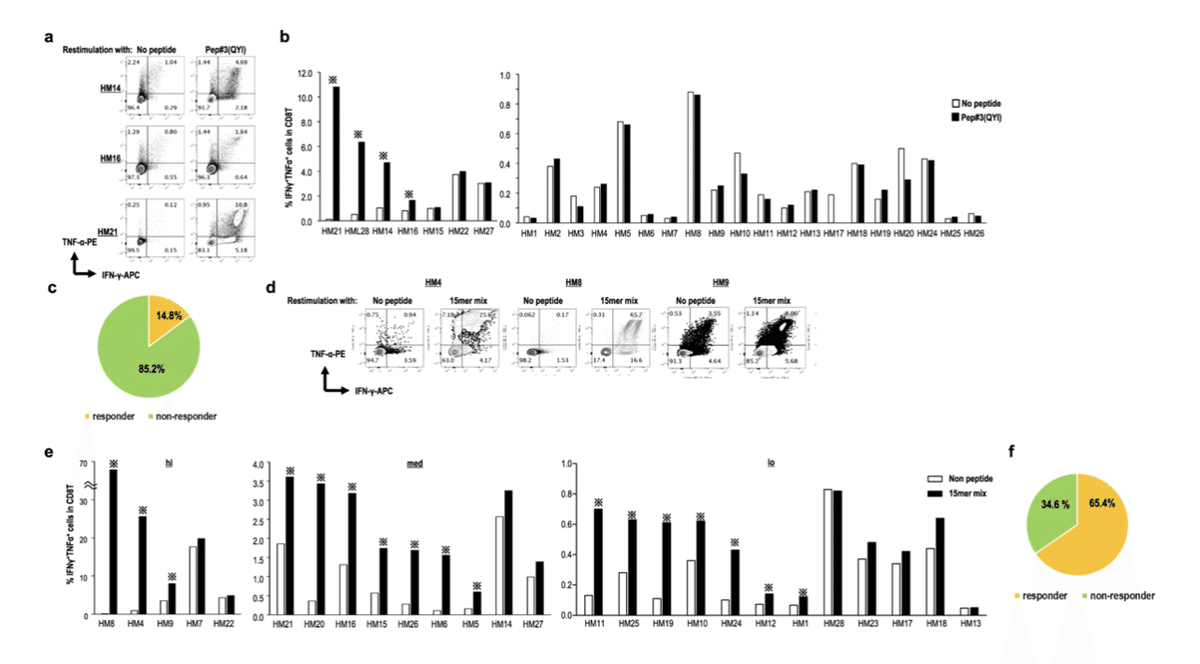
次に、これらの患者を、11 merオーバーラップした15merペプチドの4つのタイプ別に解析した(図5d-f)。驚くべきことに,HM患者の65.4%にSARS-CoV-2特異的CD8+T細胞が検出された(図5e,f)。4種類のペプチドを混合した場合のT細胞反応は,特にHM患者において,9merペプチドで誘導される反応よりも効率的であった.このように、HM患者ではUHDに比べて交差反応性のCD8+T細胞が少なかった(65.4% vs 14.8%, p < 0.001; Fisher's/Chi-square test)ことから、季節性コロナウイルス特異的メモリーT細胞は、疾患の進行や治療関連の毒性に応じて減少する可能性が示唆された。しかし、主に15merのpep#2と#3に反応する可能性のあるCD8+T細胞は、15merのペプチドミックスで再チャレンジすることで活性化することができます。さらに、15merのpep#1と#4にも反応するケースもあった(補足図4)。
CD8+T細胞はSARS-CoV-2由来のSペプチドとHCOV由来のSペプチドの間に交差反応性を示す
HLA-A24+ UHD10人とHM患者2人から作製したPep#3(QYI)特異的CD8+ T細胞が、他のコロナウイルスファミリーと交差反応するかどうかを評価し、SARS-CoV-2特異的CD8+ T細胞を定量した(図6a)。Pep#3(QYI)特異的CD8+T細胞株は、各ウイルスの関連ペプチドに反応したIFN-γとTNF-αの産生量に基づいて評価した(図6b)。UHDのPep#3(QYI)特異的CD8+T細胞株は、HCOV間で交差反応性を示した。この2人のHM患者においても、CD8+T細胞の交差反応性が保たれていることがわかった。CD8+T細胞株は、ベータコロナウイルス(SARS-CoV-1、MERS、HKU-1、OC43)には83-100%の反応を示し、アルファコロナウイルス(NL63、229E)には58.3-66.7%の反応を示した(図6c-e)。
図6:SARS-CoV-2 Pep#3(QYI)特異的CD8+T細胞の他のコロナウイルスに対する交差反応性。
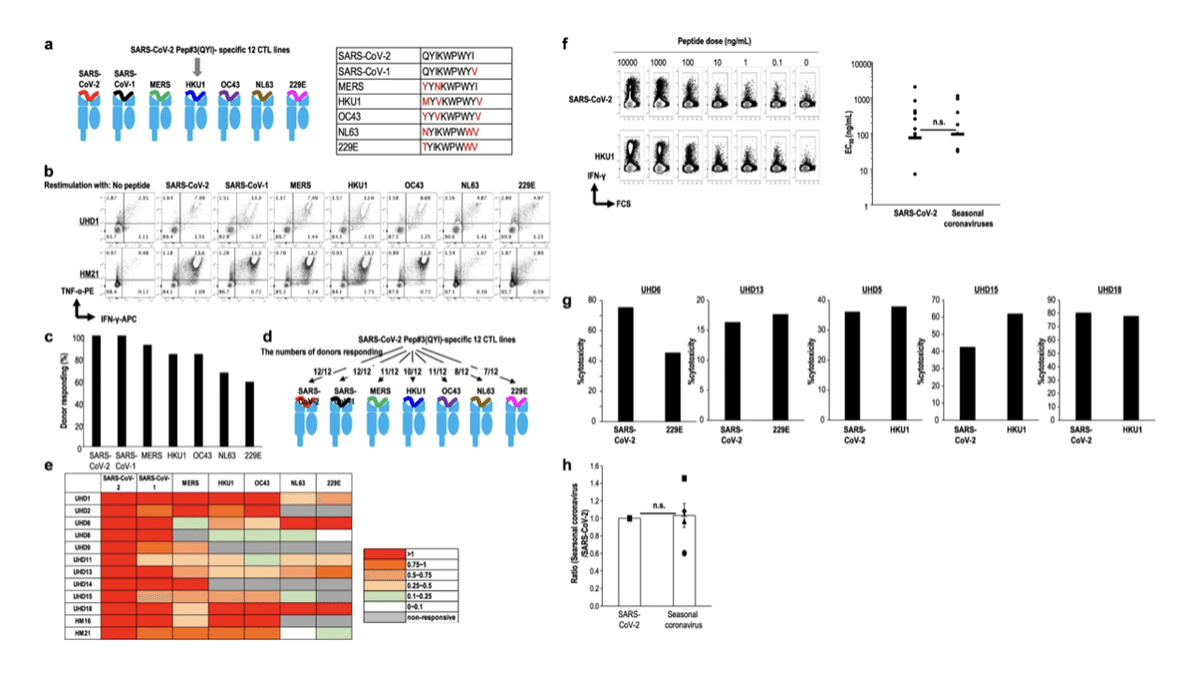
次に、Pep#3(QYI)特異的CD8+ T細胞のコロナウイルスに対する交差反応性のレベルをUHDで評価した。また、IFN-γの産生により、これらのCD8+ T細胞の機能的アビディティを評価した。ペプチド滴定実験では、SARS-CoV-2と季節性コロナウイルスで最も親和性の高いもの(HCOV-HKU1など)について、0〜10,000ng/mLの用量範囲を定めた(図6f)。EC50は,2分の1の最大応答に達するのに必要なペプチド濃度として算出した.SARS-CoV-2 SのEC50は、季節性コロナウイルスのEC50と同程度であった(Fig.6f)。これらのデータは、SARS-CoV-2の交差反応性CD8+T細胞の反応が、季節性コロナウイルスのCD8+T細胞の反応と同様の機能的アビディティを持つことを示している。さらに、異なるコロナウイルスに対する細胞障害性を評価した(図6g、h)。SARS-CoV-2由来の標的ペプチドをパルス照射したA24/CIRに対する細胞傷害活性を、各UHD細胞株においてPep#3(QYI)特異的CD8+T細胞への親和性が最も高い季節性コロナウイルス由来のものと比較した(図6g)。しかし、両者の間に統計的な差は認められなかった(Fig.6h)。これらの結果から、HLA-A24+ UHDにあらかじめ存在するメモリーCD8+ T細胞の中には、SARS-CoV-2を認識する可能性があるものもあるが、最適な条件であれば、SARS-CoV-2に十分に偏らせることができることがわかった。
単一細胞レベルでのSARS-CoV-2とHCOVの認識に対するTCRアビディティ
CD8+T細胞が様々なコロナウイルスに対して複数の反応を示す理由とそのメカニズムを理解するために、CD8+T細胞の単一TCRレベルでの交差反応性を調べた。Pep#3(QYI)で再刺激した後、CD107a+CD8+細胞をゲーティングしてPep#3(QYI)特異的CD8+T細胞株からCD8+T細胞を分離し、TCRレパートリーのシングルセル解析を行った。Pep#3(QYI)反応性CD8+T細胞のクローン性を解析するために、合計227個のT細胞をスクリーニングしたところ、5人のUHDと1人のHM患者から44個のTCRクローンタイプが同定された(補足図5)。さらに,クローノタイプの中から支配的なTCRタイプに着目し,4人のドナー(UHD3人,HM1人)から4種類のTCRαとTCRβのペアを同定した。クローン化されたTCRの特異性と機能を評価するために、TCRαとTCRβの遺伝子をSKW3-CD8AB(ヒトT-ALL)細胞株に導入し、ペプチド特異的な反応を示した(図7a)。興味深いことに、4種類のTCRレパートリーは、エピトープの認識方法が互いに異なっていた(図7b)。UHD2のTCR-T(TCR-T-1)細胞はコロナウイルス由来のすべてのエピトープによく反応したが、UHD8のTCR-T(TCR-T-2)細胞はSARS-CoV-1とSARS-CoV-2にしか反応しなかった。UHD8はどちらにも感染していなかったので、これらのデータは興味深い。Dykemaらも以前の研究27で同様の現象を報告しており、UHDにはSARS-CoV-2特異的な単反応性CD4+Tクロノタイプが検出され、未知のコロナウイルスや他の病原体に対する交差反応の可能性を示唆していた。しかし、このTCR-T-2のデータは、偶然の発見である可能性もあります。さらに、UHD6のTCR-T(TCR-T-3)細胞は、SARS-CoV-2、SARS-CoV-1、NL63、229Eに反応し、HM16のTCR-T-4細胞は、SARS-CoV-2、SARS-CoV-1、MERS、HKU1、OC43に反応したが、NL63と229Eには反応しなかった(図7b、c)。TCRαβを導入したT細胞の中には、HCOV上の関連エピトープに広く反応するタイプもあれば、2つまたは4つのペプチドにしか反応しないタイプもあった。SARS-CoV-2のエピトープに反応するT細胞のレパートリーに関するこれらの知見は、TCRαβが単一細胞レベルで選択的である可能性を示唆している。
図7:コロナウイルス間のクローンTCRの交差反応性。

次に、TCRを導入したT細胞の機能的なアビディティを、CD69の発現量(MFI)の増加によって評価した。ペプチドの滴定実験では、SARS-CoV-2と各季節性コロナウイルスに対して、10fg/mLから10μg/mLの用量範囲を設定した(図7d)。TCR-T-1は、SARS-CoV-2などと比較して、OC43に対して最も高い結合力を示した。しかし、UHDの他のTCR-T細胞(TCR-T-2からTCR-T-4)は、SARS-Co-V-2に対して最も高い抗原性を示した(Fig.7e)。TCR-T-2(単反応)と他の交差反応性TCR(TCR-T-1、-3、-4)を比較しても、機能的なアビディティに大きな違いは見られなかった(図7e)。また、過去の感染状況にかかわらず、UHDのPep#3(QYI)特異的CD8+T細胞は、単一細胞レベルで十分な交差反応性を示した(図7e)。これらのデータから、UHDのPep#3(QYI)特異的CD8+T細胞は低アビディティを示さないと結論づけた。
HLA-A*24:02に結合するCD8+T細胞とTCR複合体の結晶構造
HLA-A*24:02と3つのペプチド、すなわちSARS-CoV-2、229E、HKU1の結晶構造を決定した(図8a、表1)。これら3つの複合体構造は互いにほぼ一致しており、そのrmsd値はHLA-A*24:02-CoV2とHLA-A*24:02-229E、HLA-A*24:02-CoV2とHLA-A*24:02-HKU1、HLA-A*24:02-229EとHLA- A*24:02-HKU1の間でそれぞれ0.15Å、0.41Å、0.41Åであった。複合体構造では、HLA- A*24:02と各ペプチドの相互作用に結晶パッキングは関与していなかった。

9merのペプチドはHLA-A*24:02構造の同等の位置に同定された(Fig.8b)。ペプチド骨格の電子密度は観測されたが、側鎖の電子密度は低く、特に3つのペプチドではLys4、CoV2と229EではTrp5と7、HKU1ではPro6が観測された。これらのペプチドの骨格のコンフォメーションはよく重なっていた。9つの位置のうち、P2、P3、P6、P9の位置の側鎖は、結合ポケットの底にあるHLA-A*24:02-ペプチド間の相互作用に関与していた。一方、P1、P4、P5、P7、P8の位置にある側鎖は、溶媒領域に向けられ、複合体構造では異なる方向に向いていた(Fig.8c)。したがって、これらの側鎖はTCRと直接相互作用すると予想される。
Protein Data Bankには、これまでに31のHLA-A*24:02-ペプチド複合体の構造が登録されている。ペプチドの長さは8-merから11-merの範囲である。このうち、9merのペプチドとの複合体構造は2つある29。これらの9merペプチドと今回のペプチドを比較すると、すべてのペプチドがP2位とP9位で結合ポケットに固定され、P4位からP7位ではコンフォメーションの変化が観察された(図8d)。この領域はバルジ構造を形成しており、TCRとの推定上の相互作用部位に相当する。さらに、HLA-ペプチド-TCR複合体構造30(PDBコード3VXM)のHLA部分を本研究で述べた構造に置き換えて構造モデリングを行い、ペプチド配列がTCR/pMHC三分子複合体およびpMHC生体分子複合体の界面や固有の可塑性に与える影響を調べた。
考察
COVID-19における既存の交差反応性T細胞の役割を解明することは、COVID-19の発症と重症度を理解し、診断ツールやワクチンを開発するための最適なT細胞関与戦略を決定する上で重要である。低アビディティの交差反応性T細胞の拡大は、感染症の悪化につながる可能性があります。そのため、既存のT細胞を同定するだけでなく、機能的に有能で高いアビディティを持つ交差反応性T細胞を拡大するアプローチを見つけることが重要である。今回の研究では、SARS-CoV-2と季節性コロナウイルスの間で保存されているS2領域のアミノ酸配列に着目し、9merのペプチドを選択した。選択されたペプチドの1つであるPep#3(QYI)は、HLA-A24+UHDにSARS-CoV-2交差反応性CD8+T細胞を効率的に誘導した。これらのPep#3(QYI)特異的なUHD由来のCD8+T細胞は、多機能性を示し、季節性コロナウイルスに対しても同様の細胞毒性を示した。さらに、Pep#3(QYI)を含む免疫優勢エピトープが、3つの15merのオーバーラップするペプチドでカバーされていることを確認した。さらに、そのメカニズムを解明するために、Pep#3 (QYI)特異的TCRの交差反応性と機能的アビディティを単一TCRレベルで評価した。
既存のT細胞との交差反応性は報告されているが10,11,12,13,14,19,31、SARS-CoV-2の交差反応性T細胞が防御的であるかどうかは、まだ議論の余地がある。SARS-CoV-2交差反応性CD4+T細胞は低いアビジティを示すことが報告されており、いくつかの研究では非保護的であることが示されている24,27。しかし、UHDにおける既存のCD8+ T細胞の交差反応性を取り上げた研究は3件あります。Schulienらは、SARS-Cov-2感染前後の3人の患者の既存および誘導CD8+T細胞の分析を行い、抗ウイルスエフェクターCD8+T細胞の重要性を示した16。T細胞の交差反応性は、1つのTCRで2つ以上の異なるペプチド-MHC構造を認識することと定義できる32。Mallajosyulaらは、コロナウイルス間で保存されたペプチドを認識するT細胞がUHDに豊富に存在することを示した33。さらに、COVID-19患者ではこれらの細胞がより多く存在することをスフェロマー技術で検証しました。また、UHDと軽症のCOVID-19患者の間で、SARS-CoV-2特異的CD8+T細胞のTCRモチーフの共有が検出され33、COVID-19における既存のCD8+T細胞の保護的役割が示唆されました。最近の研究では、免疫優勢でHLA-B7制限のあるSARS-CoV-2ヌクレオキャプシドタンパク質エピトープの交差反応性CD8+ T細胞が報告されている28。この研究では、曝露したドナーと曝露していないドナーのエピトープ特異的CD8+ Tクローンタイプに共通のCDR3βモチーフが存在することを発見し、HLA-B7+の人の既存の免疫がクローンの拡大に有利であることを示唆した。今回の研究では、HLA-A24*02制限下のHCOVにおけるPep#3(QYI)特異的CD8+T細胞のTCR交差反応のパターンを単一細胞レベルで示し、実質的な機能的アビディティを示すことができた。とはいえ、HLA-A24+のUHDや感染者のTCRの特徴を調べるさらなる分子的研究が必要である。
がん患者は、COVID-19による重症化のリスクが高く、死亡率も高い20,34,35。特に、COVID-19を有するHMの成人患者は、34%の死亡リスクがあることがわかりました。60歳以上の患者は、死亡リスクが有意に高かった22。さらに、骨髄腫、リンパ腫、CLLなど、B細胞の機能が低下している患者は、標準的なワクチンに反応しない可能性があり、特別な注意が必要な場合もあります36,37。15merのペプチドミックスを用いて、SARS-CoV-2交差反応性CD8+T細胞を検出したところ、Pep#3(QYI)を用いて検出した場合と比較して、AML、CML、MMにおいて、それぞれ75%、50%、56%の高い確率で検出することに成功した。このように、15merのペプチドミックスは、CD8+T細胞誘導ワクチンとして、あるいはHMのハイリスクHLA-A24+患者におけるCOVID-19ワクチンの有効性を示す免疫モニタリングツールとして機能する可能性がある。これらの知見は,COVID-19の病態におけるHLAの重要性を示しているので,他のHLAとペプチドの相互作用についても研究すべきである。
HLA-ペプチドとTCRを結びつけるための制限された公式は、SARS-CoV-2全体のヘテロタイプの交差反応性を与える。さらに、SARS-CoV-2と季節性コロナウイルスとの間のTCRα/βの交差反応性認識の分子基盤を解明するために、HLA-A*24:02分子に結合したSARS-CoV-2のペプチドと季節性コロナウイルスの関連ペプチドの構造を調べた(図8)。得られたHLA-A*24:02-ペプチド複合体構造のデータから、P1、P4、P5、P7、P8の位置にある側鎖がTCRと相互作用することが示唆された。重ね合わせたモデル(HLA部分を本研究で発表した構造に置き換えたもの)では、上記のペプチド位置がTCRの接触面に近いことが示されている。P1/P4/P5およびP7/P8位置の側鎖は、それぞれTCRαおよびTCRβのループ部位と相互作用することができる。TCR1-4の配列を比較すると、これらのループ領域には挿入や欠失が見られ、HLA-A*24:02ペプチド複合体とTCRの間には様々な親和性があることが示唆された。さらに、SARS-CoV-2エピトープにおけるT細胞の選択的交差反応性は、HLA-A*24:02複合体上のペプチドの構造様式とTCRのコンフォメーションの配列/ループ領域の組み合わせに依存することが示唆された。今後は、HLA-A*24:02-SARS-CoV-2-S2およびHLA- A*24:02-seasonal coronavirusエピトープとの交差反応性TCR複合体の構造を解析し、交差反応性を広く裏付ける研究が必要である。
その結果、TCRとHLA- A*24:02という特異的なHLAの構造との相互作用によって、交差反応が起こる可能性があることがわかった。さらに、Pep#3(QYI)特異的なCD8+ T細胞のTCRα/βは、HCOVに対して様々なパターンの交差反応性を示した。最後に、すべてのUHDに加えてHMを持つほとんどの患者に対しても、SARS-CoV-2特異的CD8+ T細胞を刺激する可能性のある最適な15 mer混合ペプチドを決定し、その結果、刺激エピトープの可能性が示された。我々は、季節性コロナウイルスに作用するCD8+T細胞の一部が、UHDでは長期記憶細胞として残存しているのではないかと推測した。これらの交差反応性T細胞を、免疫優勢エピトープを含む一種のワクチンで刺激した場合、これらはSARS-CoV-2に偏る可能性がある。交差反応性CD8+T細胞を調節するワクチンの開発は、この病気の発症率を下げるのに役立つかもしれない。
方法
ヒトサンプルと準備
末梢血サンプルは,当研究所の健康な献血者(2020年初頭)または健康な献血者のバフィーコート(2004年~2010年)から得た(東京赤十字社,東京,日本)。血液悪性腫瘍患者(HM)28名の末梢血サンプルは、国立病院機構熊本医療センター(日本、熊本)から入手した。UHDや非被爆者のHM患者はいずれもCOVID-19に罹患しておらず、COIVD-19患者との密接な接触もありませんでした。さらに,UHDにはSARS-CoV-1やMERSの感染歴はありませんでした。健康なドナーおよびHM患者の血漿サンプルは,COVID-19 Human IgM IgG Rapid test(Abnova社)および抗SARS-CoV-2 IgG ELISAキット(EUROIMMUN社)で評価されました。製造元によると、10日目以降のサンプルにおけるユーロイムンキットの感度は94.5%であった。いずれの抗体も陽性となった患者はいなかった。この原稿では、これらのドナーを非曝露ドナー(UHD)と呼ぶ。PBMCは,Ficoll-Paque PLUS(GE Healthcare,Uppsala,Sweden)密度遠心分離により分離し,リン酸緩衝生理食塩水(PBS)で2回洗浄した後,使用するまで液体窒素で保存した.健康な血液ドナーとHM患者の特徴に関する情報は,それぞれ補足表1と2に示した.本研究は、理研IMSのInstitutional Review Board for Human Researchによって承認された。健康なドナーおよび患者全員から、ヘルシンキ宣言に従って書面によるインフォームドコンセントを得た。T細胞解析のために、フローサイトメトリーを用いてHLA-A24+のドナーを選択した。
細胞株
HLA-A*24:02を発現するCIR細胞(A24/CIR)は、滝口雅史博士(熊本大学)38よりご提供いただきました。SKW-3(T-ALL)細胞株は、RIKEN BRC(日本)から入手した。すべての細胞株は、製造者のプロトコルに従って検査され、マイコプラズマを含まないことが証明されました(Mycoplasma Detection Kit; Minerva Biolabs)。A24/CIRは、我々の手でHLA-A24の発現を定期的に検査した。国際細胞株認証委員会の登録簿(https://iclac.org)によると、SKW-3細胞がKE-37細胞と誤認されていた。我々の細胞はTCRを持たず、T細胞受容体とCD8ABを導入することで機能的にはT細胞と同様に働くことを確認しています。
エピトープの予測とペプチドの選択
HLA-A*24:02に対するSARS-CoV-2および他のHCOVのSタンパク質CD8+Tエピトープは、Immune Epitope Database and Analysis Resource (IEDB) (https://www.iedb.org/)39およびNetMHC 4.0 (http://www.cbs.dtu.dk/services/NetMHC/)40,41を用いて予測した。対応するタンパク質のアクセッション識別番号は以下の通りである。SARS-CoV-2分離株、Wuhan-Hu-1 NCBI: YP_009724390.1、SARS-CoV-1 NCBI:NP_828852.2、MERS NCBI:YP_009047204.1、HCOV(HKU1) NCBI:YP_173238.1、HCOV(OC43) NCBI:YP_009555241.1、HCOV(NL63) NCBI:YP_003767.1、HCOV(229E) NCBI:NP_073551.1です。ヌクレオカプシドのリン酸化蛋白質に含まれるNFKDQVILLは、SARS-CoV-2のドミナントT細胞エピトープとして報告されている9。
SARS-CoV-2特異的T細胞のインビトロ展開
ペプチドスクリーニング試験では、PBMC(3〜5×106細胞)を、IL-2(100U/mL)(塩野義製薬株式会社)存在下の培養液中で、表示されたペプチド(各ペプチド10μg/mL)(補足表3にまとめてある)で刺激し、各ペプチドでパルスした30Gy照射の自家PBMCを毎週再刺激した。そして、これらの細胞を21日目に分析した。Pep#3(QYI)に対するCD8+T細胞の反応を測定するために、CD8+T細胞(0.5-1×106個)をCD8 MACSビーズ(Miltenyi Biotec)を用いてPBMCから分離し、分離した細胞をIL-2(100 U/mL)の存在下で照射したPep#3(QYI)パルス化した自己PBMC(1:1比)で刺激し、さらに7日目にPep#3(QYI)パルス化した照射自己PBMCで再刺激した。その後、14日目に細胞を分析した。15merペプチドを用いてCD8+T細胞応答を決定するために、PBMC(3〜5×106細胞)を、IL-2(100U/mL)の存在下で培養液中の15merペプチド混合物(各ペプチド10μg/mL)で刺激し、7日目にペプチド混合物でパルスした照射済み自己由来PBMCで再刺激した。その後、14日目にこれらの細胞を分析した。培養物には,IL-2を含むR10培地(10%FBS,55mM 2-ME,1%ペニシリン/ストレプトマイシンを添加したRPMI-1640培地)を週2回または週3回補充した。採取した細胞をR10で洗浄した後、ブレフェルディンA(50μg/mL)(SIGMA)およびモネンシン(750ng/mL)(SIGMA)の存在下で、指示ペプチド(10μg/mL)の有無にかかわらず16時間再刺激し、フローサイトメトリーを用いて解析した。閾値を決定するために、PBMCまたはUHDから選別したCD8T細胞を用意し、IL-2の存在下でペプチドなしで培養した。その2週間後、Pep#3で再刺激し(対照細胞は再刺激しなかった)、IFN-γ産生を細胞内サイトカイン染色で解析した。そして、再刺激群のIFN-γ+ CD8+ T細胞の%の、対照群のそれと比較しての倍数変化を算出した。今回の実験では、倍数変化が1.5を超えなかった(平均±2SD:0.94±0.4)ので(補足図1c)、閾値を1.5とした。
フローサイトメトリー
抗体は,BD Bioscience社,Biolegend社,またはe-Bioscience社から購入した(補足表4にまとめた)。刺激後、FACS緩衝液(PBSに2%の熱不活化FBSを加えたもの)中でHuman TruStain FcX(BioLegend社製)とインキュベートしてFc-Blockadeを行い、表面抗体であるCD3-PE/Cy7、CD4-PerCP/Cy5、CD8-BUV737またはFITCとLive/Dead Fixable AquaまたはViolet Stain(Life Technologies社製)で30分間染色した。細胞内サイトカイン染色のために、PBS中の2%FBSで2回洗浄し、BD Cytofix/Cytoperm溶液を用いて固定および透過処理を行った後、さらに30分間、抗IFN-γ-APC、IL-2-BUV786、TNF-α-PEまたはIL-10-PEを用いて細胞内染色を行った。脱顆粒アッセイでは、再刺激時に抗CD107a-Alexa488を添加して6時間培養した。そして、Fc-Blocking後、表面抗体であるCD3-PE/Cy7、CD4-PerCP/Cy5、CD8-BUV737とLive/Dead Fixable Aqua or Violet Stain(Life Technologies社)で30分間染色し、BD Cytofix/Cytoperm solutionを用いて固定・透過処理を行い、さらに30分間抗IFN-γ-APCで細胞内染色を行った。データ解析には、LSR Fortessa X-20装置とFACSDiva (v 8.0.1) (BD Biosciences) またはFlowJoソフトウェア (v10.3B2) を使用した。
交差反応性の解析
確立したPep#3(QYI)特異的CD8+T細胞株を、Pep#3(QYI)またはHCOVの関連ペプチド(各ペプチド10μg/mL)で再刺激し、細胞内サイトカイン染色によりサイトカイン産生を解析した。各刺激ペプチドに対するTCRアビディティを評価するために、0.1μg/mLから10μg/mLまでの段階的なペプチドの投与量を用いた。TCRのアビディティは、CD8+T細胞の%IFN-γで評価した。さらに、EC50は、我々のアッセイで到達した最大%IFN-γの2分の1に達するのに必要なペプチド濃度を計算することによって推定した。
シングルセルTCRシーケンシングとレトロウイルスTCRベクターの構築
単一細胞TCRの配列決定を以下のように行った。使用したすべてのプライマーを補足表5に示す。CoV-2 pep#3特異的CD8+T細胞株を、10μMのCoV-2 pep#3の存在下または非存在下で、抗ヒトCD107a-BV421で6時間再刺激し、その後、抗ヒトCD8-PEおよびAquaで染色した。ペプチド特異的CD8+CD107a+細胞は、FACS Aria III(BD Biosciences)を用いて、96ウェルプレート内の5μLのRT-PCRミックスに単一細胞として選別した。RT-PCRミックスは、1μLの5×Prime STAR GXL Buffer、0.45μLのRT-PCR primer mix、0.4μLの2.5mM dNTP、0.05μLの40 U/μL RNase Inhibitor、200 U/μL PrimeScript II Reverse Transcriptase(Takara Bio Inc, )、0.05μLの1.25 U/μL PrimeSTAR GXL DNA Polymerase(Takara Bio Inc, )、2.95μLの無核水から構成されていた。ワンステップRT-PCRに用いたプログラムは以下の通りである。45℃で40分、98℃で1分、98℃で10秒、55℃で15秒、68℃で1分を35サイクル。得られたPCR産物は、ヌクレースフリーの水で10倍に希釈し、2nd PCRのテンプレートとして使用した。PrimeSTAR GXLを用いて,pMXs-BamHI-InFusionプライマーとTCRα用CA-rev2プライマーまたはTCRβ用CB-rev2プライマーを用いて,10μLの反応容量で2nd PCRを実施した。2nd-PCRのプログラムは以下の通りであった。98℃で1分間、98℃で10秒、55℃で15秒、68℃で1分間のサイクルを44回繰り返した。2nd-PCR産物をExoSAP-IT PCR Product Cleanup Reagent(Thermo Fisher Scientific K.K.)で処理し,TCRα用CA-rev3プライマーまたはTCRβ用CB-rev3プライマーを用いて塩基配列を決定した。シークエンス結果は、IMGTデータベース(http://www.imgt.org/IMGT_vquest/input)のV-QUESTツールを用いて解析した42。TCRをクローニングするために、ドナーの2nd-PCR産物とPBMC由来のcDNAからVDJ領域とC領域をそれぞれ増幅した。増幅にはPrimeSTAR Maxを用い、pMXs-BamHI-InFusionとCA-rev3またはCB-rev3プライマーを用いた。各断片をBamHI消化した直鎖状のpMXs-IRES-GFP(TCRα用)または-TdTomato(TCRβ用)にInFusion HD反応によりクローニングした。
SKW-3-CD8ABを発現する細胞の樹立
hCD8A-IRES-PuroおよびhCD8B-IRES-Puro VSV-Gレトロウイルスを、5μg/mLのポリブレン(Millipore社)の存在下、2300rpmで90分間、35℃で遠心分離することにより、SKW-3細胞に導入した。CD8AB発現細胞をPE-CD8A(RPA-T8)およびBV421-CD8B(2ST8.5H7)で染色し、FACS Aria IIIを用いて2回ソーティングした。SKW-3-CD8AB発現細胞は、3μg/mLのピューロマイシン(InvivoGen社)で維持した。
TCRのクロノタイピングとαβTCR細胞株の作製
濃縮したTCR VαおよびTCR Vβウイルスを用いて、2×106個のSKW-3-CD8AB細胞に、5μg/mLのポリブレン(ミリポア社製)を35℃で1500×g、1時間の遠心分離を経て感染させた。ウイルスを除去した後、細胞培養用の培地を新鮮な培地に交換した。TCRを導入したSKW-3-CD8AB細胞は、GFP+ tdTOMATO+細胞を選別して濃縮した。
TCRを導入したSKW-3-CD8AB細胞のエピトープ同定とアビディティ解析
導入したSKW-3-CD8AB細胞を、表示したペプチドでパルスしたA24/CIRと1:1の比率で16時間共培養し、フローサイトメトリーでCD69を測定して初期のT細胞活性化を検出した。ペプチドの段階的な滴定(10μg/mLから10fg/mLまで)を用いて、TCRアビディティを評価した。TCRのアビディティはCD69のMFIで評価した。EC50は、本実験で得られたCD69 MFIの最大値の2分の1に到達するために必要なペプチド濃度を算出することで推定した。
細胞障害性アッセイ
CD8+T細胞株を3週間培養した後、エフェクター細胞として採取した。T細胞の細胞障害性アッセイは、以下の方法で行った。CFSE(Molecular Probe)で標識したA24-CIR細胞を、指示したペプチドまたはDMSOで2時間パルスし、2回洗浄した。エフェクター細胞は、1×104個の標的細胞と一緒に、エフェクター/標的細胞比が12.5、25、50となるように6時間培養し、解析の直前にTO-PRO3(Molecular Probe社)で染色して死細胞を同定した。自発的な標的細胞死(SD)は、単独で培養した標的細胞を標識することで判定した。全細胞毒性(TD)のポジティブコントロールとして,標識した標的細胞をBD Cytofix/Cytoperm試薬(BD Pharmingen)で透過させた。Specific lysisは、以下の式で算出した。(Sample - SD/TD - SD) × 100。この細胞をフローサイトメトリーを用いて分析した。
タンパク質の発現と精製
HLA-A24α鎖と,N末端にヒスチジンタグとTEVプロテアーゼ切断部位を持つβ-2-ミクログロブリンを無細胞発現系で共発現させた43,44。無細胞反応混合物は、各ペプチド(CoV2、229E、HKU1)を0.15mg/mL添加して調製した。HLA-A*24:02-ペプチド複合体は、HisTrapカラム(Cytiva)を用いたクロマトグラフィーで精製し、TEVプロテアーゼ消化を行った。その後、タンパク質溶液をHisTrapカラムにかけ、ヒスチジンタグを除去した。続いて、フロースルー画分をHiTrap QおよびSuperdex200ゲル濾過クロマトグラフィー(Cytiva)を用いて精製した。HLA-A*24:02-ペプチド複合体を、50mM NaClを含む20mM Tris-HCl緩衝液(pH8.0)で濃縮した。
結晶化とデータ収集
HLA-A*24:02-CoV2ペプチド複合体の結晶は、293Kでシッティング・ドロップ蒸発拡散法を用いて、1.0M Na/Kリン酸塩(pH8.2)中で6.6mg/mLの濃度で成長させた。HLA-A*24:02-229Eペプチド複合体結晶は、0.2Mクエン酸ナトリウムと20%PEG3350で濃度6.9mg/mLになるように成長させた。HLA-A*24:02-HKU1ペプチド複合体結晶は、0.1 M Tris-HCl(pH8.5)および25% PEG3350で2.4 mg/mLの濃度で成長させた。データ収集は、10%グリセロールを凍結保護剤として、100Kで行った。データはSPring-8シンクロトロン・ビームラインのBL26B245,46,47,48およびBL32XU49にて、波長1.000Åで収集した。回折データは、ZOO/KAMOを用いて処理し、スケーリングを行った。自動化された微結晶データ処理システム50,51およびCCP4ソフトウェアスイート52を用いて、回折データの処理およびスケーリングを行った。
構造決定と精密化
Phaser53の分子置換法(MR)を用いて構造を決定した。新たに同定したペプチド(PDB ID: 4F7M)とHLA-A*24:02の複合体の座標を検索モデルとして用いた。ラマチャンドランプロットによると、CoV2複合体では97.4%/2.6%、229E複合体では97.4%/2.6%、HKU1複合体では97.7%, 2.3%が好ましい領域と許される領域にあることが示された。データ収集と精密化の統計は、Table 1にまとめられている。図中のリボンモデルはPyMOLソフトウェア(http://www.pymol.org)を用いて描かれている。
統計と再現性
統計解析は,StatMate V (Nippon 3B Scientific Inc.)を用いて行った。2群間の比率差の比較には,Fisher's/Chi-square 検定を用いた。独立した2群間の比較にはMann-Whitney U-testを,ノンパラメトリックな多重比較にはTurkeyまたはNeuman-Keuls検定を用いてデータを解析した。p < 0.05を統計的に有意とした。生物学的複製の数またはサンプルサイズは、図の凡例に記載されている。
報告書の概要
研究デザインに関する詳しい情報は、この記事にリンクされているNature Research Reporting Summaryをご覧ください。
データの公開
HLA-A*24:02-ペプチド(CoV2, 229E, HKU1)複合体の構造は、RCSBのProtein DataBank(PDB)にアクセッションコード7EJL, 7EJM, 7EJNでそれぞれ登録されている。TCRの配列データは、BioProjectデータベース(PRJNA779816)に登録されている。本文中で示したグラフの基礎となるすべてのデータは、補足データ1に掲載されている。
日本語訳サイト↓
この記事が気に入ったらサポートをしてみませんか?
