MDS(多次元尺度構成法)の理論とPython
1.多次元尺度構成法とは?
次元削減手法の一種であり、各データ間の距離を保ちながら次元削減する手法です
例えば、首都圏の位置関係について次元削減したいとします。
私たちが生活しているのは3次元空間であり、
緯度経度そして高さが存在します。
例えば富士山の麓と新宿は経度も緯度も違えば高さも異なります。
そのような3次元データを2次元に次元圧縮を行うことを目的としているモデルになります。
2.扱うデータ
今回扱うデータはこのサイトから渋谷、新宿、富士見の経度、緯度、標高のデータを取得してきました。
今回はこのデータを3次元から2次元へ次元圧縮していきたいと思います。


3.まずは理論から
MDSで第一に注目していきたいのが、「距離」です
データ間の距離依存を保ちたいのが目標なので、距離行列というのを作成します。

内積の分配法則などを用いることにより、距離行列を3項に分解させることができました。
そして、後に距離行列を変換させていくために中心化行列を用います。
まずは、中心化行列について説明していきます。

このJnをデータに掛けることにより、偏差を出力させることができます。これを中心化行列と言います。
このJnを左から掛け合わせることにより、各特徴量の平均からの偏差を出すことができ、
Jnを右から掛けることにより、データ間の平均からの偏差を出すことができます。
右からかければ、同じ値が縦に並ぶ感じになります。
そして、距離行列を左右から中心化行列に用いたJnをかけ合わせることにより、分解した距離行列をこのような形にすることができます。第二項の計画行列XXtのみが残る形となります

そしてこの Jn × XXt × Jn が次元削減したYYt行列とほど同じになれば良いことになります。
XXtの形を見てみると分散共分散行列に似ていることがわかります。つまり、この中心化されたJn × XXt × Jnの固有値固有ベクトル問題を解き、
最終的に次元削減したYYtを求めます。
より詳しく固有値固有値ベクトルまで解きたい場合は別の資料を参考にしてみてください。後日また記事にもしたいなとも思っています。
4.モデル実装
ではここから理論が終わったところでPythonによる実装を行なっていきたいと思います。
4.1 必要なモジュールをインストール及びデータの準備
# 必要なモジュールのインポート
import matplotlib.pyplot as plt
import japanize_matplotlib
import numpy as np
import pandas as pd
from mpl_toolkits.mplot3d import Axes3D
import time
from sklearn.manifold import MDS
# データの準備
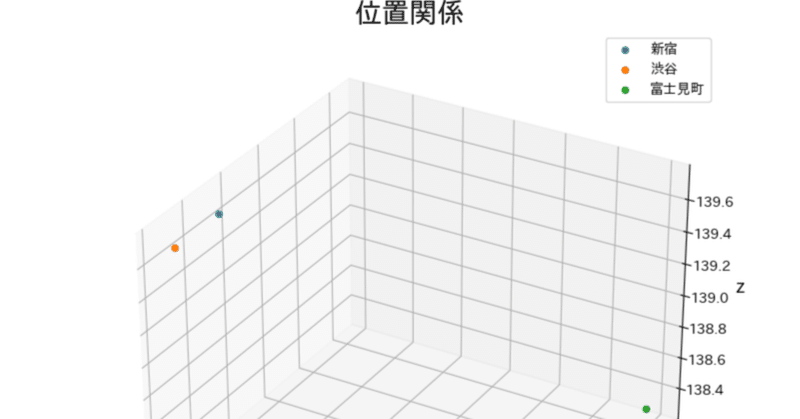
X = np.array([[24.7 , 35.718, 139.703] , [32.9, 35.660,139.711],[1183.5,35.910,138.302]])
df = pd.DataFrame({"標高":X[:,0] , "緯度":X[:,1], "経度":X[:,2]} , ["新宿","渋谷","富士見町"])4.2 データの可視化(次元削減前)
経度、緯度、標高のデータのため、距離がとても関係しているデータですね。こういう場合にはMDAというのはとても便利なのです。
では実際に可視化していきましょう。
# Figureを追加
fig = plt.figure(figsize = (8, 8))
# 3DAxesを追加
ax = fig.add_subplot(111, projection='3d')
# Axesのタイトルを設定
ax.set_title("位置関係", size = 20)
# 軸ラベルを設定
ax.set_xlabel("x", size = 14)
ax.set_ylabel("y", size = 14)
ax.set_zlabel("z", size = 14)
# 曲線を描画
for i in range(len(X)):
ax.scatter(X[i,:][0] , X[i,:][1],X[i,:][2] , label=df.index[i])
ax.legend()
plt.savefig("位置関係.png")
plt.show()
4.3 モデル実装
MDSの実装はsklearnを用いることによってとても簡単に実装することが可能です。ではコードを書いていきます。
#モデルのインスタンス化(次元削減後の特徴量の数をn_componentsに記載)
%time mds = MDS(n_components=2)
#訓練データを次元圧縮
%time Y = mds.fit_transform(X)
#こんな感じで時間を計測することが可能
CPU times: user 16 µs, sys: 5 µs, total: 21 µs
Wall time: 28.8 µs
CPU times: user 11.5 ms, sys: 3.4 ms, total: 14.9 ms
Wall time: 14.1 ms4.4 結果の可視化(次元削減後)
次元圧縮されたデータを可視化してみましょう。
3次元のデータと比べて、距離があまり変わらずに次元削減できたことがわかりますね。

5.まとめ
以上でMDAの理論及び実装が終了しました。
実装は簡単ですが、理論の部分がやはり複雑ですね。
結構距離が関係してくるモデルとか、次元削減なんか
あとは数理最適化なんか色々な場面で固有値固有ベクトルの問題が出てきます。
固有値固有ベクトルはある行列が正方行列の場合のみに用いることができるもので、違う場合は特異値問題なんかも使われます。
なんにせよ、固有値固有ベクトルについてはこれからも仲良くしていきたいと思いました。
この記事が気に入ったらサポートをしてみませんか?
