ヒト同士のゲノムはどれくらい違うのか
ヒトとチンパンジーのゲノム配列は、98%以上似てるという言説はよく見る。人間同士でも個人差があるけど、定量的にどの程度ゲノム配列に差があるか自分で確認しようという試み。
核染色体の比較
2018年時点で、チンパンジーリファレンスゲノムは、28億bpほどあり、5000個近い場所の定まってないscaffold(長さにして2億bpほど)がある。余ったscaffoldを無視して、ヒトゲノムと比較すると、大体、全塩基の1~2%くらいで一塩基置換が検出できる。ヒトとチンパンジーのゲノムが98%とか99%同じという数字は、この結果から来ている。
一塩基バリアント以外にも、一部の遺伝子は、遺伝子重複によって、遺伝子数が人によってかなり違うという報告もあるけど、以下では、調べやすい一塩基バリアントのみを対象とする。
最近は、高等生物でも個体ごとに全ゲノムを読むコストが大分下がった。ヒト個人単位でのゲノム配列は、データが公開されていたりする(Human Pangenome Reference Consortium)。しかし、これらの個人単位で読んだらしい配列は、assemblyレベルがscaffoldまでで、多少扱いが面倒くさい。
AssemblyレベルがChromosomeかCompleteになってるヒトゲノム配列となると、あまり数は多くない。個人単位で読んだ配列は、Craig Venter(ヒトゲノム計画の立役者とされるアメリカ国籍の人)のしかない。この中に、中国リファレンスゲノムと韓国リファレンスゲノムがあるので、このへんを使うことにする。ここに登録はされてないが、検索したら、日本のリファレンスゲノムもあった。
インドはIndiGenomesで、VCF(Variant Call File)を公開している。一塩基置換の場合は"VRT=1"というタグ付があり、それ以外の挿入、欠失の場合は、"VRT=2"というタグ付がされている。VRT=1になってるvariantの数を数えると、16079000個だったので、ヒトゲノムサイズが30億bpとすると約0.5%ほど。これらのバリアントは、複数の"インド人"から集めたものだろうから、個人レベルでは、もっと少ないのだと思う。なお、ここでは、"XX人"という単語は、単に、特定の国や地域に住んでる人という以上の含意はない。
特に、東アジア諸国のデータを重点的に探したわけでなく、欧米諸国が自国の参照配列を作成したりしてるのを発見できなかった。標準のヒトゲノム参照配列(主にアメリカ在住の男性から作成したとされる)で十分と判断したのかもしれない。それか、東アジアほど、国民の同質性を信じてない(中国は広いし国民の同質性を信じてるか知らないけど)のかもしれない。
アラインメントツールとしては、LASTZを使用する(version 1.04.37 released 20230822)。使用できるメモリを増やすために、lastz_32をmakeした方がいい。異なる種のDNA配列を比較する時には、デフォルトのパラメータで大抵は問題ないが、今回は、類似度の高い配列同士の比較になるので、多少パラメータの設定を考えてやる必要がある。
極端な話、デフォルトパラメータだと、同一配列を比較しても、そこそこのミスマッチが出てきて、一塩基置換があるように見えてしまう。種が違えば、同一遺伝子でも配列は大きく異なる場合があり、仕方ない。また、遺伝子重複などがあって、個人間でも、重複遺伝子数が違ったりするらしい。その場合、ある人のゲノム配列の一領域と似た配列が、別の人のゲノム上では複数存在するというようなことが起こりえる。従って、人間同士でも単純に一対一対応するというわけではない。
方法の原理的な限界もあって、正しいパラメータの設定というのはない。厳しい条件下では、少しでも配列が似てないとアラインメントされなくなり、逆に緩い条件では、相同性の低い配列同士でもアラインメントされてしまう。このへんはトレードオフなので、最終的には勘で選ぶことになる。目安としては、同一配列の比較で検出するミスマッチが多すぎず、また、ヒトとチンパンジーの比較で、検出されるミスマッチが減りすぎないような範囲でパラメータを選べばいいだろう。
今回使用したコマンドは以下の通り(lastz_32はデフォルトのlastzよりメモリが多く使えるよう)
lastz_32 $seq1 $seq2 --hspthresh=50000 --chain --gfextend --step=20 --format=maf
また、今回、使用した配列は、以下の通り。
hg38: ヒトゲノム参照配列(アメリカ人由来?)
JG2.1.0 :日本人ゲノム参照配列
Southern Han Chinese Genome Assembly: 中国南部の人から作成した参照配列らしい
KOREF 2.1: 韓国人参照配列
J Craig Venter: Craig Venterの個人配列(2008年頃作成)
CHM13hTERT human cell line: 培養細胞株CHM13hTERT。一対の染色体が完全に同一でヒトゲノム配列の完全決定を目的として作成されたらしい
ヒト以外のゲノム配列として、比較用に、チンパンジー(panTro6)とボノボ(panPan3)を使った。
比較結果は、以下の通り。22対の常染色体とX,Yの性染色体のアライメント結果を集計して合算した。
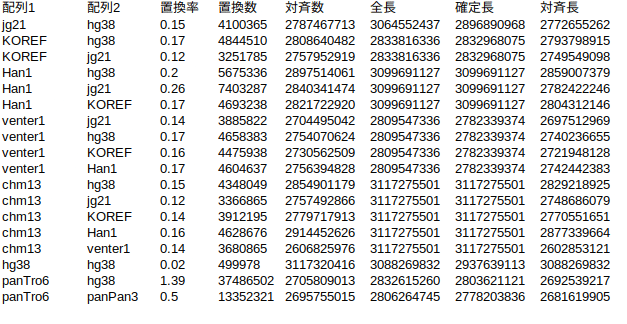
表の項目の説明。解析の健全性確認のため、色々と集計しているが、置換率だけ見ればいい。
置換率(%):置換数/対斉数を%で表示したもの
置換数 :アラインメントされた配列間で塩基が一致してないものの総数(一方ないし両方がNの場合はカウントしない)
対斉数 :アラインメントされた塩基ペアの総数。複数領域にアラインメントされたら重複カウント
全長 :配列1の全長
確定長 :配列1のNじゃない塩基数
対斉長 :アラインメントされた配列の全長。複数領域にアラインメントしても重複カウントしない。対斉数より小さい
ヒトとチンパンジー(panTro6)で置換率が、1.39%なので、両者は98.6%くらい相同性がある。hg38とhg38は同一配列なので、置換率は0になってほしいが、諸々の都合で、そうはならない。しかし、他に比べれば、大分小さいので、大きな問題ではないだろう。
まず、人間同士は、チンパンジーとボノボよりもゲノムの差異が小さいことが分かる。ボノボは、ピグミーチンパンジーという名前だったこともあるし、両者は交配可能らしい。チンパンジーかボノボかクイズ出されても、正答できそうにない程度には両者は似ている。


また、十分なサンプル数が有るとは言えないけど、人間同士のゲノムの差は、居住地の地理的距離と関係してるのかは分からない。一応、置換率が、最も小さいのは、KOREFとjg21で、日本と韓国で似てるというのは尤もらしい気もする。
直感的には、地域間の移住が少ないとすれば、ある地域の住民は遠い親戚みたいなもので、似てそうな気もする。仮に、いとこ同士で、相同組み換えを考慮しないとして、genericには、ゲノムの1/8を共有することになる。残りの7/8で平均的に置換率が0.2%としても、全体としては0.175%になる程度。近親交配を繰り返さない限り、集団内での相同性は一定水準が維持されるのかもしれない。
一方、2つの集団が分岐すれば、独立に進化していくことになるが、数万年程度では影響は小さい。ヒトとチンパンジーの分岐時期を通説通り700万年前と仮定した場合、10万年でヒトゲノムに入る一塩基置換は、全配列の0.01%程度。つまり、通説通りに"ホモ・サピエンスの出エジプト"があって、それ以後、(別の集団を介してすら)全く交流してない二集団があったとしても、両集団のゲノムを資格した場合、置換率は、元々存在した置換率に0.02%程度上乗せされるくらい。
個別の一塩基バリアントについては、特定の地域に偏って確認される例が多数知られている。そういうバリアントは、通常、表現型に影響している。よく研究されてる例に限れば、それらは、遺伝子上にあって、翻訳タンパク質のアミノ酸に変化を起こす非同義置換だろう。アミノ酸配列は変えず、遺伝子の発現量や発現パターンを変化させるタイプの変異もあるかもしれないが、多分、研究は少ない。
一方、ここで検出しているバリアントの殆どは、遺伝子上にあるとは限らず、表現型にも影響してなくて、特に有益でも有害でもないと思われる。タンパク質コード領域は、ゲノムの2%程度と言われる(概算だが、遺伝子数2万で、タンパク質の平均サイズを1000アミノ酸とすれば、1アミノ酸につき3bpなので、(2000010003)/30億=0.02)。
従って、hg38配列と比較して、300万〜600万の一塩基バリアントがあっても、非同義置換は、最大でも10万個を超えないだろう。遺伝子数が約2万と考えれば、これはまだ多すぎる気もするし、非同義置換は入りにくくて、これより少ないのかもしれない。あるいは、機能に影響しない非同義置換が結構あるのかもしれないが分からない。知られてるタンパク質に限れば、非同義置換は列挙できるが、今回はやってない。
定量的には、(チンパンジーとヒトの違いが1.5%というのと同じ意味で)ヒト同士のち外は、0.15~0.2%くらいということになるが、この数値は信用できるのだろうか。
多分、シーケンスミスが、どれくらいの頻度で入ってるかは検討されるべきなんだろう。例えば、シーケンスミスが1/2000の確率で起きると仮定すると、同一の配列を二回読んだ場合、それそれに、0.05%の頻度で間違った塩基が入り、2つのシーケンス結果を比較すると、0.1%程度の不一致があるように見えるだろう。チンパンジーとヒトの比較であれば、この程度のシーケンスミスは問題にならないとしても、ヒト同士の比較では、大きな問題となり得る。
そんなにミスがないとは思うけど、具体的な数値は不明なので、定量的に信用できるとは言い切れない。
ミトコンドリアDNAの比較
hg38、中国、日本の参照配列には、ミトコンドリアDNAも含まれてたので、デバッグ目的で比較してみる。
ミトコンドリアDNAは環状なので、どこを始点にするか決まりがないが、中国のデータは、hg38とずれてるので、それを合わせた方がいい。結果は、以下の通り。

ミトコンドリアDNAは小さいので、古くから大量のシーケンスがされてる。1987年の(分子生物学界隈の基準では)大変古い論文Mitochondrial DNA and human evolutionには、average sequence divergenceが0.32%だと書いてある。この論文のサンプルには、アフリカ人とかも含まれるので、より分岐が古く、差も大きくなるだろう。
Abstractには、ミトコンドリアDNAの分岐時期が20万年前と書いてあって、よく見る数字だけど、本文には、14〜29万年前(14.3〜28.5万年前)と書いてある。これは一塩基バリアントを獲得する速度が一定と仮定しての推算で、ミトコンドリアDNAは、ゲノムと違って、大部分がタンパク質コード領域なので、仮定の妥当性も怪しい。
その後も、色々と検討して多くの推定値が出版されてる(最近は、16万年が人気っぽい?)けど、1987年の推定範囲から大きな乖離はない。分子時計に基づく種の分岐年代も、文献によって最大2倍くらい違う数字が出てることは多い。現在の所、分子時計の精度がそんなものだと思っておくべきだろう。
ゲノムに関しては、大型類人猿(ヒト、チンパンジー、ボノボ、ゴリラ、オランウータン)で、一塩基バリアントを獲得する速度が一定だという仮定は、割と尤もらしい。これは、大型類人猿のゲノムを比較シてみると分かる。例えば、ゴリラ&ヒト、ゴリラ&チンパンジー、ゴリラ&ボノボで、一塩基置換率がほぼ同一なので、分岐後、どの種も同じペースでゲノムが変化していると考えるのが最も単純な説明。
一世代で、どれくらいゲノムが変化するか調べた研究によれば、マウスなんかは、大型類人猿より早くゲノムが進化していく。
チンパンジーとボノボ、チンパンジーとヒトのミトコンドリアDNAの比較をすると、ゲノムを比較した場合よりずっと多くの一塩基バリアントがあるように見える。つまり、ミトコンドリアDNAの変化速度は、ゲノムが一塩基バリアントを獲得するより、ずっと早いということになるけど、ヒト同士の比較では、ミトコンドリアDNAとゲノムで一塩基バリアントの割合は、大きな差がない。
これは、矛盾してるわけではなく、ミトコンドリアDNAの分岐時期に生きていた現代人Xさんの祖先は、100人か1000人か知らないけど、とにかく、その時点で沢山いたはず。全員がアフリカにいたというわけでもない。その先祖たちのゲノムが、交配と相同組み換えによってモザイク状に混ぜ合わさって、20万年分の変異を入れたのが、Xさんのゲノムということになる。ミトコンドリアDNAの分岐時期にも、既に"ヒト同士"のゲノムの差は当然あったはずで、出発点が同一ではない。
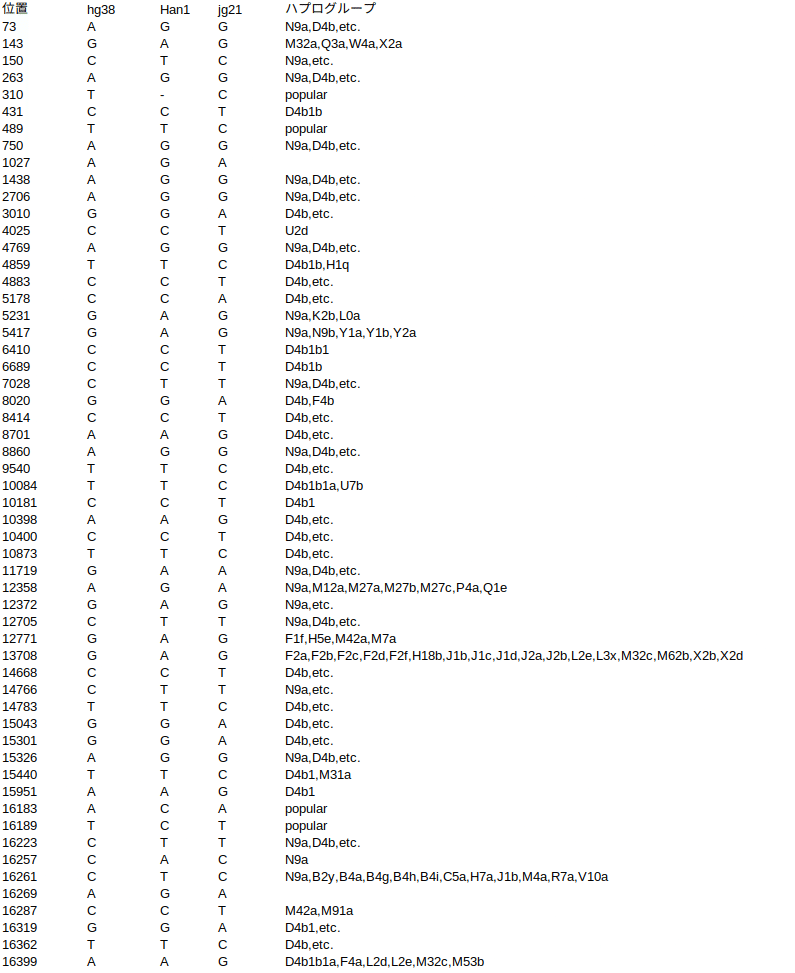
デバッグ目的なので、一塩基バリアントを列挙して、一番共通するバリアントを持つハプログループを(日本人と中国人のそれぞれで)特定する。ミトコンドリアハプログループは、沢山あって、ここにメジャーな分類が載ってる。更に細分されたサブグレードは載ってないものもあるので、特定できなかったバリアントを別のサイトで補完した。
位置は、hg38の配列の位置。"popular"や"etc."とあるのは、20以上のメジャーハプログループに含まれてたバリアント。日本人はD4b、中国人はN9aと共通するバリアントが最多で、それぞれ、日本と中国で多いと言われるやつなので、ミトコンドリアDNAの比較は間違ってなさそう。
この記事が気に入ったらサポートをしてみませんか?
