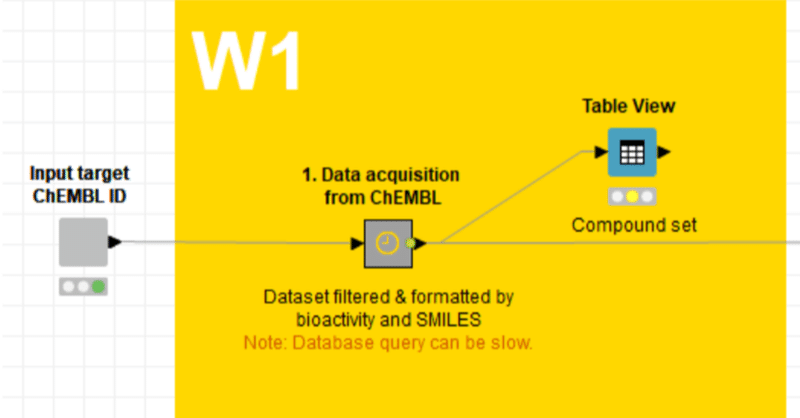
【W1】ChEMBLから化合物データを取得_01_概要
前回はTeachOpenCADDのKNIME workflow全体の話をしました。
今回はW1-8の最初のパートW1について説明を始めます。
【本パートの目的】
ChEMBLからデータを抽出する方法の学習:
• ある特定の標的に対して評価済みのリガンドを見つける
• 取得可能な生理活性データでフィルタリング
• pIC50値の計算
• データフレームを結合し、取り出した分子を描画
(引用元)
丁寧な説明があるのでぜひ一読をお勧めします。
入力:
ある特定の標的のChEMBL ID
出力:
表形式での化合物の構造データと生理活性データ
ではKNIME上で見ていきましょう。KNIMEの操作法なども今回は少し説明します。
【KNIMEでの入力】

上のアイコン(コンポーネントといいます)をダブルクリック、もしくは右クリックして”Configure”を選択すると下記ウィンドウが出ます。
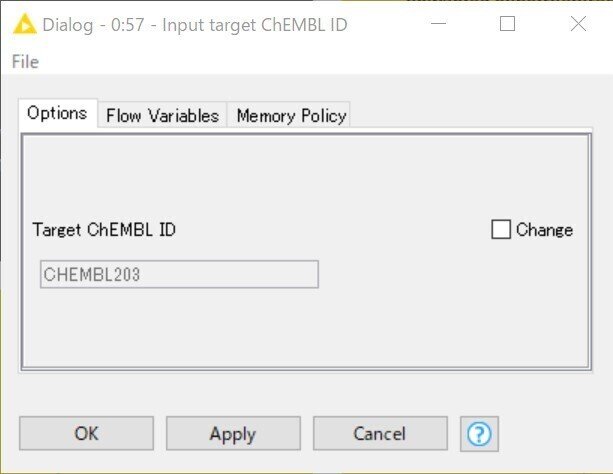
CHEMBL203がデモデータのターゲットEGFR kinaseのChEMBL IDです。
右の「Change」にチェックを入れると変更できますが、今はしないでください。
デモデータでまずはworkflow(WF)全体の流れを説明したいからです。
今回は設定を変えないので、「OK」「Apply」「Cancel」「X」どのボタンを押してもいいので、ウィンドウを閉じてください。
【KNIMEでの出力】
出力は何通りかあるのですが、今回はTable Viewで見てみます。上の上のアイコン(Table Viewノードといいます)を右クリックし、
“Interactive View: JavaScript Table View“を選択して少し待つと、
下記のウィンドウが開きます。
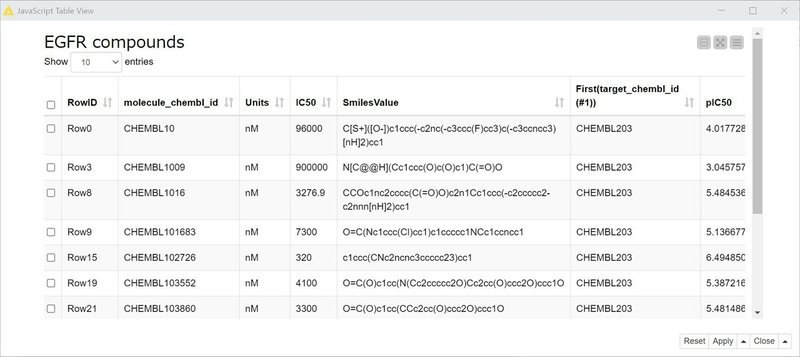
各列(カラムともいいます)について:
RowID: 行のID
molecule_chembl_id: 各化合物のChEMBLデータベースでのID
Units: 各アッセイの活性値の単位 今回のworkflowはIC¬50値のみにフィルターしてある
IC50: IC50値
SmilesValue: 各化合物の構造式のSMILES形式での表記
First(target_chembl_id (#1)): ターゲットのChEMBL ID
(今回はEGFR kinaseのCHEMBL203)
pIC50: log10(IC50値)
データ数など:
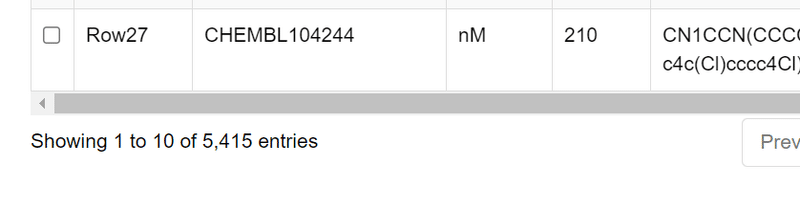
一番左下に5415行のデータ中の1~10番目のデータを表示していると示されています。
データのソート(並べ替え)など:
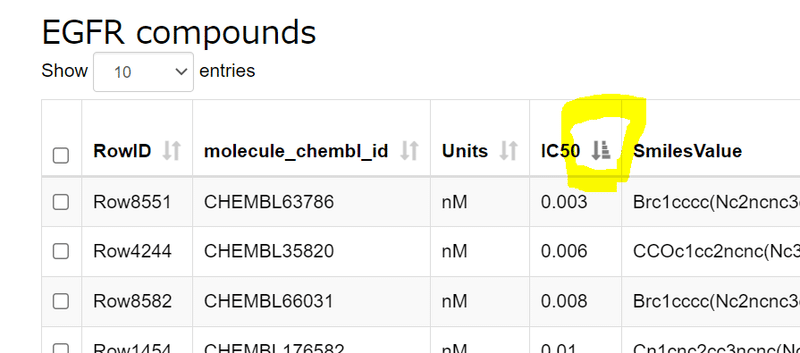
また、各カラムの名前の横に上下矢印が表示されているのですが、そこをクリックすると昇順、降順、ソートなしの順に並べ替え(ソート)が実行されます。
下図はIC50値が最も小さいすなわち活性最強の化合物から順に並べ替えた結果です。CHEMBL63786の化合物はIC50 = 0.003nM、強いですね。構造式も見たいでしょうが、次のWF (W2部分)まで待ってください。
今回はKNIMEで詳しくデータを見る初めての記事だったので細かく説明させていただきました。
今後はもっと大まかに説明していきますのでご容赦ください。
【おまけ:KNIME操作のTips】
WFをなるべく見やすくするため、ウィンドウを拡げたり、表示倍率を変えたりすると便利です。
記事を読んでいただきありがとうございます。 先人の智慧をお借りしつつ、みなさんに役立つ情報が届けられたらと願っています。 もしサポートいただけるなら、そのお金はKNIMEの無料勉強会の開催資金に充てようと思います。
