
まさ @sambadouroの嘘を暴く
こんにちは、翡翠です。
簡単にバレる嘘をつく頭の悪いアカウントを見つけたので、パパッと片付けたいと思います。笑

1. 「感染症研究所の論文は未発表」という嘘
GENBANK登録の取下げした時点で全て崩壊。しかも論文未発表 https://t.co/xFaxuOEVIT pic.twitter.com/fF0ScSvNhI
— まさ (@sambadouro) April 30, 2021
まさ @sambadouro
GENBANK登録の取下げした時点で全て崩壊。しかも論文未発表
午前4:38 · 2021年5月1日·Twitter Web App
彼のツイートの画像がこちらです。

よく見ると、(と言ってもそんなによく見なくてもすぐにわかりますが、)REFERENCE(この配列情報に関連した文献などの意)には、『1』と『2』があります。

それぞれに、AUTHORS(著者)、TITLE(タイトル)、JOURNAL(雑誌)という3つの項目があります。
彼のように、これを一緒にしてしまうのは何も分かっていない証拠です。

実際に登録されたSARS-CoV-2の遺伝子配列の掲載されたページがこちらになります。
Severe acute respiratory syndrome coronavirus 2 2019-nCoV/Japan/AI/I-004/2020 RNA, complete genome
https://www.ncbi.nlm.nih.gov/nuccore/LC521925.1
そこには、以下の記載があります。
GenBank: LC521925.1 (click to see this obsolete version)
Record removed. This record was removed at the submitter's request. Please contact ddbjupdt@ddbj.nig.ac.jp for further details.
訳)削除されたバージョンを表示するにはクリックしてください。
この登録された配列は、送信者の要求により削除されました。 詳細については、ddbjupdt @ ddbj.nig.ac.jpにお問い合わせください。
指示の通りに『LC521925.1』をクリックすると、削除されたSARS-CoV-2の遺伝子配列を見ることができます。
まず、『REFERENCE 1』から解説します。

REFERENCE 1
AUTHORS Sekizuka,T., Matsuyama,S., Shirato,K., Nao,N., Itokawa,K.,
Hashino,M., Tanaka,R., Yatsu,K., Suzuki,T., Suzuki,M., Hasegawa,H.,
Wakita,T., Takeda,M. and Kuroda,M.
TITLE Complete genome sequence of Wuhan seafood market pneumonia virus 2019-nCoV/Japan/AI/I-004/2020 isolated from a patient in Japan
JOURNAL Unpublished
これは国立感染症研究所(感染研)が、この遺伝子配列をデータベースに登録したときのタイトルになります。

https://www.uniprot.org/citations/-63600402734208703
学術雑誌に投稿したものではありません。
したがって、『論文』として『Unpublished(未発表)』となるのは当然のことです。
そして、大事なのは『REFERENCE 2』です。

見ると、REFERENCE 2の後ろに『(bases 1 to 29848)』と書かれています。(REFERENCE 1の方には書かれていません。)
これが非常に大事なポイントで、『この1~29848塩基の長さの遺伝子配列の根拠となる文献はREFERENCE 2である』ことを示しています。
REFERENCE 2 (bases 1 to 29848)
AUTHORS Sekizuka,T., Matsuyama,S., Shirato,K., Nao,N., Itokawa,K.,
Hashino,M., Tanaka,R., Yatsu,K., Suzuki,T., Suzuki,M., Hasegawa,H.,
Wakita,T., Takeda,M. and Kuroda,M.
TITLE Direct Submission
JOURNAL Submitted (03-FEB-2020) Contact:Tsuyoshi Sekizuka National
Institute of Infectious Diseases, Pathogen Genomics Center; 1-23-1
Toyama, Shinjuku-ku, Tokyo 162-8640, Japan
そして、JOURNALの項目には、『Submitted(提出済み)』と書かれています。
論文の提出後、この遺伝子配列が削除されたため情報が更新されていませんが、この提出された論文は、米国科学アカデミー紀要(PNAS)に掲載されました。
Proc Natl Acad Sci U S A. 2020 Mar 31;117(13):7001-7003. doi: 10.1073/pnas.2002589117. Epub 2020 Mar 12.
Enhanced isolation of SARS-CoV-2 by TMPRSS2-expressing cells
https://www.pnas.org/content/117/13/7001
TMPRSS2発現細胞を使うと新型コロナウイルス SARS-CoV-2が効率良く分離できる
https://www.niid.go.jp/niid/ja/basic-science/virology/9486-virology-2020-2.html
PUBLISHED: 2020年3月16日
「論文として発表されていない」は、嘘です!
この論文には、下記の文章が記載されています。
...
Unexpectedly, the NGS data showed contaminated mycoplasma sequences (Mycoplasma hyorhinis and Mycoplasma arginini) from VeroE6/TMPRSS2 cells. CPE in VeroE6 cells persistently infected with SARS-CoV was enhanced by infection with Mycoplasma fermentans (8), but whether a similar situation exists for SARS-CoV-2–related CPE in this cell line is unclear.
訳)予期しないことに、(RNAシーケンスの)NGSデータには、汚染されたVeroE6/TMPRSS2細胞に由来する、マイコプラズマの遺伝子配列(Mycoplasma hyorhinisおよびMycoplasma arginini)が存在することが分かりました。 SARS-CoVに持続的に感染したVeroE6細胞の細胞変性効果(CPE, Cytopathic effectの略)は、Mycoplasma fermentansの感染によって増強されることが報告されていますが(参考文献8)、このVeroE6/TMPRSS2細胞へのSARS-CoV-2感染による細胞変性効果も、同様に生じるかかどうかは不明です。
https://www.pnas.org/content/117/13/7001
簡単に言えば、「実験に使用したVeroE6/TMPRSS2細胞に『マイコプラズマ』という細菌の一種が混入していた」ということです。
今回見つかったもので、Mycoplasma hyorhinisはブタに感染するマイコプラズマで、Mycoplasma argininiはウシに感染するマイコプラズマです。
一般に風邪などの呼吸器疾患を引き起こすMycoplasma pneumoniaeや、リウマチ性疾患との関連が示唆されているMycoplasma fermentansとは種類が異なります。
感染研ではヒトに対する病原体だけでなく、動物に感染する病原体も取り扱っていますから、その検体などに触れた人が培養室に出入りすることで培養液などに混入し、細胞が汚染されてしまったのかもしれません。
実験で使用する細胞培養液は『ろ過滅菌』という方法で、0.22 μmの穴の開いたフィルターを通して細菌などを除去します。しかしながら、小さなマイコプラズマはこのフィルターを通過してしまいます。あってはならないことではありますが、食品への異物混入と同様に、細胞培養をしていれば時々起こり得ることです。
マイコプラズマは目に見えて細胞にダメージを与えることはありませんが、細胞の様々な機能を阻害し、実験結果の信頼性を損なわせることが、最大の問題となります。
マイコプラズマ:効果的な検出と対処方法
マイコプラズマは、細胞への目に見える損傷を引き起こさない一方で、細胞の代謝や増殖、タンパク質合成、サイトカイン分泌、さらには DNA や RNA にも深刻なダメージを与え、実験結果の信頼性を損なわせます。
https://www.cosmobio.co.jp/support/technology/a/mycoplasma-effective-detection-treatment-blg.asp
2007年に、SARS-CoVとMycoplasma fermentansをVeroE6細胞に共感染させると、ほとんどの細胞が死んでしまうことが分かりました(参考文献8)。

Arch Virol. 2007;152(5):1019-25. doi: 10.1007/s00705-006-0924-7. Epub 2007 Feb 7.
Enhancement of cytotoxicity against Vero E6 cells persistently infected with SARS-CoV by Mycoplasma fermentans
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7087332/
今回、Mycoplasma hyorhinisとMycoplasma argininiが感染したVeroE6/TMPRSS2細胞にSARS-CoV-2を感染させても、これほどの細胞へのダメージは見られませんでした。
ただし、全く影響がないとは言い切れず、マイコプラズマに汚染されていないVeroE6/TMPRSS2細胞を用いて同様のSARS-CoV-2感染実験を行うと、SARS-CoV-2の細胞変性効果は今回の論文の結果よりも弱く見えるかもしれません。
このマイコプラズマの混入が、実験結果を『完全に否定』することはありません。
SARS-CoV-2がVeroE6/TMPRSS2細胞に感染した証拠として、まず、SARS-CoV-2の細胞変性効果による、粒状の細胞や複数の細胞が融合してできた多核巨細胞を確認しました。

(画像:https://www.pnas.org/content/117/13/7001)
ウイルス感染により変性した細胞の形はウイルス毎に異なりますので、簡易的にウイルスの種類を判別することができます。

ウイルスによる細胞変性効果(CPE)の観察
http://www.eiken.yamagata.yamagata.jp/biseibutsu/CPE/virus_CPE.html
山形県衛生研究所
この粒状の細胞や多核巨細胞の出現は、マイコプラズマ感染で起きることではありません。(先に述べたように、マイコプラズマ感染による細胞変性の程度の差はあるかもしれません。)
そして、典型的なコロナウイルス粒子の形状を電子顕微鏡で観察しました。

(画像:https://www.pnas.org/content/117/13/7001)
また、感染研の発表の2日前に、オーストラリアのピーター・ドハーティ感染免疫研究所が、SARS-CoV-2の分離・培養に成功したと発表しました。
もちろん、論文としても発表されています。
Med J Aust. 2020 Jun;212(10):459-462. doi: 10.5694/mja2.50569. Epub 2020 Apr 1.
Isolation and rapid sharing of the 2019 novel coronavirus (SARS-CoV-2) from the first patient diagnosed with COVID-19 in Australia
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7228321/
感染研と同様、SARS-CoV-2の特徴的な細胞変性効果(CPE)を確認しました。

(画像:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7228321/)
そして、ウイルス粒子の形状を電子顕微鏡で観察しました。

(画像:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7228321/)
分離したSARS-CoV-2は、『MT007544』としてデーターベースに登録されています。(削除されていません。笑)
Severe acute respiratory syndrome coronavirus 2 isolate SARS-CoV-2/human/AUS/VIC01/2020, complete genome
https://www.ncbi.nlm.nih.gov/nuccore/MT007544.1
これでもまだ「新型コロナウイルスの分離は確認されていない!」、「新型コロナウイルスは存在しない!」と言うのでしょうか?
確かに、感染研のホームページなどで、日本語しか分からない一般人でも英語の学術論文やデータベースなどを見なくても分かるよう、削除の経緯を説明していないというのは問題だと思います。(残念ながら、感染研の人たちが思っている以上に一般人は賢くありません。手取り足取り教えないと私が大変な思いをします。泣)
しかしながら、いくら無知な情報の受け取り側と言えど、世界から見たらイチ島国の研究機関が遺伝子の配列情報を削除した、イコール「SARS-CoV-2は存在しない!」はさすがに見識が狭すぎます。
1年以上も「感染研が削除したからSARS-CoV-2は存在しない」と言っているのは、わざととしか思えないほどバカらしい話だと思いますが?
次。
2. 「健康安全研究センターの登録した情報は完コピ」という嘘
ていうかはっきり言って
— まさ (@sambadouro) April 30, 2021
健康安全研究センターのこれはhttps://t.co/p2zwPRqvQF
中国研究チームのこれhttps://t.co/c10fxEApsA
の完コピです。見比べてみて
まさ @sambadouro
返信先: @Osamu_iigaさん
ていうかはっきり言って
健康安全研究センターのこれは
https://ncbi.nlm.nih.gov/nuccore/LC529905.1…
中国研究チームのこれ
https://ncbi.nlm.nih.gov/nuccore/MN908947.3…
の完コピです。見比べてみて
午前5:47 · 2021年5月1日·Twitter Web App
完コピ?
そこまで言うのならば、見比べてみましょう。
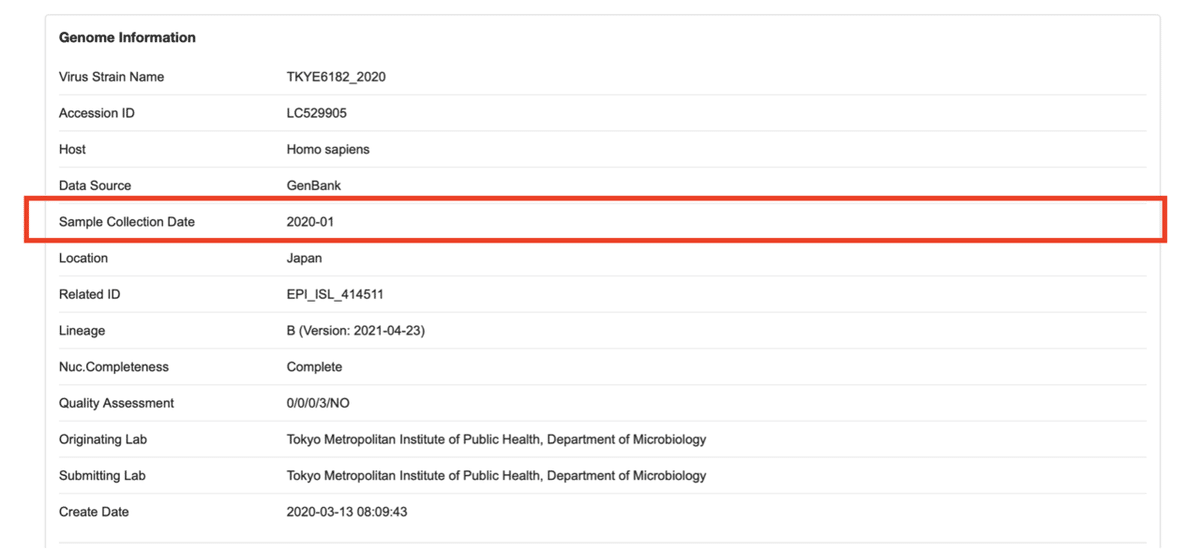
東京都健康安全研究センターの登録したSARS-CoV-2の配列情報
Severe acute respiratory syndrome coronavirus 2 TKYE6182_2020 RNA, complete genome
GenBank: LC529905.1
https://www.ncbi.nlm.nih.gov/nuccore/LC529905.1
復旦大学の研究チームが武漢で最初に分離したSARS-CoV-2(Wuhan-Hu-1株)
Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1, complete genome
GenBank: MN908947.3
https://www.ncbi.nlm.nih.gov/nuccore/MN908947.3
両方とも29903塩基の長さのSARS-CoV-2の遺伝子配列です。
東京都健康安全研究センターの『LC529905』も論文として発表されていますので、こちらを紹介します。
Jpn J Infect Dis. 2020 Jul 22;73(4):320-322. doi: 10.7883/yoken.JJID.2020.137. Epub 2020 Apr 30.
Characteristics of SARS-CoV-2 isolated from asymptomatic carrier in Tokyo
https://www.jstage.jst.go.jp/article/yoken/73/4/73_JJID.2020.137/_article
もちろん論文にも、電子顕微鏡で撮影したウイルスの画像も掲載されています。

(画像:https://www.jstage.jst.go.jp/article/yoken/73/4/73_JJID.2020.137/_pdf/-char/ja)
赤色の四角で囲った上側がWuhan-Hu-1(NCBI Reference sequence)で、下側が東京都健康安全研究センターのLC529905です。
黒色のマークが示すように、2つの遺伝子配列を比較すると、3ヶ所に変異があることが分かりました。(具体的には、15324番目、25810番目、29303番目に変異があることが分かりました。)

(画像:https://www.jstage.jst.go.jp/article/yoken/73/4/73_JJID.2020.137/_pdf/-char/ja)
私も確認のために、『GENETYX』という配列比較ソフトウェアを使って、2つの配列を比較しました。
その結果、一致したのは全29903塩基中29900塩基で、3塩基の違いがあることが分かり、15324番目、25810番目、29303番目に変異があることを再確認することできました。

29303番目、C⇄T(画像最下段)

また、別のデータベースにも『SNP No. 3』という記載があります。
SNPは『一塩基多型』という意味で、一塩基の違い(変異)が何ヶ所あるか示しています。したがって、SNP No. 3は、変異が3ヶ所あることを示しています。

(画像:https://bigd.big.ac.cn/ncov/genome/accession?q=LC529905)
ぜひ実際に、15324番目、25810番目、29303番目の塩基に違いがあるか、2つの配列を比較して自分の目で確かめてください。
Severe acute respiratory syndrome coronavirus 2 TKYE6182_2020 RNA, complete genome
GenBank: LC529905.1
https://www.ncbi.nlm.nih.gov/nuccore/LC529905.1
Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1, complete genome
GenBank: MN908947.3
https://www.ncbi.nlm.nih.gov/nuccore/MN908947.3
『完コピ』は言い逃れできない嘘です!
中国・武漢市から遠く離れた東京で分離されたSARS-CoV-2が、たった3塩基の違いしかないのは不思議に思うかもしれません。

(画像:https://www.jiji.com/jc/article?k=2020060201065&g=int)
論文にはもちろんこのSARS-CoV-2が、どのような人から得られたサンプルであるかが記載されています。
見ると、『中国からの旅行者または帰国者』と書いてあります。
SARS-CoV-2 specific sequences were detected from pharyngeal swabs of three individuals who were travelers or returnee from China,...
訳)SARS-CoV-2特異的な遺伝子配列は、3人の中国からの旅行者または帰国者の咽頭スワブから検出されました。
https://www.jstage.jst.go.jp/article/yoken/73/4/73_JJID.2020.137/_pdf/-char/ja
このコロナ禍で、「RNAウイルスは変異しやすい」ということは、常識のように広く知られるようになりましたが、その変異のしやすさを正確に理解している人は少ないと思います。
コロナウイルスはその他の多くのRNAウイルスと異なり、『3'->5' Exoribonuclease』という変異を修復することができるタンパク質を持っています(SARS-CoV-2ではこのタンパク質は『nsp14』とも呼ばれるものです)。
Proc Natl Acad Sci U S A. 2006 Mar 28;103(13):5108-13. doi: 10.1073/pnas.0508200103. Epub 2006 Mar 20.
Discovery of an RNA virus 3'->5' exoribonuclease that is critically involved in coronavirus RNA synthesis
https://www.pnas.org/content/103/13/5108
下の図は、球の大きさで、そのウイルスのスパイクタンパク質の『遺伝的多様性』を視覚的に表したものです。(球が大きいほど多様性があることを示しています。)

Proc Natl Acad Sci U S A. 2020 Oct 6;117(40):24614-24616. doi: 10.1073/pnas.2017726117. Epub 2020 Sep 21.
Low genetic diversity may be an Achilles heel of SARS-CoV-2
https://www.pnas.org/content/117/40/24614
この中で左端のSARS-CoV-2の球は見えないくらい小さい、すなわち遺伝的多様性が低いことが分かります。
ヒト免疫不全ウイルス1型(HIV-1)やC型肝炎ウイルス(HCV)に対する効果的なワクチンは未だに存在しません。その原因の一つが、スパイクタンパク質の多様性です。
一方で、SARS-CoV-2は多様性が低いことから、例えば、ファイザー社のmRNAワクチン『BNT162b2』は、新しく出現したイギリス変異株B.1.1.7と南アフリカ変異株B.1.351に対する感染リスクをそれぞれ89.5%、75.0%減少させ、重症化リスクをいずれも100%減少させることができました。
驚異的ですね。
N Engl J Med. 2021 May 5. doi: 10.1056/NEJMc2104974. Online ahead of print.
Effectiveness of the BNT162b2 Covid-19 Vaccine against the B.1.1.7 and B.1.351 Variants
https://www.nejm.org/doi/full/10.1056/NEJMc2104974
ウイルスにとって変異は、諸刃の剣です。
変異をすることでヒトの免疫から逃れたり、薬剤の効果を失わせたりすることができる一方で、あまりに変異が起きると自身のタンパク質の機能が損なわれ、複製できなくなってしまいます。
コロナウイルスは、その変異によって自身のタンパク質の機能が損なわれることがないように自身の変異を修復できるExoribonucleaseを持つに至りましたが、そのウイルスにとっての利点が、幸いヒトにとっても1つのワクチンで様々な変異株に対応できる可能性があるという利点になりました。
このExoribonucleaseの働きもあり、SARS-CoV-2の変異率は『~0.8 x10^-3 substitutions per site per year』、SARS-CoV-2の配列を3万塩基とすると、30,000 x0.8 x10^-3 で24、すなわち1年でおよそ24ヶ所に変異が生じると推定されています(2020年8月の時点)。
Curr Biol. 2020 Aug 3;30(15):R849-R857. doi: 10.1016/j.cub.2020.06.031. Epub 2020 Jun 11.
On the evolutionary epidemiology of SARS-CoV-2
https://www.sciencedirect.com/science/article/pii/S0960982220308472
したがって、2019年12月に武漢で最初に登録されたSARS-CoV-2と、2020年の1月に中国から日本に来た人から採取されたSARS-CoV-2の遺伝子配列の違いが3塩基だったということは、特におかしなことではないでしょう。
(画像:https://bigd.big.ac.cn/ncov/genome/accession?q=LC529905)
以上。
嘘つきは地獄に落ちますように。

この記事が気に入ったらサポートをしてみませんか?
