
【R言語】多群比較検定のフォーマットを統一する③Bonferroni補正編
こんにちは。Rで多群比較検定のコードをコツコツ書いているえいこです。
最近は、「多群比較検定のフォーマットを統一する」ということをしています。
と言うのも、この後グラフを描いていく時にpの値をグラフ内に書きたい!って時に検定ごとにp値の引っ張り方が違ったらコードを書くのがめんどくさい。
いっそのこと出力フォーマットを統一してしまえー!ってことで、現在頑張って統一しているところです。
Tukey-KrammerとDunnett検定についてはやってきているので、今回はパラメトリック検定の最後!Bonferroni補正です。
ずーっとR言語の方のnoteの更新が途絶えていたのですが。3000字くらい書いたこの記事がどっかに飛んでいってしまったので、また1から作り直していたためです。まだまだR勉強は終わりませんよー。
では、早速やっていきましょう!
Bonferroni補正の検定結果の中身を再確認してみよう
出力結果を見るためには、Bonferroni補正をしてみないとわからないですね!ってことでコードを打って実行します。
test<-pairwise.t.test(data$value,data$order,
paired=F,pool.sd = F,
p.adjust.method="bonferroni")
test
ほー!マトリックスみたいな形で出力されるんですね!
View関数で中身を見てみましょう...
やっぱりclass関数で確認すると、”matrix”と書かれました。

と言うことで、matrixの形からデータを取り出すことを考えた方が良いと言うことがわかりました。
データの並べ方を再確認
どう言う順番でデータを並べたかったのかをもう一度確認しましょう。(すぐに忘れてしまうので、一回一回確認するのが大事!)

一列目に比較したグループ、二列目にpの値を書いていきます。
一列目のグループは、上からaと何かを比較したもの。それが終わったら、aとの組み合わせ以外で、bとなにかを比較していく...と言う順番。
Bonferroni補正の出力されたmatrixをみてみると、一列目を上から順番に並べるとaと何かを比較したものが網羅できそうです。
次にそれ以外で、bとの比較は二列目。順番に列数が増えて行くのが予想できます。
イメージ的には...

大きい歯車j(列歯車)と小さい歯車i(行歯車)の二つの歯車を組み合わせて回していきます。
このイメージをもとに for文を書いていきます。
for文のイメージを具体的に掴んでいく
はい、じゃぁfor文を書きましょうといきなり言われても無理ですよね。
なので一つ一つ歯車がどうやって移動していくのか確認していきましょう。今回は入れ子状態のfor文を書いていくのですが、Rのfor文は癖があるらしくどうやって回るか簡単なコードを書いて調べるのが良いそうです。
例えば...
for (j in 1:5){
for (i in 2:5){
print(j*i)
}
}こんな感じのコードを書いてみて検証してみましょう。

j=1で、iの歯車が2~5で回っています。同じように、j=2になるとiは2〜にリセットされて5まで回ります。j=3になると、iはまた2~5で回っています。
Rのfor文の回り方が把握できました。
では、実際のマトリックスを使ってfor文を書いていきましょう。
イメージ図はデータ数が多いので、実際に使った2×2のデータを使って考えていきたいと思います。

for文一回転目(j=1, i=1)の時、a vs bのp値を持ってくる
for文二回転目(j=1, i=2)の時、a vs cのp値を持ってくる
for文三回転目(j=2, i=1)の時、b vs bの値はNA
for文四回転目(j=2, i=2)の時、b vs cのp値を持ってくる
今のところNAの値は考えないでfor文を書いていきます。
まずは、大きい歯車jのコードから...
大きい歯車jはマトリックスの左から右に一個ずつ列を移動していきます。1列目からどこまで行くかというと、列数の終わりまで。イメージ図でいうとcがある三行目まで動きます。
Rにはマトリックスの列数を引っ張ってくる関数(ncol())があるので、”ncol()”を使って書いていきましょう。
for (j in 1: ncol(test[[“p.value”]]){
#小さい歯車を回す
}という感じ。
次に、小さい歯車iを回していくことを考えます。
小さい歯車はマトリックスの行を上から下に一個ずつ行を移動していきます。大きい歯車と同じように1から行の終わりまで動きます。
行数を引っ張ってくる”nrow()”関数を使って書いていきます。
for (j in 1: ncol(test[["p.value"]])){
for (i in 1:nrow(test[["p.value"]])){
#pの値を引っ張ってくる
}
}次に、pの値を入れていく空ベクトルの準備と実際にpの値を参照するコードを書いていきます。
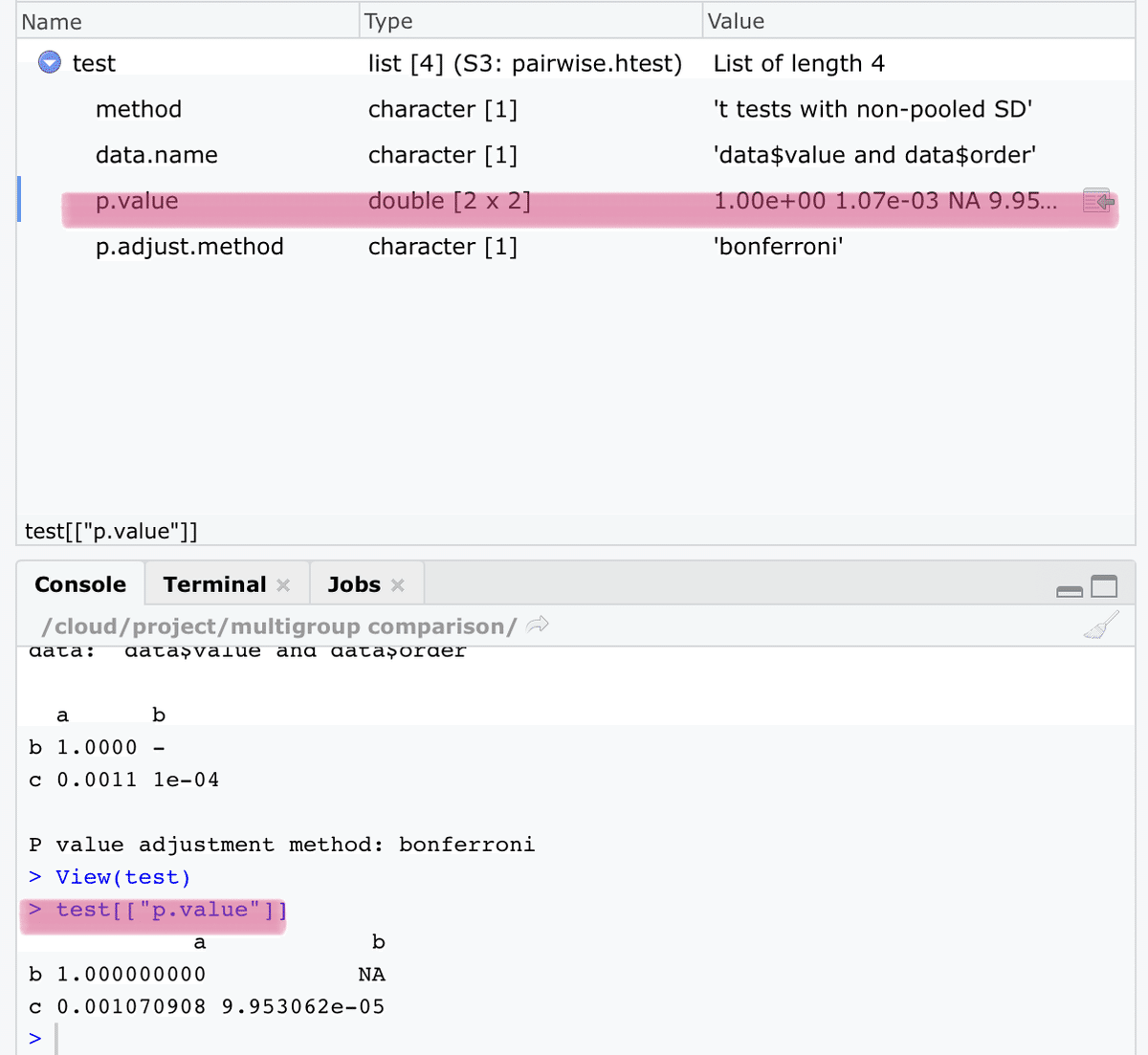
pの値はtest[[“p.value”]]の、[i(行),j(列)]を参照すれば良いので...
p.val <- c() #空のベクトルを用意しておく
for (j in 1: ncol(test[["p.value"]])){
for (i in 1:nrow(test[["p.value"]])){
p.val<- c(p.val, test[["p.value"]][i,j])
}
}こんな感じのコードでOKです。
p.valを実行してみると...

NAの位置が3番目に来ていて、期待通りにfor文が回っていることがわかりました。
後は、群の組み合わせ用のコードを書いていきます。
出力されたtest[[“p.value”]]のマトリックスには行と列にそれぞれ名前がついているのでそれを引っ張ってくれば良いのでは?と思って調べてみると...
行の名前や列の名前を引っ張ってくる関数があるんですねー。列の場合は、”colnames()”、行の場合は”rownames()”という関数です。
実際に使ってみると...
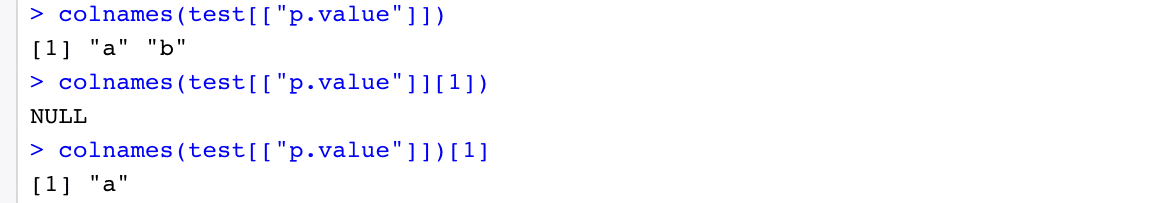
一行目は、”colnames()”関数をはじめて使ってみた時ですが、colnamesというだけあって、全ての列名が出力されました。
それぞれの要素だけを取り出したいと試行錯誤したのが三行目と五行目です。
五行目の結果から、”colnames(test[[“p.value”]])[1]”とすると一列目の名前を引っ張れることがわかりました。これを”rownames()”にも応用して、コードを書いてみると...
p.val<- c()
com<- c()
for (j in 1: ncol(test[["p.value"]])){
for (i in 1:nrow(test[["p.value"]])){
p.val<- c(p.val, test[["p.value"]][i,j])
com<- c(com, c(paste(colnames(test[["p.value"]])[j],
rownames(test[["p.value"]])[i],sep =":")))
}
}p.valという変数にp値が、comという変数に群の組み合わせが入っています。
出力結果をデータフレームにする
後はお馴染み、出力結果をdata.frameの形にしていくだけです。
df<- data.frame(comb=com, p.val=p.val)で、作ったdfを実行すると...

あ、、、そうでした。3番目はb vs bとなっていてpの値がNAだったんです。
NAだけを取り除く関数って有ったような...
そうそう、Steel検定のコードを書くときの最初のデータのクオリティチェックのところで”complete.cases”という関数を使ってたんでした!
今回も”complete.cases”を使ってNAが入っている行を削除しておきましょう。
df <-df[complete.cases(df),]dfを実行すると...

完成しました!
ふぅ〜、第二の難所クリアです。
後は、ノンパラメトリックの出力結果を揃えて、グラフを書くコードを書いていきたいと思います。
それでは、また!
最後までお読みいただきありがとうございます。よろしければ「スキ」していただけると嬉しいです。 いただいたサポートはNGS解析をするための個人用Macを買うのに使いたいと思います。これからもRの勉強過程やワーママ研究者目線のリアルな現実を発信していきます。
