Pythonでピークフィッテイング
こんにちは!CheMLです.
今回はPythonを使ったピークフィッテイングについての紹介です.
ピークフィッテイングができるソフトやプログラムはたくさんありますが,もちろんpythonでも可能です.Excelのソルバーでも簡単に実装できますが,速度や使いやすさの観点ではpythonの方が断然おすすめです!それでは早速フィッティングを行っていきましょう.
Gaussianフィッティング

今回はXPS(X線光電子分光)のスペクトル(酸素1s軌道)を用意しました.scipy(※1)のoptimizeパッケージに入っているcurve_fit()という関数を使ってGaussianフィッティングを行います.使い方は簡単です.curve_fitの中身に必要な情報を引数として与えるだけでフィッティングができちゃいます.
curve_fit(関数, 入力データx, 入力データy, [初期値])
ちなみにcurve_fit関数の最適化アルゴリズムにはlevenberg-marquardt法というものが使われています.これはGauss-Newton法と最急降下法を組み合わせた手法で,現在幅広く用いられている最適化の手法です.levenberg-marquardt法については今後詳しく解説する予定です.
curve_fitは戻り値としてフィッティングされたパラメータと係数の共分散行列を返します.そのため,空の変数はp_optとp_covの2つ用意しています.あとは結果のプロットと決定係数の計算,そして結果の出力(csv)です.決定係数の計算は手動でやっていますが面倒な方はpandasのcov()メソッドやscikit-learnのmetrics.r2_score()を使ってもいいと思います.
以下がソースコードです.
import numpy as np
import pandas as pd
from scipy.optimize import curve_fit
import matplotlib.pyplot as plt
#データ読み込み---------------------------------------------------------
sample_data = pd.read_csv('sample.csv', skiprows = 4, header = None)
x = sample_data.iloc[:, 0]
y = sample_data.iloc[:, 1]
#関数の定義------------------------------------------------------------
def gaussian_func(x, mu, sigma, A, s):
g = np.exp((-(x-mu)**2)/(2*sigma**2))
g_scaled = A * g + s
return g_scaled
#パラメータ初期値の設定---------------------------------------------------
mu0 = 532.5
sigma0 = 4
A0 = 1400
y0 = 0
initial_param = [mu0, sigma0, A0, y0]
#フィッティング------------------------------------------------------------
p_opt, p_cov = curve_fit(gaussian_func, x, y, p0 = initial_param)
#フィッテイングされた係数とその95%信頼区間の計算---------------------------------
var = np.diag(p_cov)
stdev = np.sqrt(var)
CI_95 = 2 * stdev
FWHM = 2 * np.sqrt(2 * np.log(2)) * p_opt[1]
#フィッテイングデータのプロット-------------------------------------------------
xd = np.linspace(min(x), max(x), 500)
estimated = gaussian_func(xd, p_opt[0], p_opt[1], p_opt[2], p_opt[3])
plt.plot(xd, estimated, label="Estimated curve", color="red")
plt.scatter(x, y, label="raw data", color="blue")
plt.xlabel("Binding energy / eV")
plt.ylabel("Counts")
plt.xlim(525, 540)
plt.legend()
plt.savefig('fit-plot.png', Transparent=True, bbox_inches="tight")
plt.show()
#決定係数の計算--------------------------------------------------------
def estimated_value(x_raw):
return gaussian_func(x_raw, p_opt[0], p_opt[1], p_opt[2], p_opt[3])
error_sum = 0
for i in range(len(x)):
error = (y[i] - estimated_value(x[i]))**2
error_sum = error_sum + error
R2 = 1 - (error_sum / (np.var(y) * (len(x)-1)))
#各種パラメーターの表示----------------------------------------------------
p_opt = p_opt.astype('float16')
CI_95 = CI_95.astype('float16')
estimated_params = pd.DataFrame(
[[p_opt[0], p_opt[1], p_opt[2], p_opt[3], FWHM, R2], [CI_95[0], CI_95[1], CI_95[2], CI_95[3]]]
).T
estimated_params.index = ['mu', 'sigma', 'A', 'y0', 'FWHM', 'R2']
estimated_params.columns = ['Value', '95% CI']
print(estimated_params)
#解析結果の出力------------------------------------------------------------
output_data = pd.DataFrame([x, y]).append(pd.DataFrame([xd, estimated])).T
output_data.columns = ['raw_x', 'raw_y', 'estimated x', 'estimated y']
output_data.to_csv('estimated_data.csv', index = False)
estimated_params.to_csv('estimated_params.txt', sep = '\t')以下が実行結果です.savefig()で得られたpng画像を載せました.きれいにフィッテイングできていますね.

(ちなみに高解像度な画像を得るにはオプションにdpi=300を追加するといいです.300くらいで論文・レポートには十分だと思います.Transparent=Trueとすることで背景が透明になります.bbox_inches="tight"は画像サイズをグラフサイズに合わせるためのオプションです.)
各種パラメーターは以下にように表示されました。決定係数は0.9962と良好にフィッテイングできていることが分かります。
Value 95% CI
mu 532.500000 0.015221
sigma 1.420898 0.016678
A 1409.000000 13.507812
y0 -8.914062 4.785156
FWHM 3.345671 NaN
R2 0.996224 NaN擬Voigtフィッティング
以上ではpythonでGaussianフィッティングする方法を紹介しました.しかし実際にはXPSのスペクトルはハイゼンベルグの不確定性幅(寿命に関する線幅,τは寿命,ℏはディラック定数)
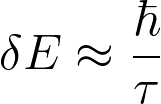
を持つためGaussianとLorentzianを畳み込んだVoigt関数を使用すべきです.(*Voigt関数は収束性は悪いため畳み込みの代わりにGaussianとLorentzianを足し合わせた擬Voigt関数を用いることが多いです.)
上記のソースコードを以下のように改造すれば擬Voigt関数でのフィッティングが行えます.
import numpy as np
import pandas as pd
from scipy.optimize import curve_fit
import matplotlib.pyplot as plt
#データ読み込み---------------------------------------------------------
sample_data = pd.read_csv('sample.csv', skiprows = 4, header = None)
x = sample_data.iloc[:, 0]
y = sample_data.iloc[:, 1]
#関数の定義------------------------------------------------------------
def Voigt_func(x, mu, sigma, A, s, p):
g = np.exp((-(x-mu)**2)/(2*sigma**2)) * p
l = (sigma**2 / (((x-mu)**2) + sigma**2)) * (1-p)
v_scaled = A * (g + l) + s
return v_scaled
#パラメータ初期値の設定---------------------------------------------------
mu0 = 532
sigma0 = 2
A0 = 1000
y0 = 2
p_gauss = 0.8
initial_param = [mu0, sigma0, A0, y0, p_gauss]
#フィッティング------------------------------------------------------------
p_opt, p_cov = curve_fit(Voigt_func, x, y, p0 = initial_param, bounds=([-np.inf, -np.inf, -np.inf, -np.inf, 0], [np.inf, np.inf, np.inf, np.inf, 1]))
#フィッティングされた係数とその95%信頼区間の計算---------------------------------
var = np.diag(p_cov)
stdev = np.sqrt(var)
CI_95 = 2 * stdev
#結果のプロット-----------------------------------------------------------
xd = np.linspace(min(x), max(x), 500)
estimated = Voigt_func(xd, p_opt[0], p_opt[1], p_opt[2], p_opt[3], p_opt[4])
plt.plot(xd, estimated, label="Estimated curve", color="red")
plt.scatter(x, y, label="raw data", color="blue")
plt.xlabel("Binding energy / eV")
plt.ylabel("Counts")
plt.xlim(525, 540)
plt.legend()
plt.savefig('fit-plot.png', Transparent=True, bbox_inches="tight")
plt.show()
#決定係数の計算--------------------------------------------------------
def estimated_value(x_raw):
return Voigt_func(x_raw, p_opt[0], p_opt[1], p_opt[2], p_opt[3], p_opt[4])
error_sum = 0
for i in range(len(x)):
error = (y[i] - estimated_value(x[i]))**2
error_sum = error_sum + error
R2 = 1 - (error_sum / (np.var(y) * (len(x)-1)))
#各種パラメーターの表示----------------------------------------------------
p_opt = p_opt.astype('float16')
CI_95 = CI_95.astype('float16')
estimated_params = pd.DataFrame(
[[p_opt[0], p_opt[1], p_opt[2], p_opt[3], p_opt[4], R2], [CI_95[0], CI_95[1], CI_95[2], CI_95[3], CI_95[4]]]
).T
estimated_params.index = ['mu', 'sigma', 'A', 'y0', '%gauss', 'R2']
estimated_params.columns = ['Value', '95% CI']
print(estimated_params)
#データの出力------------------------------------------------------------
output_data = pd.DataFrame([x, y]).append(pd.DataFrame([xd, estimated])).T
output_data.columns = ['raw_x', 'raw_y', 'estimated x', 'estimated y']
output_data.to_csv('estimated_data.csv', index = False)
estimated_params.to_csv('estimated_params.txt', sep = '\t')以下が実行結果です.Gaussianフィッティングとほとんど差異がありません.

Value 95% CI
mu 532.500000 0.015259
sigma 1.420898 0.017044
A 1409.000000 19.921875
y0 -8.914062 7.589844
%gauss 1.000000 0.077148
R2 0.996224 NaNGauss関数の割合が1.00にフィッティングされていて,全くLorentzianの寄与がないことが分かります.なおGaussianの割合は使用した装置によってかなり異なるので注意が必要です.
まとめ
以上,今回はscipyのcurve_fitを用いたピークフィッテイングでした!
化学実験のデータ解析では頻繁に使える手法ですので,ぜひ有効活用してみてください.
ここまで読んでいただいて、ありがとうございます!ご意見・ご感想はコメント欄,またはこちらまで.フォロー&スキしていただけると張り切って投稿できます(笑)
実行環境
Spyder(anaconda3)
python 3.7.9
windows10
*2020年9月現在では最新版のpython3.8.5に対応していないライブラリも多いため,計算化学ユースにおいてはpython3.7の使用をお勧めします。
※1 Scipy: 科学技術計算用pythonライブラリ(今回のようなフィッティングだけでなくスムージングや関数の微分積分など幅広い演算が可能です。本noteでも多用します。)
★筆者のつぶやき★
「XPSスペクトル」はスペクトルを2回言ってるから違和感あるな~
この記事が気に入ったらサポートをしてみませんか?