
機械学習で創薬探索のプロセスをかじってみました(パート3:データーセットの準備と機械学習モデルの構築)
バイオインフォマティクスに疎い私ですが、創薬探索のプロセスを経験すべく、機械学習で化合物のデータを用いた分析をしてみました!
パート1、2に引き続き、Data Professor という名前でユーチューバーをされているタイ人でバイオインフォマティクスの元准教授の Chanin Nantasenamat さんのビデオ Bioinformatics Project from Scratch - Drug Discovery Part 3 (Dataset Preparation) と Bioinformatics Project from Scratch - Drug Discovery Part 4 (Model Building) の内容に沿って分析しました。説明欄にビデオで使用されているコードのリンクも掲載されています。なお、ここで記述したコードは、自らがやりやすいように一部変更を加えていますのでご了承下さい。
概要
エストロゲン受容体 α1(estrogen receptor alpha 1) に対する阻害活性が調べられている化合物の化学構造の特徴を Pubchem フィンガープリントとして取得する。
pIC50 値(エストロゲン受容体 α1 に対する阻害活性のレベル)を目的変数(Y)、および化合物の Pubchem フィンガープリントのデータを説明変数(X)とする、エストロゲン受容体 α1 に対する阻害活性を推測するための学習モデルを構築する。
開発環境
MacBook Air
Google Colaboratory
Python 3.6.9
まず、Bioinformatics Project from Scratch - Drug Discovery Part 3 (Dataset Preparation) の内容に沿ってご説明します。
ライブラリーとデータの準備
PaDEL-Descriptor のダウンロード
それぞれの化合物の特徴は、化学構造に基づいて数値で表わす(分子記述子)ことができるのですが、PaDEL-Descriptor は、その数値を計算するためのオープンソースソフトウェアです。これを用いて、特定の部分構造の有無を正規化したベクトル 0と1で表す Pubchem フィンガープリントを取得します。
PaDEL-descriptorに関する論文は以下になります。
分子記述子やフィンガープリントに関しては以下の記事を参考にしました。
Data professor さんの GitHub からPaDELをダウンロードします。
! wget https://github.com/dataprofessor/bioinformatics/raw/master/padel.zip
! wget https://github.com/dataprofessor/bioinformatics/raw/master/padel.shダウンロードした Zip を unzip します。
! unzip padel.zipデータの準備
Pubchem フィンガープリントを計算するのに必要な、化合物のSMILES 表記の情報を準備します。前回のパート2で作成した、エストロゲン受容体 α1に対する阻害活性が調べられている ChEMBL データベースの化合物のデータファイルをダウンロードします。
import pandas as pd
df3 = pd.read_csv('estrogenRa_bioactivity_data_pIC50.csv')
df3
化合物の化学構造の情報が格納されたSMILES 表記のカラムとchembl IDのカラムのみを取り出し、csv形式で保存します。
selection = ['canonical_smiles','molecule_chembl_id']
df3_selection = df3[selection]
df3_selection.to_csv('molecule.smi', sep='\t', index=False, header=False)保存したデータの最初の五行を確認します。
! cat molecule.smi | head -5
データの行数も確認します。
! cat molecule.smi | wc -l3543
PaDEL descriptors によるフィンガープリントの計算
化合物の詳細な特徴をPubchem フィンガープリントとして取得します。
パート2では、lipinski 関数を用いて化合物の特徴の情報(分子量、疎水性、水素結合供与体、水素結合受容体)を得ました。こちらは経口薬として相応しいか判断するための全体的な特徴を分析したものでした。対して Pubchem フィンガープリントは、より小さな局所的な部分の特徴まで表したものになっています。化合物をレゴブロックで組み立てた構造物であるとすると、一つ一つのブロックの特徴まで分析していると説明できます。
この辺りの説明は Bioinformatics Project from Scratch - Drug Discovery Part 4 (Model Building) でされています。
まず、PaDELを実行する前に、PaDELファイルの中身を確認してみましょう。
! cat padel.shjava -Xms1G -Xmx1G -Djava.awt.headless=true -jar ./PaDEL-Descriptor/PaDEL-Descriptor.jar -removesalt -standardizenitro -fingerprints -descriptortypes ./PaDEL-Descriptor/PubchemFingerprinter.xml -dir ./ -file descriptors_output.csv
この各ファイルの説明は以下になります。
-jar
Java で稼働する jar ファイルを使用するのに必要です。
-Djava.awt.headless=true
このオプションはGoogle Colab でも表示可能にするために加えてあります。
-removesalt
化学構造に含まれているNa-Cl などの塩や小さい有機酸を自動的に除きます。
-standardizenitro
上述の不必要な化学構造を除去する方法が書かれています。
-fingerprints
フィンガープリントを計算するためのプログラム
descriptortypes ./PaDEL-Descriptor/PubchemFingerprinter.xml
フィンガープリントのタイプ、PubchemFingerprintを指定しています。PaDELはほかにも十種類以上のフィンガープリントを扱えるようです。
descriptors_output.csv
結果を格納するファイル
では、実際にPaDEL を実行します。私の3543のデータでは17分かかりました。
! bash padel.sh
作成されたファイルのリストを確認します。
! ls -l
説明変数X と目的変数 Yのデータの準備
説明変数 X (Pubchem フィンガープリント)
descriptors_output.csv ファイルとして出力された Pubchem フィンガープリント の結果を見てみましょう!化学構造の881 種類の特徴の有無を0と1で表したデータが出力されました。
df3_X = pd.read_csv('descriptors_output.csv')
df3_X
特徴の種類が多すぎるので、ここでは13番目の特徴までを載せています。
この分野は私にとっては専門外のため、一つ一つのカラムがどのような化学構造を示しているのか等のデータの内容の詳細までは踏み込めないのですが、ご了承ください(^_^;)。
Name カラムにあるChembl IDの情報は必要ないので除きます。
df3_X = df3_X.drop(columns=['Name'])
df3_X
目的変数 Y (pIC50)
パート2で作成した estrogenRa_bioactivity_data_pIC50.csv ファイルから、エストロゲン受容体 α1 に対する阻害活性を表すpIC50 値の情報だけを取得します。
df3_Y = df3['pIC50'] #pIC50の情報だけとる
df3_Y
X と Y の結合
以降の解析で扱いやすくするため、 X と Y のデータを結合して一つにまとめます。
dataset3 = pd.concat([df3_X,df3_Y], axis=1)
dataset3

この結合したデータを新しいファイル名で csv 形式で保存します。
dataset3.to_csv('estrogenRa_bioactivity_data_pIC50_pubchem_fp.csv', index=False)ここからは、Bioinformatics Project from Scratch - Drug Discovery Part 4 (Model Building) の内容に沿ってご説明します。
回帰分析モデルの構築
pIC50 (ターゲット分子に対する阻害活性)を目的変数(Y)、および化合物の科学的特徴を表す Pubchem フィンガープリントを説明変数(X)として、機械学習モデルを構築します。今後調べたい化合物を、既知のデータで学習したこのモデルを用いて検証することで、阻害活性がどれくらいあるかを推測することができるのです。
必要なライブラリーをインポートします。
import pandas as pd
import seaborn as sns
from sklearn.model_selection import train_test_split
from sklearn.ensemble import RandomForestRegressor上で保存した estrogenRa_bioactivity_data_pIC50_pubchem_fp.csv ファイルを読み込みます。
df = pd.read_csv('estrogenRa_bioactivity_data_pIC50_pubchem_fp.csv')不要な pIC50 のカラムを除き、X とします。
X = df.drop('pIC50', axis=1)
X

pIC50 のデータのみを持つ Y を準備します。
Y = df.pIC50
Y
X と Y の次元の確認
X.shape(3543, 881)
Y.shape(3543,)
低分散の特徴の削除
Pubchem フィンガープリントの特徴の中には、化合物間でほとんど差がない(分散がない)ものも存在します。その特徴は学習には適していないので、次のコードで除きます。
from sklearn.feature_selection import VarianceThreshold
selection = VarianceThreshold(threshold=(.8 * (1 - .8)))
X = selection.fit_transform(X)除いた後、次元を確認します。特徴の種類の数が 881から 138 に大幅に減少しました。
X.shape(3543, 138)
トレーニングとテスト用のデータセットの準備
やっと、ここまできました!
X と Y をそれぞれ8 対 2 に分割し、トレーニングデータセット、テストデータセットとします。トレーニングデータセットを用いて学習モデルを構築後、テストデータセットを構築したモデルに分析させて正解率を確認します。
X_train, X_test, Y_train, Y_test = train_test_split(X, Y, test_size=0.2)X と Y それぞれのトレーニングデータ、テストデータの次元を確認します。2834:709 ≒ 8:2 に分けられているのが分かりますね。
X_train.shape, Y_train.shape((2834, 138), (2834,))
X_test.shape, Y_test.shape((709, 138), (709,))
ランダムフォレストによる回帰モデルの構築
ランダムフォレストとは、複数の決定木を用いて学習を行い、それぞれの出力の多数決を予測値とするアンサンブル学習モデルです。ランダムフォレストに関する詳細は、以下の記事で分かりやすく説明されています。
まず、ランダムフォレストを用いてトレーニングデータを学習させます。次に、学習済みモデルでテストデータを分析させて、どのくらい正しくpIC50 値を予測できているかを確認します。その評価には、決定係数R^2を用います。決定係数は、推定された回帰式が、実際のデータに対してどのくらいピッタリ当てはまっているか(当てはまりの度合い)を表しています。値が1に近いほどその度合いは高いということになります。
決定係数に関しては以下の記事が参考になります。
では実際にやってみましょう。
import numpy as np
np.random.seed(100) # 実行に度に同じ乱数が使われるようにする。
model = RandomForestRegressor(n_estimators=100) # n_estimators は決定木の数
model.fit(X_train, Y_train) # トレーニングデータで学習モデルの構築
r2 = model.score(X_test, Y_test) # テストデータを用いて決定係数を算出
r20.6263753585311719
むむ.. 算出された決定係数は高いとも低いとも言えない値ですね ….
テストデータと予測データ間でpIC50 値を比較
テストデータと予測データで、pIC50 値 がどれだけ一致しているかを散布図で可視化してみます。
そのためにまず、テストデータを使った学習済みモデルによる pC50 阻害活性の予測値 Y_pred を取得します。
Y_pred = model.predict(X_test)そして、pC50 阻害活性の実測値 Y_test と予測値 Y_pred を散布図上で比較してみます。
import seaborn as sns
import matplotlib.pyplot as plt
sns.set(color_codes=True)
sns.set_style("white")
ax = sns.regplot(Y_test, Y_pred, scatter_kws={'alpha':0.4})
ax.set_xlabel('Experimental pIC50', fontsize='large', fontweight='bold')
ax.set_ylabel('Predicted pIC50', fontsize='large', fontweight='bold')
ax.set_xlim(0, 12)
ax.set_ylim(0, 12)
ax.figure.set_size_inches(5, 5)
plt.show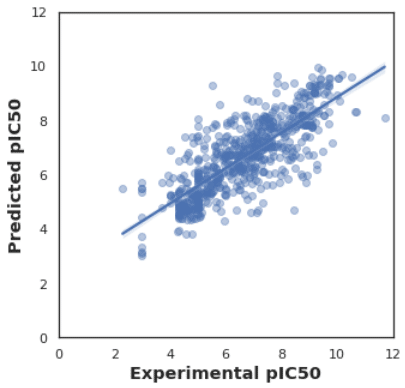
データを見る限り、そこそこは予測ができているようにはみえますが..
最後に
今回でようやく機械学習モデルの構築までたどり着くことができました!
次回のパート4では、同じデータを用いて他の機械学習モデルの構築も試み、モデル間で結果を比較してみたいと思います。
この記事が気に入ったらサポートをしてみませんか?
