
【高校生物】遺伝子⑤「どのようなバイオテクノロジーがあるのか?」
~プロローグ~
1993年にノーベル化学賞を受賞したキャリー・マリスは、当時のガールフレンドを乗せてハイウェイをドライブしている時に、PCR法の原理をひらめいた。DNAの二本鎖は、加熱すれば一本鎖に分離する。二つのプライマーを両端につなげば、その間の領域は酵素によって複製されるだろう。今度は新しくできたDNAの二本の鎖をほどいて一本にする。再びプライマーを結合させれば、酵素はもう一度新しい鎖の上で働き始めるだろう。2本のコピーから4本ができ、次のサイクルではそれが8本になり・・・増幅はどんどん続いていくだろう。ちなみに、ノーベル化学賞の受賞が決まった時、マリスはサーフィン中であった。
「良いアイディアというものは、思ってもみない時に浮かぶことも、たまにはあるんだ。」キャリー・マリス
★テストに出やすいワード
①制限酵素
②DNAリガーゼ
③ベクター
④PCR法
⑤電気泳動法
要点:PCR法は、DNA複製を繰り返してDNAを増幅する方法である。
● PCR法(PCR=ポリメラーゼ連鎖反応、polymerase chain reaction)は、DNAの特定の領域を増幅する方法である。
①DNAを約95℃で処理⇒ 塩基同士の水素結合を切り、二本鎖を一本鎖にほどく。
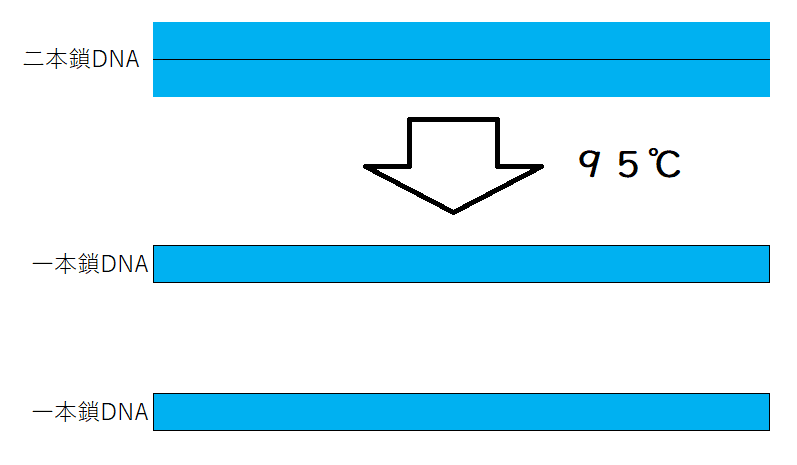
②約60℃で、増幅させたい配列の両端にプライマーを結合させる。

③約70℃で、DNAポリメラーゼを働かせ、DNAを複製させる。

④生じた二本鎖DNAを再び95℃でほどき、同じサイクルを繰り返す。
*あらかじめ、増幅させたい領域を含むDNA、プライマー、4種のヌクレオチド(DNAの材料。4種あるのは、塩基が4種あるから)、DNAポリメラーゼなどを加えておけば、温度変化は装置が勝手に繰り返してくれる。
*ヌクレオチドには、DNAの材料であるデオキシヌクレオシド三リン酸(デオキシリボースと塩基とリン酸3つが結合したヌクレオチド)を用いる。dNTPと表記される(Nは「AかTかCかGが入りますよ」という記号。dATP、dTTP、dCTP、dGTPの4種が使われる。先頭のdは、「デオキシリボース」を表している)。
★PCR法では、好熱菌由来の、高温でも失活しないDNAポリメラーゼを用いる。(非常によく問われる!)
*下の講義では少し詳しくPCR法の過程を解説している(ふつう増幅させたい領域はDNAの途中にある)。
講義動画【PCR法】
講義動画【PCRを用いた感染症の検査】
(RNAを遺伝物質としてもつウイルスの検出)
講義動画【プライマーを設計する問題】
雑談:プライマーが短すぎると、色々な場所に結合してしまう。実際、上のの動画のように4塩基しかない短いプライマーを使ってしまうと、DNAの色々なところにベタベタとプライマーげ結合し、様々な副産物が生じてきてしまう。ただ、異常に長いプライマーを作ると、DNAと結合する確率が減り、産物の収量が下がる(プライマーのサイズが大きいほどアニーリング効率は低下する)。それに、長いプライマーは高価である。
雑談:PCRの温度の設定は実験条件によって異なる(しかし、困ったことに、入試で温度が問われることがある。一応教科書の値を覚えておこう)。
要点:電気泳動法は、長さの異なるDNAを分けるための方法である。
● 電気泳動法:ゼリー状のゲルにDNAを注入し、電場をかける。DNAはリン酸部分が負(-)に帯電しているので、+の方向に進む。
(DNAがどちらの方向に進むのかはよく問われる。)

● 分子が大きい(つまりDNAが長い)ほうがゲルの中を進みにくく、移動速度が遅いので、DNAを長さごとに分けることができる。
雑談:あらかじめ長さがわかっているDNA(マーカーという)を購入し、一緒に電気泳動を行えば、マーカーのバンドとサンプルのバンドの位置を比べることで、長さ未知のDNAの長さを特定できる。
講義動画【電気泳動法】
雑談:電気泳動像は下の写真のような感じ。これは大学時代にRT-PCRの産物を流したもの(mRNAを逆転写してDNAにしたものを電気泳動に流したもの)。一番下がネガティブコントロール。ーRTは逆転写しなかったという意味(産物があるはずないので、バンドが出るはずない)。中段はポジティブコントロール(ヒストンをコードする遺伝子由来の産物。どの細胞でも基本的に発現しているので、必ずバンドが出るはず。このバンドが出なければ実験は失敗している)。アフリカツメガエルの初期胚に、ある遺伝子のmRNAを注入し、BMP4をコードする遺伝子の発現がどう変化したかを見ている(写真は実際の実験結果の一部)。バンドが少しがたついているのがわかるだろうか。これは僕が実験が下手だったからである。

挿絵:電気泳動と研究者

要点:DNAは制限酵素とDNAリガーゼを使うことで切り貼りできる(遺伝子組換えを行うことができる)。
雑談:ちなみに、一般に、「遺伝子組み換え」ではなく、「遺伝子組換え」と書く。
(1)制限酵素
● 制限酵素は、二本鎖DNAの特定の配列を認識し、切断する酵素である(認識配列と切断部位が制限酵素ごとに決まっている)。はさみのような役割を持つ。
雑談:下図は制限酵素と認識配列の例。下図の制限酵素は、「エコアールワン」「バムエイチワン」と読む。認識配列、切断部位は問題文に示されるので覚えなくてよい。たとえば、EcoRⅠは、「5'GAATTC3'(3'CTTAAG5')」の配列を認識し、図の点線のところでDNAを切断する。

発展:制限酵素の切り口
異なる制限酵素で切った切り口同士を連結させた場合、配列が変わり、切り出しに使った制限酵素では切れなくなることもある。
たとえば、制限酵素であるSalⅠとXhoⅠの認識配列は下図のとおりであるが、それぞれの制限酵素で切ったものを結合すると、もうどちらの制限酵素でも切れなくなる(どちらの制限酵素の認識配列とも異なる配列が出来上がる。端のCとGに注目)。

(2)DNAリガーゼ
● DNAリガーゼは、2つの2本鎖DNAをつなぎ合わせるはたらきをもつ。のりのような役割を持つ。
雑談:DNAリガーゼは、DNA連結酵素とも呼ばれ、隣接したDNA鎖の3'-OH末端と、5'-リン酸末端をリン酸ジエステル結合で連結する酵素である。

(3)遺伝子組換え
● 遺伝子を運ぶもの(遺伝子部分のDNAを入れておくもの)をベクターという。ベクターにはプラスミドという細菌のもつ小さな環状DNAが使われることが多い(他にも、ウイルスがベクターとして使用されることも多い)。
雑談:「ベクター」という語はもともと「運ぶもの」を意味する。数学・物理学の「ベクトル」も同じ語源である。
雑談:厳密には、ベクターは「組換えDNA実験において制限酵素などにより切断したDNAの断片をつないで増幅させるために用いる自律的増殖能力をもつ(宿主細胞内で増えることができる)小型のDNA分子」のこと。
雑談:細胞内で複製し、安定に娘細胞に分配されること、細胞内から容易に回収できることなどが良いベクターの条件である。
<Q.ベクター=プラスミドと思っていい?…厳密には、あまりよくない。ベクターは「遺伝子の乗り物」みたいなイメージ。ベクターには、プラスミド(原核細胞がもつ環状の小さなDNA)の他にも、ウイルスなどが使える。ベクターが「乗り物」、プラスミドが「自転車」みたいなイメージ。プラスミドの他にも、いろいろなベクターがある。>
● 以下のような手順で大腸菌に生物Aの遺伝子Aを導入し、生物Aのタンパク質Aを大腸菌につくらせることができる。
①生物Aの遺伝子Aを制限酵素で切り出す(DNA断片を得る)。
②遺伝子の運び屋(ベクター)として使うプラスミド(原核生物がもつ小さな環状DNA)を①と同じ制限酵素で切る。
③①で得たDNA断片と②のプラスミドを、DNAリガーゼでつなぐ。
雑談:遺伝子Aが乗ったDNAのような、プラスミドに組込もうとするDNA断片を「インサート」と呼ぶ。DNAリガーゼによってインサートとプラスミドがくっつくことを「ライゲーション」(DNA同士が繋がる反応)と呼ぶ。
④③のプラスミドを大腸菌に導入する。大腸菌は、我々が導入した遺伝子Aを発現し、タンパク質Aを合成する。また、大腸菌は、我々が導入したプラスミドごと分裂してくれるので、簡単に遺伝子を増幅できる。

● 外来遺伝子が導入された生物をトランスジェニック生物という(少し狭い定義だがトランスジェニック生物を「単離した遺伝子を胚的な細胞に注入したとき、その遺伝子を新たに染色体に組み込んだ生物個体のこと」と定義することも多い)。
講義動画【遺伝子組換え】
発展:組み込まれる遺伝子の向き
遺伝子には決まった方向性がある(開始コドンのある方→終止コドンのある方という向きがある)。遺伝子(DNA断片)をプラスミドに組み込む際は、方向性にも注意する。逆向きに遺伝子が組み込まれてしまうと、そこから正常なタンパク質は生産されない。
1種類の制限酵素で組み込みたいDNA断片とプラスミドを切った場合、(切り口が同じなので)DNA断片が逆向きに組み込まれてしまう可能性がある。

面倒だが、2種類の制限酵素を使って、DNA断片の両端の塩基配列を変えれば(そしてプラスミドもその2種類の制限酵素で処理し、切り口をDNA断片が特定の向きに入るようにすれば)、上のような失敗はなくなる。

講義動画【DNAの組み込まれる向き】
<Q.どうして大腸菌にヒトのDNAを組込むの?意味はあるの?…たとえば、ヒト成長ホルモン遺伝子を導入したプラスミドを大腸菌の中に入れて大腸菌を増殖させると、大腸菌は自分の持つ染色体DNAのほかに、あなたが導入したプラスミドも複製してくれるので便利である。このように、『DNA断片を増幅させる操作』をクローニングという。大腸菌にヒトの遺伝子を導入し、ヒトのタンパク質を転写・翻訳によってつくらせることも可能である(その際は、大腸菌などの細菌にスプライシングのしくみが無いことに注意。スプライシングが起こってはじめて正常なタンパク質の情報を持つような場合は、あらかじめスプライシングが起きた後の成熟mRNAを逆転写してcDNAとし、それをプラスミドに組み込む必要がある)。>
講義動画【cDNA】
<Q.制限酵素って何を「制限」しているの?…元々は細菌の持つ、ファージ(ウイルス)DNAを破壊し「ファージの生育を制限する」酵素である。それを人類がバイオテクノロジーに利用している(細菌は自分のDNAが切断されないよう、自身のDNAの一部をメチル化している。メチル化されたDNA領域は制限酵素から守られることが知られている)。制限酵素の名前はどの細菌に由来しているかを表している。たくさんの種類(認識・切断配列がそれぞれ異なる)がカタログになっている。研究者は、自身が切断したい配列に合わせて制限酵素を選ぶ(遺伝子の切り出しに使う制限酵素は、切断できれば何でもいいわけではない。たとえば、大切な遺伝子も切断してしまうような認識配列を持つ制限酵素は使えない)。>
発展:回文配列
制限酵素の認識配列は回文配列になっていることが多い。

<Q.回文配列って何?…たとえば、2本鎖の一方の鎖が3’AAAA5’という配列だったとすると、もう一方の鎖の配列は、全然違う5’TTTT3’である。しかし、回文配列の場合(たとえば上の図のEcoRⅠの認識配列を見よ)、3’GAATTC5’という配列の、もう一方の鎖の配列を見てみると、5’CTTAAG3’となっている。3’側から読むと、全く同じ配列になっている。もともと、回文は、前から読んでも後ろから読んでも同じ文という意味。「たけやぶやけた」などが回文である。>
発展:塩基配列の出現確率
n塩基対の配列の出現確率
①回文配列ではない場合
{(1/4)のn乗}×1/2
②回文配列の場合
(1/4)のn乗
講義動画【塩基配列の出現確率】
要点:特殊なプラスミドを用いることで、遺伝子組換えが起きたDNAが導入された大腸菌を簡単に選別することができる。
● 特殊なプラスミドを用いることで、遺伝子組換えが起きたDNAが導入された大腸菌を簡単に選別することができる。たとえば、ブルー・ホワイトセレクション(青/白スクリーニング blue/white screening )という方法が有名で、よくテストに出る。以下にブルー・ホワイトセレクションの流れを記す。
雑談:「目的とする特定の性質をもつ物質・生物などを、特定の操作で多数の中から選りすぐること」を、一般にスクリーニングと呼ぶ。
・遺伝子Aを大腸菌に導入することを目的とする。
(大腸菌の分裂とともにその遺伝子が複製されるので、たとえば貴重な遺伝子を増幅させることができる。また、大腸菌が遺伝子Aを発現し、遺伝子Aから発現したタンパク質を大量に得ることができる。)
・何の工夫もしないでプラスミドを切って、遺伝子Aをつなぐ操作を行い、大腸菌に導入する操作を行っても、プラスミドを受け取らなかった大腸菌や、遺伝子Aが組み込まれなかった(失敗作の)プラスミドを受け取った大腸菌が混在していて、遺伝子Aが組み込まれた大腸菌を選び出すことができない。
なので、工夫が必要である。たとえば以下のような特殊なプラスミドを使う(様々な便利なプラスミドが販売されている)。
(1)「アンピシリン(大腸菌を殺す抗生物質)の作用を抑える遺伝子」と、「βガラクトシダーゼの遺伝子」をもつプラスミドを用意する。

(2)制限酵素とDNAリガーゼを使って、遺伝子Aを(1)のプラスミドに組み込む(ただし、成功率は100%ではない。遺伝子Aが組込まれなかったプラスミドも混ざってしまう[たとえば、以下の雑談で記すようなセルフライゲーションが起きる])。
組み込む位置はβガラクトシダーゼの遺伝子の中にする(つまり、遺伝子Aが組み込まれた場合、βガラクトシダーゼの遺伝子は分断され、破壊される)。

雑談:プラスミドを切断して、そこにDNA断片(インサート)をDNAリガーゼで繋ぐ操作を行う際、一度切ったプラスミドの末端が再び繋がってしまう(つまり、輪ゴムのようだったDNAを切って紐のようにしたのに、紐の末端同士が再びつながってまた輪ゴムのようになってしまった)ことがある。このような反応を「セルフライゲーション」と呼ぶ。

(3)大腸菌に(2)のプラスミドを導入する。すると、以下の①~③3種類の大腸菌が得られる。
①遺伝子Aが組み込まれなかった(失敗した)プラスミドが導入された大腸菌。
②遺伝子Aが組み込まれた(成功した)プラスミドが導入された大腸菌。
③プラスミドを受け取れなかった大腸菌。
*もともと大腸菌が持っていたDNAは描いていない。

*大腸菌にDNAを効率よく取り込ませる方法にはいくつか種類が知られている(たとえば、急激な温度変化による方法[ヒートショック法]など)。
(4)アンピシリンを含まない培地で①~③を培養すると以下のようなコロニーが観察される。
*コロニー:培地上に形成された、細菌やカビ類、培養細胞などの目に見える塊。たとえば細菌は、適当な環境条件(栄養・温度・pHなど)で速やかに分裂増殖し、一般に18~48時間の培養によりコロニーを形成する。
*アンピシリンを含まない培地では、アンピシリン耐性遺伝子を持っていなくても生育できる。つまり、この培地では①も②も③も生育できる。
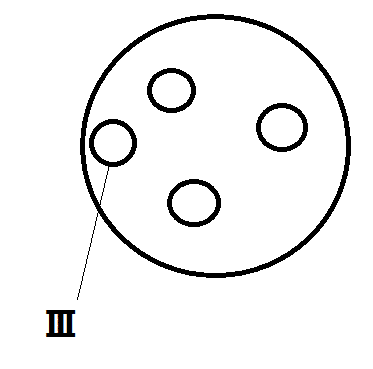
(5)さらに、コロニーの位置関係がそのままになるように(滅菌したビロードなどをつかって、ハンコを押すように移し替えたりする。一つ目の培地全体にビロードをべたっとくっつけて、ビロードをコロニーに触れさせる。そのまま位置関係を変えず、そのビロードを二つ目の培地にくっつける。このようにして別の培地にコロニーを写し取ることができる。このような手法をレプリカ法という)、アンピシリンとX-gal(エックスギャル)を含む培地で①~③を培養する。すると、以下のようなコロニーが見られる。
*アンピシリンは抗生物質であり、アンピシリン耐性遺伝子をもたない大腸菌は、アンピシリンを含む培地では生育できない。
*X-gal・・・βガラクトシダーゼの基質の1つ。市販されている。βガラクトシダーゼによって分解され、青い不溶性の産物ができる(これにより、βガラクトシダーゼを発現しているコロニー[青色]を容易に見分けることができる)。X-galの正式な物質名は5-bromo-4-chloro-3-indolyl-β-D-galactopyranoside。

③のコロニーはⅢである。アンピシリンの入っている培地で生育できなかったからである(プラスミドが入らなかった大腸菌はアンピシリン培地で生育できない)。
①のコロニーはⅠである。アンピシリン培地で生育し(したがってプラスミドを受け取っており)、かつ、βガラクトシダーゼ遺伝子が発現していたからである(遺伝子Aが組み込まれていれば、βガラクトシダーゼ遺伝子は破壊されているはずであり、青い産物は生じないはずである)。
②(我々が求めていた大腸菌)のコロニーはⅡである。アンピシリン培地で生育できるからプラスミドは受け取っている。X-galを分解できていないから(コロニーは白いから)、βガラクトシダーゼ遺伝子が破壊されている=遺伝子Aが組み込まれたプラスミドを受け取っている。
このように、遺伝子Aが導入されたプラスミドを持つ大腸菌(②)だけを簡単に選別できた。
問題:上で説明したの条件で実験を行う。(5)で使用する培地に、アンピシリンを加え忘れてしまった。アンピシリンを加え忘れなかった場合に比べて、コロニーのでき方にはどのような違いが出ると考えられるか?最も適当なものを(あ)~(う)から1つ選べ。
(あ):青いコロニーが増える。
(い):白いコロニーが増える。
(う):変化はない。
答え:(い)
③のコロニーも生育できるようになる。③はβガラクトシダーゼ遺伝子を持っていない(プラスミドを受け取っていない)ので、培地にX-galがあっても、白いコロニーになる。
雑談:上の実験(ブルー・ホワイトセレクション)のような実験で使用されるプラスミドには、βガラクトシダーゼ遺伝子の内部に、様々な種類の制限酵素の認識配列がいっぱい並んだ、「マルチクローニングサイト」という領域が存在する。そのため、我々がどんな制限酵素で遺伝子Aを切り出してきても、プラスミドに組み込みやすいようになっている。
講義動画【ブルー・ホワイトセレクション】
要点:ジデオキシ法は、DNAの塩基配列を決定する方法である。
● ジデオキシ法(サンガー法)…塩基配列が未知のDNAの配列を決定する方法。
① DNA合成の際に、その材料となる通常のヌクレオチドのほかに、特殊なヌクレオチド(ジデオキシヌクレオチド)を加えておく。
*ジデオキシヌクレオチド:ddATP、ddTTP、ddGTP、ddCTP。『3’にOHを持たない』、ジデオキシリボースという糖が糖部分に使われている。
*DNAポリメラーゼは3’末端のOHに次のヌクレオチドをつなぐことを思い出そう。ジデオキシヌクレオチドが取り込まれたら、そこで鎖の伸長が停止する。
② 特殊なヌクレオチド(ジデオキシヌクレオチド)が伸長中のDNA鎖に偶然取り込まれた時点でDNAの合成は止まる。また、特殊なヌクレオチドは、A、T、G、Cの塩基によって、それぞれ異なる蛍光色素を結合させておく。
③ すべての長さを網羅したDNA断片を合成させる(何度も複製を繰り返す。すると、特殊なヌクレオチドが取り込まれるタイミングによって、長さが異なるDNA鎖がたくさん生じる。早々に特殊なヌクレオチドが取り込まれた場合は短いDNA鎖が出現し、複製が最後の方まで進んだ後に取り込まれた場合は長いDNA鎖が出現する。どちらの場合も、標識された特殊なヌクレオチドが合成された鎖の末端部にくっ付いている)。
それらのDNA鎖を電気泳動で鎖の長さで分ける。
④ 合成されたDNA断片の末端に結合した色素の種類を順にたどることにより、鋳型となったもとのDNAの塩基配列を知ることができる。
講義動画【ジデオキシ法(サンガー法)】
要点:植物細胞はプロトプラストにすれば融合させることができる。
● 以下のような手順で、植物細胞同士を融合させることができる。
①ペクチナーゼ(ペクチンを分解する酵素)で細胞間の結合を解く(ペクチン:細胞の接着に働く多糖)。
②セルラーゼ(セルロースを分解する酵素)で細胞壁(主成分はセルロース)を分解し、プロトプラストを得る。
*プロトプラストとは細胞壁を除いた全細胞内容。細胞壁をもたない動物細胞では細胞とプロトプラストの区別はない。
下図はイメージ(原形質連絡などは描いていない)。

③プロトプラストをポリエチレングリコールという化学物質やセンダイウイルスで処理するとプロトプラスト同士が融合する。
例)ジャガイモとトマトを融合=ポマト、オレンジとカラタチを融合=オレタチ
雑談:ポマトは、トマトとジャガイモの両方の収穫が得られるだろうという期待のもと作成された。しかし、結局、できたトマトとジャガイモは非常に小さく、売り物にならなかった。生じる同化産物の量が一定なので、栄養が分散されてしまったと考えられている。
雑談:非常に育てにくいが有用な薬用の植物の細胞と、役には立たないが育てやすい植物の細胞を融合させたことがあったという。期待したのは「育てやすい有用な薬用の植物」である。結果、できたのは、「非常に育てにくい役に立たない雑草」であったという。
雑談:プロトプラストは分子生物学的な研究の役に立つ。植物細胞のセルロースでできた厚い細胞壁は、効率的な遺伝子導入の障壁となる。
雑談:ふつう、異種間の細胞を融合させても、染色体が脱落してしまうのが普通である(融合細胞が成体まで育つのはまれである)。馬の細胞とヒトの細胞を融合させても、ケンタウロスやウマ娘ができる可能性はほぼない。
雑談:ポリエチレングリコールが細胞融合を促進させる原理は完全にはわかっていない。ポリエチレングリコールは生体膜の状態を変えるはたらきを持つと考えられている。
雑談:センダイウイルスは東北大学(だから「センダイ」ウイルスである)のM.Kuroya, N.Ishidaらが報告した、細胞融合作用をもつウイルスである。センダイウイルスが細胞融合を促進させる原理は完全にはわかっていない。
発展:アグロバクテリウム
アグロバクテリウムという細菌は、植物細胞に感染し、感染した細胞をでたらめに増殖させる。結果、クラウンゴールという植物性の腫瘍(コブ:Gall)が生じる。根と茎との境界部(根頭:Crown)にできるのでクラウンゴールという(根頭にコブができるのは、土との摩擦で傷ができやすく、アグロバクテリウムに感染しやすくなるためと考えられている)。
このコブは、その細菌が直接作るのではない。まず、アグロバクテリウムのもつTiプラスミドと言うプラスミドの一部が植物細胞内に入り、そのDNAが核DNAに取り込まれる(TiプラスミドのT-DNA領域という領域のみが切り出され、植物細胞内に送り込まれる。T-DNAは細胞核DNAに組み込まれ、形質転換が起こる。これは、自然界に見られる遺伝子組換えの例の一つである)。
T-DNAにはオーキシン(の働きをするインドール酢酸)、サイトカイニン(の働きをするイソペンテニルアデニン)合成酵素遺伝子があり、それらの遺伝子が発現することで、組織の増殖が起こる。
感染した植物細胞は、必要以上のオーキシンとサイトカイニンを作ってしまう。すると、細胞は、脱分化した細胞を分裂で生み出し、増殖してコブができる(オーキシンとサイトカイニンによって未分化な細胞がつくられることは、後に学ぶ)。
T-DNAの領域を導入したい遺伝子と入れ替え、アグロバクテリウムに戻して植物細胞に感染させれば、植物細胞のDNAに意図した遺伝子を導入することができる。
あまり細かい所は問われない。余裕がある高校生のみ、「クラウンゴールをつくるアグロバクテリウムが遺伝子導入に利用できる」ことを知っておけばよい。下図はイメージ。

雑談:実は、アグロバクテリウムから植物細胞へのDNAの移行の機構は、細菌における接合のプロセスに似ている。まるでアグロバクテリウムは、植物細胞と交配しているかのようである。
<Q.植物細胞への感染は、アグロバクテリウムにとってどんなメリットがあるの?…知らなくてよいが、過剰なオーキシンとサイトカイニンの働きで増殖する植物細胞は、細胞外にオピン(オパインともいう。クラウンゴールの組織で特異的に生産される一群の化合物の総称。アミノ酸とピルビン酸の縮合体などが含まれる)を放出する(T-DNAには、オーキシン合成酵素遺伝子、サイトカイニン合成酵素遺伝子だけでなく、オピン合成酵素遺伝子が存在する)。アグロバクテリウムは、細胞から漏れてくるオピンを栄養源として生きている(植物自身はオピンを利用できない)。つまり、アグロバクテリウムは、植物細胞を、都合の良い物質を作り出す工場に変えてしまうのである。このような現象は、植物に対する「遺伝的植民地化」とも呼ばれる。>
発展:茎頂培養
茎頂分裂組織はウイルスなどに汚染されていないので、分離して培養することで、ウイルスフリー植物の作成に用いることができる。
雑談:インドのGuhaとMaheshwariは、花粉発育の研究の過程で、偶然にも葯内の花粉から半数体植物が発生することを発見した。人為的に半数体植物を作り出し、コルヒチン処理によって染色体を倍加させれば、純系を一気に作成できる。単離した花粉だけを培養して植物体を得ることは難しく、実際には花粉の入った葯のまま培養することが多い。これを葯培養(やくばいよう)という。ただし、葯培養も、一部の種以外には、まだほとんど適用できていない。
発展:キメラマウスを利用したノックアウトマウスの作り方
*2つ以上の異なった遺伝子型の細胞からなる生物をキメラという。
例)全身の細胞の遺伝子A(正常)を遺伝子a(機能を失っている)に変わったマウス(ノックアウトマウス)をつくりたい。
①相同組換えを利用して、ES細胞の遺伝子Aの1つを遺伝子aに変える。
(ES細胞の遺伝子型がAAからAaに変わる。相同組換えについては高校範囲外)
②ES細胞をマウスの複数の初期胚に注入し、複数のキメラマウスを得る。
(ES細胞が将来何に分化するのかについてはランダム。)
③導入したES細胞が生殖細胞に分化したキメラマウス(遺伝子型Aの配偶子の他に、遺伝子型aの配偶子をつくる)を同士を交配し、ノックアウトマウスを産ませる(遺伝子型aの配偶子同士が受精して、遺伝子型aaのノックアウトマウスが生まれる)。
*③について:本資料では『ワトソン遺伝子の分子生物学 第6版』に基づいて解説をしているが、実際は、(ES細胞が生殖細胞に分化した)キメラマウスと野生型のマウスを交配し、遺伝子型A aのヘテロ接合体のマウスを作成することが多い。このヘテロ接合体のマウスを維持していけば、その後ノックアウトマウスを安定して作成できるようになる(ヘテロ接合体[遺伝子型Aa]のマウス同士の交配により、4分の1の確率で遺伝子型aaのマウスを得ることができる)。
講義動画【キメラマウスを利用したノックアウトマウスの作り方】
雑談:キメラは、ギリシャ神話に登場する頭がライオンで体がヤギ、尾がヘビである動物「キメラ」に由来する。
雑談:相同組換えの発生頻度は、非相同組換え(全然関係ないなところに遺伝子が挿入されてしまう)の1000分の1以下と低いが、あらかじめ特別な配列を挿入しておくことで、現在では相同組換え体を効率よく選別する技術が確立されている。 また、クリスパー系を用いて、特定の位置で鎖を切断し、相同組換えを誘導する方法もある。細胞は、破壊されたDNAの修復に、ES細胞に導入された変異遺伝子を選ぶ確率が高い。正常な遺伝子がクリスパー系によって選択的に破壊され、効率よく変異遺伝子に置き換わっていく。
雑談:はじめにES細胞を用いているのは、ES細胞が、もともと初期胚から取ってきて樹立した(つくった)細胞だからである。つまり、また初期胚に戻すことで、マウスの発生に参加できる細胞なのである。
雑談:遺伝子を破壊する方法はいろいろある。たとえば、翻訳領域の始まりの方を少し削除し、フレームシフトを起こさせたり、別な遺伝子(これを薬剤耐性遺伝子にすれば、あとで選別に使える)を途中に割り込ませて標的遺伝子を分断したりすることが多い。
雑談:相同組換えは、 似たDNA塩基配列間の交換反応である(その仕組みについては、完全には解明されていない)。その効率は、相同性の割合や、相同な領域の長さに依存する。高度な配列の相同性が相同組換えを促進することが知られている。
相同組換えは配偶子形成における減数分裂時にも起こる。また、化学物質や放射線によりDNA二重鎖切断が引き起こされた場合にも起こり得える(二本鎖切断は相同組換えによって修復されることがある。組換えに関わる様々なタンパク質が、近くにある相同な無傷のDNAを利用して修復を行う。この修復は、自然条件下では、多くの場合DNAの複製直後、2つの娘DNAが近くにある時に行われる)ことも知られている(相同組換えの詳しい過程は複雑なので、大学で学んでほしい)。
相同組換えを利用して、標的遺伝子を目的の遺伝子に置き換えることができる。下図はイメージ(標的遺伝子と相同な部分をもつDNA[中央部は比較的自由に変えられるが、両端は標的細胞と相同な配列にしておく必要がある]を細胞に導入した場合、相同部分で組換えを起こし、外来DNAがゲノムに取り込まれる。組換えは、DNAを切断するエンドヌクレアーゼや、DNAリガーゼなどの酵素によって進行する)。
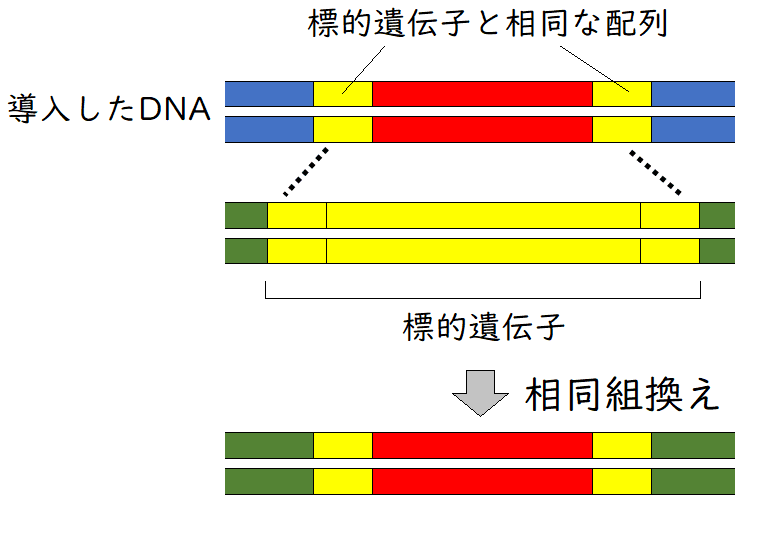
発展:ノックアウトマウス
あるマウスの目的の遺伝子が二個とも完全に不活性化されていたり、失われたりしている場合、そのようなマウスをノックアウトマウスとよぶ。ノックアウトマウスの作成によって、ノックアウトされた遺伝子の働きを知ることができる。
遺伝子の ノックアウトは、非常に重要な分子生物学実験の手法である。たとえば、機能未知の遺伝子をノックアウトした時に、発生過程で羽が無くなったら、その遺伝子は羽を作ることに関係していたのかもしれない、といったふうに研究を進める(教室の黒板消しを教室から取り去ったら、黒板が汚くなっていく。これにより、「黒板消しは黒板を綺麗にする機能をもつ」という事実がわかる。遺伝子をノックアウトすることによる研究はそんなイメージ)。
*当然こんなに話は単純ではない。実際は、ある遺伝子をノックアウトしても、他の遺伝子がその機能をバックアップするなんてこともある。つまり、同じ機能をもつ複数の遺伝子が存在する。また、羽ができなくなったといっても、様々な形質発現のステップの、どこにその遺伝子が関与していたのかわからない。少しずつ研究を進めていくしかない。
問題:複数の異なる遺伝子型の細胞からなる個体を何というか。
答え:キメラ
問題:人の体細胞は基本的にすべて同じゲノムをもつので、人は当然キメラとは言えない。しかし、例外的に、異なるゲノムをもつ体細胞がある。1つ答えよ。
答え:B細胞(遺伝子の再編成により、ゲノムが異なっている)
雑談:遺伝子組換えと遺伝子導入の違い
遺伝子組換えと遺伝子導入は、基本的に、字面のままの意味であるが、同じようなニュアンスで使われることも多い。一応以下に解説したが、高校生は気にしなくてよい。
「遺伝子組換え(gene recombination)」は、一般に、「外来遺伝子を人為的に細胞に導入し、その細胞の遺伝子に組込んだり、一部を置き換えたりすること」を指す。遺伝子組換えを、組換えDNA実験(生物から抽出してきたDNA断片や、人工的に合成したDNAを、試験管内でベクターに結合させ、細胞内に導入して、増幅させる実験)と同義とすることもある。
「遺伝子導入(gene transfer)」は、一般に「遺伝子を細胞に導入する操作」を指す。しかし、遺伝子導入の意味を「遺伝子を細胞に導入し、その遺伝子を発現させたり、その細胞のゲノムに付加させたりする操作」と広くとらえることもあり、遺伝子組換えとの厳密な使い分けは難しい。なお、導入された遺伝子が細胞内のDNAに組込まれず、細胞内で短期間しか存在しないような場合を一過性遺伝子導入と呼び、DNAに組込まれた場合を安定遺伝子導入と呼ぶことがある(一過性遺伝子導入の場合は、狭義の遺伝子組換えとは異なる)。
なお、「遺伝子組換え」、「遺伝子導入」の他にも、「遺伝子操作(gene manipulation)」という語もある。遺伝子操作は「遺伝子組換えの技術を用いて、遺伝子を単離・解析したり、遺伝子に人為的操作を加えたりすること」を指すことが多い(遺伝子操作はよく使われる便利な用語ではあるが、「操作」の意味が広く、あまり定義がはっきりしない。一般的ではないが、広義の遺伝子操作に、細胞融合実験などを含める場合もある)。
要点:遺伝的に同一である個体や細胞(の集合)をクローンという。
● ガードンはカエルのクローンをつくった。
雑談:山中伸弥教授とジョン・ガードン博士は、2012年、ノーベル医学・生理学賞を同時に受賞した。
● ガードンの実験:
① 白化個体(アルビノ)の上皮細胞(分化した細胞)の核を、別の黒色個体(野生型)の未受精卵に移植した(黒色個体の未受精卵の核は、あらかじめ壊しておいた)。
② 結果、核移植を受けた未受精卵の一部は、完全な個体にまで成長した(白化個体が生じたことから、移植された核の遺伝情報が発現し、発生が起こったことがわかる)。
この実験から、上皮細胞(分化した細胞)の核の中にも個体に必要なすべての遺伝子が揃っていることがわかった。
雑談:1997年にはクローンヒツジのドリーが誕生した。技術的には、ヒトのクローンも作製できるだろうと考えられている。ただし、倫理的な問題から、禁止されている。

講義動画【ガードンの核移植実験】
発展:クローンヒツジの作製(ドリーの誕生)
ウィルムットは、クローン羊のドリーを作製した。
①ヒツジの乳腺細胞を低栄養条件で培養した(ふつうは10%のウシ血清を用いる。この時は0.5%のウシ血清が用いられた)。飢餓状態になった細胞は、細胞周期を停止する(細胞はG0期という細胞周期に誘導される[G0期に入った細胞は、最小限の転写のみが起こる静止状態となる]。どうしてG0期に誘導する必要があるのかについては、まだ完全には解明されていない)。
②①の細胞を、あらかじめ核を除去しておいた卵母細胞(減数分裂第二分裂中期で停止している)と融合させることで、核移植を達成した。
③②を胚盤胞期まで発生させたところで、第三のヒツジ(仮母)の子宮に移植した。そして、クローン羊「ドリー」が誕生した(乳腺細胞の供与体と遺伝的に同一な子羊、ドリーが誕生した)。

①ヒツジの乳腺細胞を低栄養条件で培養した。飢餓状態になった細胞は、G0期という細胞周期に誘導される。
②①の細胞を、あらかじめ核を除去しておいた卵母細胞と融合させた。
③②を胚盤胞期まで発生させたところで仮母の子宮に移植した。そして、クローン羊「ドリー」が誕生した。
雑談:ドリーは、乳腺細胞の供与者(ドナー)であるヒツジと遺伝的に同一の核ゲノムを持っている。しかし、ミトコンドリアゲノムが異なる。これはドリーが、除核した未受精卵の細胞のミトコンドリアDNAを受け継いでいるからである。
雑談:上記のヒツジでの成功の後、マウス、ラット、ネコ、イヌ、ウマ、ラバ、ウシなど、様々な哺乳類のクローンが同様の方法(核移植法)によって作製された。
雑談:最初のクローン作製戦略(ウィルムットらによる)では、核移植は細胞融合によって行われたが、現在では、ドナー細胞から核を単離して、単離した核を卵母細胞にマイクロインジェクション(顕微注入)する方法などがとられている。
雑談:2003年2月、ドリーは6歳7か月で死んだ(当時、朝のニュースで報道されていたのを今も覚えている)。ヒツジの寿命は11~12歳なので、やや短命である。ドリーは、老齢のヒツジに見られる肺炎の合併症に苦しみ、安楽死させられた。ドリーの早すぎる死と関節炎の状態から、ドリーの細胞は正常なヒツジの細胞と比べて、健康的ではなかったという意見がある。これは、最初に移植された核の再プログラム化が不完全であったことが原因であるとも言われている。
発展:DNAマイクロアレイ
● スライドグラスのような小さな面上に、多数の少しずつ異なる配列を持つDNAを規則正しく並べ、別のヌクレオチド鎖と反応させ、それらが結合したかどうかのデータを大量に取得する装置あるいは手法をDNAマイクロアレイという。
(実験の例)
①正常な細胞とがん細胞からmRNAを抽出する。
②①のmRNAを逆転写酵素で処理してそれぞれcDNAをつくる。
③正常な細胞由来のcDNAを赤色に、がん細胞由来のcDNAを緑色にそれぞれ標識する。
⓸③をスライドグラス上に並んだDNAと反応させる(スライドグラス上には、様々な遺伝子の配列のDNAが規則正しく設置してある)。
⑤スライドグラス上で
赤色になった個所の配列:正常な細胞のみで発現していた遺伝子の配列
(正常な細胞にのみ存在していたmRNA由来のcDNAがそこの配列に結合して赤色が付いた。)
緑色になった個所の配列:がん細胞のみで発現していた遺伝子の配列
(がん細胞にのみ存在していたmRNA由来のcDNAがそこの配列に結合して緑色が付いた。)
黄色になった個所の配列:両方の細胞で発現していた遺伝子の配列
(正常な細胞に存在していたmRNA由来のcDNAも、がん細胞に存在していたmRNA由来のcDNAもそこの配列に結合して、両方の色が付いた。)
無色の個所の配列:両方の細胞で発現していなかった遺伝子の配列
(そこに設置してあった配列に結合したcDNAはなかった)
雑談:現在では、マウスやヒトの全ゲノム情報をカバーするための特別なマイクロアレイが購入可能である。
講義動画【DNAマイクロアレイ】
以下、上の動画(DNAマイクロアレイ)についての雑談。
・スイライドグラスのようなものに、多数の少しずつ異なるDNAなどの分子を規則正しく並べて、別の分子と反応させ、それらの間の相互作用のデータを取得する手法、あるいはそのための装置をマイクロアレイという。
・ガラス上に2万個以上の部屋(スポット)を用意できるので、たとえばヒト遺伝子のすべて(約2万個)の発現を網羅的に解析することができる。
・動画では2色を使ったが、1色で、色の濃さを解析して発現の大小を比べることもできる。 また、実際は、2種類の発光波長はコンピュータで重ね合わせ、擬似カラーで表示させる。
・あらかじめ、解析に使う配列がわかっていなければこの実験はできない。
・DNAをガラス状に設置する方法はいろいろある。たとえば、そのガラス上でDNAを化学的に合成することもできる。
・加えたcDNAとガラス上のDNAが結合するように、核酸が塩基対を形成することを「ハイブリダイゼーション」という。
・cDNAを使わず、直接mRNAとDNAをハイブリダイゼーションさせることも多い。
発展:電気泳動法を利用して遺伝子型を求める方法・DNA鑑定
(受験勉強の最後に学べばよい。たまに考察問題に出るが、特に覚えておく知識はない。)
● 現代においては、細胞が特定の遺伝子を持っているかどうかを、PCRと電気泳動を用いて推定することができる(かつては、ある個体の遺伝子型を推定するには交配実験を行うしかなかった。しかし現代では細胞が1個でもあれば、その細胞の遺伝子型を推定することができる)。
講義動画【電気泳動法とDNA鑑定】
以下、上の動画に関する雑談。
・マイクロサテライトは、数回から100回を超えて繰り返す場合がある。
・遺伝子をホモに持つ場合、そこから生じるPCR産物を電気泳動で流すと、ヘテロに持つ場合より(DNA断片が多くできるので)バンドが太くなる場合がある。つまり、実は、電気泳動は、DNAの長さだけではなく、DNAの相対的な量も推定できる実験法なのである(そのバンドの明るさをコンピュータ処理することで相対的な量を推定できる)。
・上の動画では、「マイクロサテライトを遺伝子と読んではならない」と言っているが、遺伝子の中に繰り返し配列がある場合もある。
・2塩基から数塩基の繰り返しをマイクロサテライト、数塩基から60塩基の繰り返しをミニサテライト、それ以上の塩基の繰り返しをマクロサテライトと呼ぶこともある。マイクロサテライトの例には動画に出てきたCAのリピートがある。ミニサテライトの例にはテロメア様配列の繰り返しがある。
・ハンチントン病の場合など、遺伝子に近い位置にある繰り返し回数の異常が疾患を引き起こすことが明らかになりつつある。
・本来は、サテライトDNAは、真核生物の染色体を断片化した際に、大部分の配列と塩基組成が異なるために、異なる分画に分別されるDNAを指す(その領域が、単純な塩基の繰り返しであることが分かった)。
雑談
議論しなければいけないこと
● バイオテクノロジーによって遺伝子組換えを行う際は、どのようなことに注意すればよいか。
我々が設計した遺伝子を持った生物が研究室の外にで出て、生態系に影響を及ぼす可能性がある(初めて生化学的手法を用いてDNA組換えを行ったバーグは、遺伝子組み換え実験規則の必要性を強く訴えた)。
たとえば、除草剤耐性遺伝子や害虫抵抗性遺伝子を導入した作物を考える。
適当に実験を行えば、それらの遺伝子が研究室から漏れるかもしれない(他の生物の体に付着したりすることによって)。
周囲に生息する近縁種や非遺伝子組換え作物と交配するとどうなるだろう。時には、畑に生える雑草が除草剤耐性遺伝子をもつようになることもあり得よう(僕の大学でも、多くの遺伝子組換えを行ったアフリカツメガエルが飼育されていた。そのような遺伝子組換えアフリカツメガエルは、厳重に管理しておく必要があった。もし1匹でも水槽から逃げ出せば、研究棟中を大捜索する必要がある)。
