生成AIを用いたAI創薬の実践 Part1 -タンパク質言語モデル基礎編
チャットボットやコードアシスタントを中心に生成AIの活用が進んでいますが、生成AIの活用は他の応用分野にも見ることができます。その中でも我々が特に注目するのは創薬分野です。創薬の分野では、膨大な分子の組み合わせから新しい薬を開発するのに多くの時間と資金がかかるため、AIを活用することで創薬サイクルを短縮する試みが日々行われています。Transformerの登場以降、アミノ酸や化学式を学習したタンパク質言語モデルや生化学モデルを用いた研究が、AmgenやGenentech 、日本においてもアステラス製薬などの製薬企業を筆頭に活発に行われています(2024年7月に、アステラス製薬様がwandbを導入したプレスリリースが出されました "アステラス製薬に、MLOpsプラットフォームWeights & Biasesを導入~AI創薬を支援~")。
日本においては、生成AIを用いた創薬開発の注目が高くなりつつある一方、具体的な実装に対するイメージやグローバル企業が導入するワークフローのキャッチアップに課題を感じる研究者が多いという現状があります。そこで、Weights & BiasesはNVIDIAやマクニカ、Tokyo1(NVIDIAと三井物産が運営する日本最大級の創薬イニシアチブ)などと共同で、「タンパク質言語モデルの事前学習・ファインチューニング」や「生化学モデルを用いた分子構造の最適化」などの座学やハンズオンの提供を2024年の前半から展開しています。2024/7の段階では、累計で9社・約80名の研究者の方にご参加いただき、非常に注目度が高い活動になっています。本ブログではそのエッセンスをまとめています。創薬業界に関係がない方も、生成AIの一つの活用事例として読んでみていただければと思います。
本ブログは複数部構成になっています。Part1では、タンパク質言語モデルの基礎について解説します("O=C(OC)c(c(O)ccc1)c1"のような化学式の配列を学習した生化学モデルもありますが、ここではタンパク質言語モデルを例に解説をします)。Part2では、この分野で注目を集めるフレームワークであるBioNeMoとWandBを用いて、タンパク質言語モデルの事前学習およびファインチューニングの実装の具体例を示します。タンパク質言語モデルや生化学モデルの事前学習や継続事前学習、ファインチューニング、最適化について、2024年7月時点で日本語で書かれた記事は少ないため、実装に関心のある方の役に立つことを願っております(生化学モデルを用いた最適化についても、実践例をもとにworkflowを洗練させた後、Part3としてblogを書こうと思っています)。それでは、Part1: "AI創薬の基礎やタンパク質言語モデルの基礎"をみていきましょう。
AI創薬の必要性
1つの新薬が販売承認を得て上市するまでには、10年以上の非常に長い年月を要します。そのため、医薬品開発は、新薬開発期間と費用の短縮、開発成功確率の向上が重要になりますが、これらの実現に向け、AIが活用されてきました。
近年注目を集めるバイオロジクス(biologics)は、ターゲット分子に"のみ"作用する「特異性と選択性」という特性を高く持ち、副作用が比較的少ないという利点があります。また複数の特異性を持つよう設計することも可能であることから、注目が集まっています。
バイオロジクスの代表例である抗体医薬品を例に、開発をプロセスをみていきましょう。抗体医薬品の候補となる抗体は多岐にわたりますが、実際に実験(wet実験)できる回数には制限があります。そのため、事前に有望な抗体を高精度で予測することは、開発期間の短縮、コスト削減、そして成功確率の向上に直結します。一方で、分子量が大きく、高機能な抗体医薬品(高分子医薬品)の開発は、その多様性から困難です。このような状況下で、AIの活用が創薬プロセスの革新的な解決策として期待されています。AIを活用することで、最適な候補物質を選定や従来の方法では見逃されていた可能性のある有望な候補の発見を通して、開発プロセスを加速させることが期待されています。

タンパク質言語モデル(Protein Language Models: pLMs)とは
創薬におけるAIの活用例の一つとして、タンパク質言語モデルをみていきましょう。タンパク質は生命の分子レベルでの主要な担い手であり、体内でさまざまな重要な機能を果たします。例えば、酵素として化学反応を促進したり、構造タンパク質として細胞の形を維持する役割を持っていたり、信号伝達や免疫反応にも関与しています。これらのタンパク質はアミノ酸という20種類の基本単位が連なってできた配列(例外もあります)から成り、その構造と機能はこのアミノ酸配列によって決定されます。
タンパク質言語モデルは、アミノ酸配列を自然言語でいうところの文字として扱ったモデルです。このモデルを活用することで、アミノ酸配列の特徴を深層学習によって自動的に抽出し、その機能や構造を予測することができます。最近の研究では、Transformerアーキテクチャに基づくモデルが中心となっています。

タンパク質は、その構造がそれが持つ機能と密接な関係にあるため、タンパク質言語モデルが注目を集める以前から、アミノ酸の配列から構造を推定する研究が活発に行われてきました。タンパク質立体構造予測では、DeepMindのAlphaFold2などのモデルが非常に有名ですが、タンパク質言語モデルは、その応用例が構造予測だけではなく、ファインチューニングを行うことで結合予測や配列予測、フォールド認識、タンパク質機能予測などさまざまな用途に使うことができる点が特徴的なポイントです。また、AlphaFold2などのタンパク質立体構造予測モデルでは、多重配列アラインメント(Multiple Sequence Alignment: MSA)などと呼ばれる類似のパターンを持った配列情報も入力として用意する必要がありましたが、タンパク質言語モデルでは、そのタンパク質の配列情報のみから構造を予測することができるという特徴も持っています。構造がすでに知られている配列が多い領域では、まだAlphaFold2などのタンパク質立体構造予測モデルの方が予測精度が高いことが知られていますが、知見が少ない配列においては、タンパク質言語モデルを用いた予測の方が精度が高いケースがあることが報告されています。
代表的なタンパク質言語モデルとして、UniRep、ProtTrans、ProtT5、ESM1、ESM2などがあります。それぞれの特徴は以下の表を参照してください。

タンパク質言語モデルを自社で活用するために
例えば上記のタンパク質言語モデルの中でもESM2は、ライセンスに従えば誰でも利用することができますが、公開されているモデルはすべての領域の配列データを用いて学習されているわけではないため、製薬企業個社が強みを持つ(ないしは開発を進めたい)領域において、そのまま利用するだけでは十分な精度が出ないことが指摘されています。実際、Amgenのデジタルバイオ医薬品創薬ディレクター Christopher Langmeadは、生成AIを活用してバイオ医薬品の創薬と開発を強化(NVIDIAの事例)の中で以下の述べています。
“公開されているモデルは限られていたため、当社独自のデータでカスタムモデルを事前トレーニングする必要がありました。...”
このようにタンパク質言語モデルを実ユースケースで活用していくためには自社のデータをモデルに追加する必要がありますが、その方法を見ていきましょう。以下の図は、タンパク質言語モデルに自社のデータを取り入れ、ダウンストリームタスクに適応する流れを示しています。事前学習・継続事前学習・ファインチューニングの流れについては、大規模自然言語モデルと同様です。

事前学習・継続事前学習の段階で自社データ(アミノ酸配列データ)を使った学習を行うことで、自社データを取り入れることができます(知識獲得のために事前学習と継続事前学習のどちらから進めるについてはまだ決定的な答えがない状況ですが、AmgenやAstellasの例ではモデルのアーキテクチャを使いつつゼロから事前学習から実施する方法をとっています)。事前学習・継続事前学習を行なった後は、ダウンストリームタスクに対応するためにファインチューニングを行います。例えば、あるターゲットタンパク質にペプチド結合するアミノ酸バインダーを得たい場合は、上図右のようにターゲットタンパク質とそれに結合するアミノ酸バインダーをつなげた配列を学習させることで、所望のアミノ酸配列を出力するモデルを得ることができます。
補足:
ペプチド結合とは、アミノ酸同士が連結してペプチドやタンパク質を形成する際に生じる化学結合のことです。ペプチド結合は、タンパク質の主鎖を形成する基本的な結合であり、その構造と安定性がタンパク質の形状や機能に大きく影響を与えます。
実例: 公開データを用いた抗体に特化したタンパク質言語モデルの構築
自社のデータが大量にないケースも多く見られますが、公開データを用いて独自にモデルを学習し、社内のデータを検証や予測に使用した興味深い成功例がありますので、その例を紹介します。
以下は、2024年のGTCでアステラス製薬のNateさんが発表された内容"BioNeMo on DGX: A Robust Platform for Developing and Implementing Reproducible Generative AI in Drug Discovery - Insights from Astellas"に基づいています(紹介にあたり、図などの掲載は本人に許可をいただいております)。
タンパク質言語モデルを使って一般的なタンパク質の特性( 熱安定性など)を予測する場合は、良好な精度になりますが、タンパク質言語モデルを抗体固有の特性の予測に利用すると、精度が低下することが知られています。

そこで、アステラス製薬では、抗体固有のタンパク質言語モデルの構築を行いました。大規模な言語モデルのトレーニングに使用できるほどのデータがなかったため、公開データであるOAS(Observed Antibody Space)データセットが利用されました。OASデータセットは抗体配列のデータベースで、80の異なる研究から収集された、10億以上の抗体配列を含んでいます。アステラス製薬では、OASデータを独自にフィルタリングをし、トレーニングデータセットを用意しています。
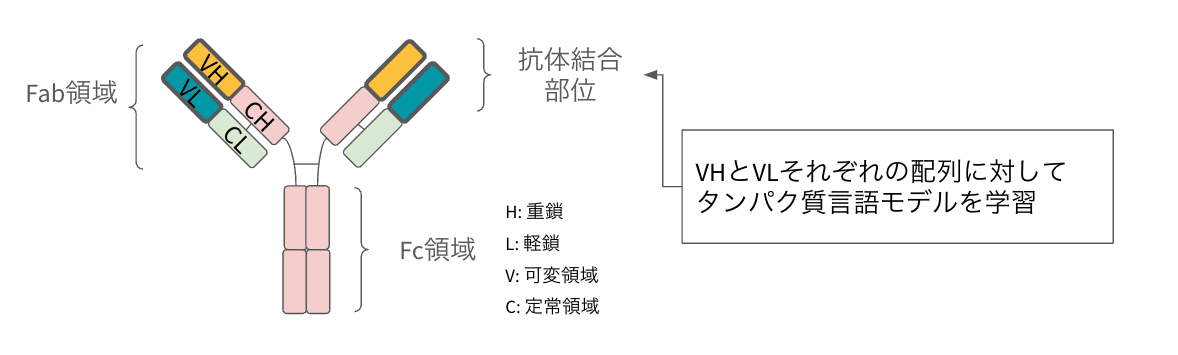
モデルのアーキテクチャにはESM-1nvを利用し、抗体の中のVHとVLに対してそれぞれ個別のトレーニングを実施し、独自モデル"astABpLM"を構築しました。なお、この研究ではVHとVLそれぞれ個別に学習をさせたところに新規性があります。分離をして学習をした方が生物学的に意味のある配列の特徴を獲得することができるのではないかとう仮説のもと、このような興味深い方法が取られています。
詳細: VLには0.45億の配列、VHでは1.21億の配列データを使用し、学習は1node 8 A100 GPUsが使用されました。
以下の結果は、アステラス製薬社内の予測タスクにおいて、独自に開発をしたastABpLMがESM-2より高い精度を実現したことを示しています。

独自の学習には公開データを使いながら、検証や予測には社内のデータを活用した事例ですが、タンパク質言語モデルを創意工夫しながら、社内のユースケースに当てはめる興味深い事例であると考えます。
最後に
この記事では、創薬ドメインに特化した生成AIの活用例について解説をしました。生成AIのドメイン特化したユースケースの開発を引き続きWeights & Biases はご支援していきます。この記事が製薬企業の方以外にも、インサイトがある記事になっていれば幸いです。
Part2では、Weights & Biases を用いた具体的な実装方法について解説をしていきます。
この記事の内容に関し、ご質問やフィードバックがありましたらcontact-jp@wandb.comまでご連絡ください。
この記事が気に入ったらサポートをしてみませんか?