
書記のBio/Chem-Info日誌#16 UCSF Chimeraでリガンド結合部位を観察する
UCSF Chimeraを用いることで,立体構造を可視化することができる。
準備
以下よりダウンロード:
今回は,β2受容体である「3SN6」の構造を用いる。PDBファイルをダウンロードしておく。

UCSF Chimeraで開いたものがこちら。

操作には,3ボタンマウスを用いると便利である。以下に操作方法をまとめた,まずはこれらに慣れておきたい。
分子の回転:左クリック + ドラッグ
分子の並進:ホイールボタン + ドラッグ
分子の拡大縮小:右クリック + ドラッグ or スクロールホイール
原子の選択:Ctrlキー + 左クリック
選択の追加:Ctrlキー + Shiftキー + 左クリック
選択範囲の拡大縮小:(拡大)上矢印キー、(縮小)下矢印キー
選択解除:何も無いところでCtrlキー + 左クリック or メニュー「Select」→「Clear Selection」
向きを変えたものがこちら。

Presetsには様々なテンプレートがあり,中でもInteractive1を用いると色分けができる。

リガンド構造の表示
Select→Residue→(リガンド名)をクリックし,リガンドを選択する。この時,Action→Focusをクリックすると,以下のようにリガンドが中心に表示される。

今回は,ドッキングのために周辺の残基(4Å以内)をわかりやすく表示することにする。
周辺の残基を指定するには,Select→Residue→(リガンド名)でリガンドを選択し,Select→Zoneで以下の画面になる。赤枠部のように変更しOKを押すと,周辺残基が選択されるようになる。

選択したら,Action→Atoms/Bonds→ball & stickで,周辺残基がわかりやすくなった。
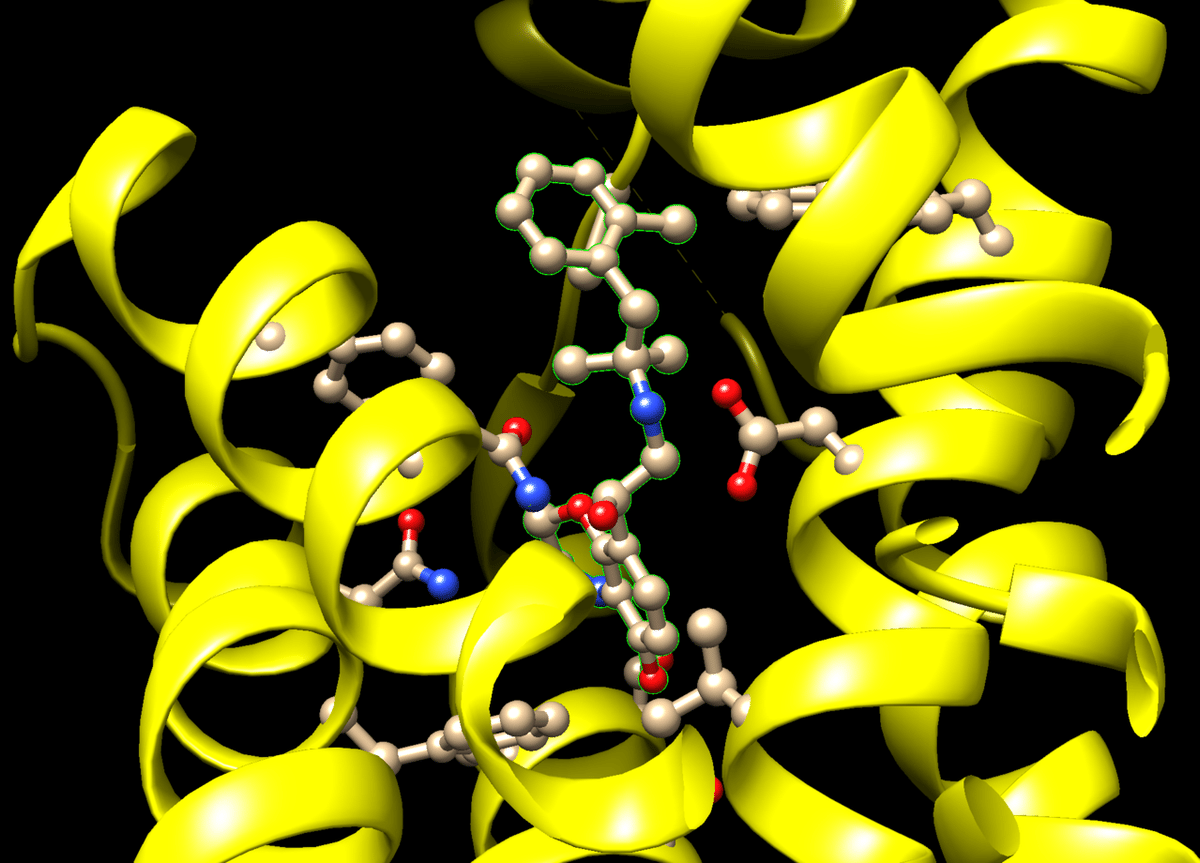
リガンドの表示は,Action→Atoms/Bonds→stickでスティックに戻し,Action→Color→by elementで色分けしておく。
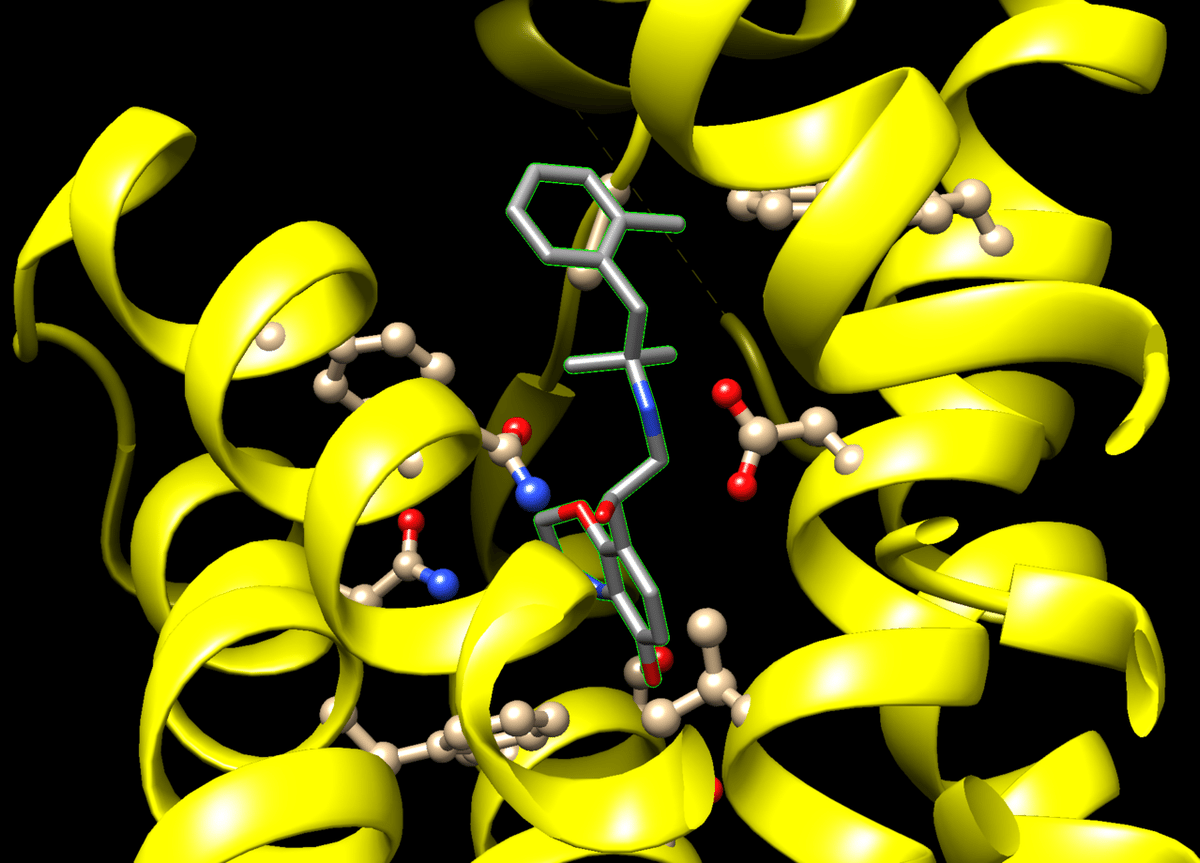
次に残基情報を表示する。Select→Zoneで周辺残基を選択し,Action→Label→residue→name+specifierで表示する。

ここで,Action→Label→optionから文字のフォントを変えることができる。
次に,水素結合を表示する。
リガンドを選択し,Tools→Structure Analysis→FindHBondで以下の画面が表示される。赤枠部を変更しクリックする。

Action→Libbon→hideでリボン構造を隠したものがこちら。これでリガンド周囲の構造がわかりやすくなった。
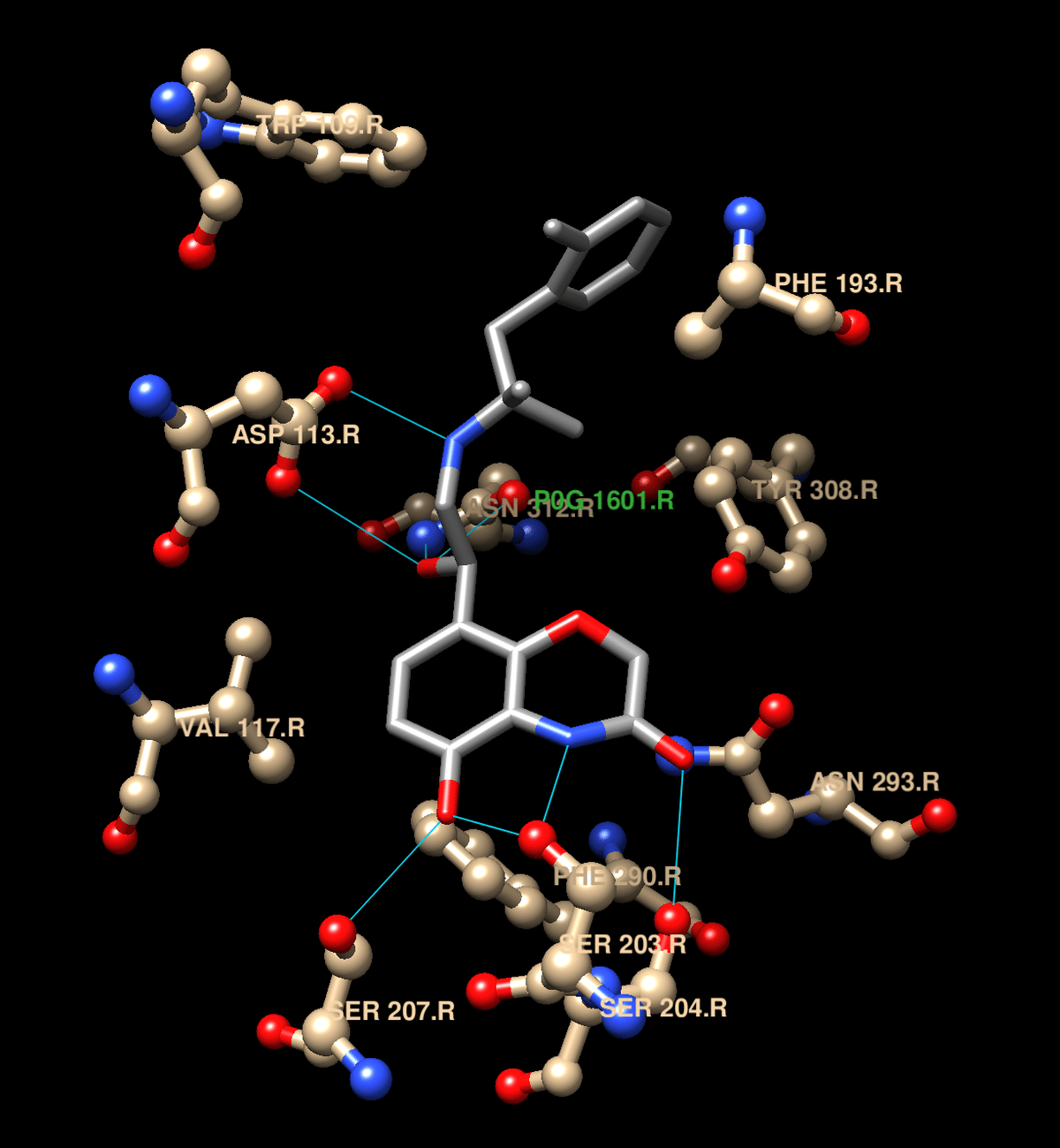
完成したら,File→Save Session As ...でファイルを保存しておく。
本記事のもくじはこちら:
学習に必要な本を買います。一覧→ https://www.amazon.co.jp/hz/wishlist/ls/1XI8RCAQIKR94?ref_=wl_share
