Data Science with Julia && Python (2) - NumpyもJulia環境下で使う
Juliaを使いはじめて良いなと思った所はPythonのライブラリが使えることです。当然ながら全く同じ事ができるわけではないですが、Data Scienceでよく使われるものがJulia上でもちょっと知ればできるのは大きいです。
今回はNumpy「も」Julia環境で使いながらJuliaで同様の結果を得る方法を検討してみます。
Julia上でPythonのライブラリを使うには前回書いたようにPyCallというパッケージが必要です。PyCallをインストールしていなければ
import Pkg; Pkg.add("PyCall")とREPL上で入力してインストールします。早速PyCallを使いましょう
using PyCallではnumpyをインポートします。
julia> np = pyimport("numpy")
私の環境では新しくPython3.6環境をcondaで作成し(py36env)、そこのPATHを通しています。これでnumpyが使えるようになりました。3x3のArrayを作ってみましょう。
julia> arr_2d = np.array([[5,10,15],[20,25,30],[35,40,45]])
ここで30を選択しようとする場合Pythonならarr_2d[1][2]でしょうか。これはJulia環境下ではエラーとなります。Juliaは0-based indexではなく1-based indexですから30を選ぶ場合は
arr_2d[2,3] # 2nd row, 3rd columnとなります。もし[[10,15], [25,30]]を得ようとするなら、PythonとJuliaでは
python> arr_2d[:2, 1:]
julia> arr_2d[1:2, 2:end]です。Pythonでは最初の2は含みませんが、Juliaでは両方がinclusiveです。個人的にはJuliaの方がいろいろと直感的ですが、Juliaの場合Pythonの1:のように省略するとエラーがでますので、2:end(もしくは2:3)とする必要があります。
1から10の配列を作成する場合は以下のどちらでも構いません。
arr = np.arange(1,11)
arr = Array(1:10)繰り返しますがJuliaは1-based indexなので1:10です。JuliaのArrayのほうがシンプルなのでJuliaで同様の事ができる場合はJuliaの方式を試していくのがこのシリーズです。
例えばelementが5以上を選択する場合は
python> arr[arr > 5]
julia> arr[arr .> 5]>の前にあるドットはelementごとに計算することを明示しています。このドットを忘れるとエラーがでます。
python> a = np.arange(50).reshape(5,10)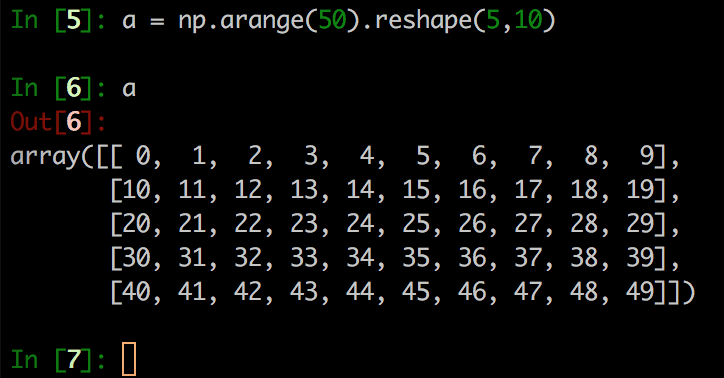
Pythonだと5x10のArrayができます。ですがJulia環境下では.reshapeのようにMethodを書くようにするとエラーがでます。np.arange(50)はできるのですが、reshapeする場合は
julia> reshape(np.arange(50), (5,10))とする必要があります。頭の中では以下のような結果を想像されたかもしれませんが・・・

実際に表示されるのは以下のようにコラムベースです。

Python(やC)はrowベースで表示するのですが、Julia(やFortrun, Matlab, R)はcolumnベースです。Columnベースの方がPerformanceが良いようでこうなっているのが分かりますが、慣れるまでは大変です。あえてPythonと同じ結果を出そうと思うならpermutedimsを使いますが、後述するLinearAlgebraというパッケージを使えばadjointという機能が使えます。
reshape(a,10,5)'最後のシングルクォーテーションは間違いではなくadjointを使う印です。

numpyを利用したりJuliaを使って済むものなどは基本的に多いです
python> np.zeros(10)
julia> zeros(10)python> np.ones(10)
julia> ones(10)python> np.ones(10) * 5
python> np.zeros(10) + 5
julia> ones(10) * 5
julia> zeros(10) .+ 5
Juliaでする場合、要素ごとに計算する際にはドットが必ず必要なので慣れるまではnumpyを使うのもいいかもしれません。Python>と記しましたが上記すべてJulia上で同じ結果を出します。以下もそうです。
np.arange(10,51)
Array{Int}(10:50)np.arange(10,51,2)
Array{Int}(10:2:50)
3x3 Matrixを0から8の数字を用いて作るなら
reshape(np.arange(9),(3,3))
reshape(0:8,3,3)しばらくは.reshapeのようにオブジェクトのあとにMethodをつけない形に煩わさしさを感じるかもしれませんが、慣れればシンプルです。
3x3 Identity Matrixを作る場合はnumpyはシンプルでいいですね
np.eye(3)Juliaだけでする場合はLinearAlgebraのパッケージが必要になります
using LinearAlgebra
Matrix{Float64}(I,3,3)0から1の範囲でランダムな数字を出す際はJuliaの方がシンプルです
np.random.rand(1)
rand(1)np.random.randn(25)
randn(25)0.01から1まで均等に100等分された数字で10x10 Matrix作成する場合
reshape(np.linspace(0.01,1,100),(10,10))
reshape(0.01:0.01:1,(10,10))0から1までに20等分された数字を出す場合
np.linspace(0,1,20)
LinRange(0,1,20)どっちを使ってもいいですね
Juliaでも同様の結果を簡単にだせるのですが、numpyに慣れている場合はnumpyを使えばいいと思います。.reshape()や.shapeのようなMethodの形が使えないという点を気をつけて利用していきましょう。

1から25で5x5の配列を作成します。上記から[2, 7, 12]、もしくは一番下の行を取り出したい場合は

数字に関してはいろいろ書けると思いますが、直感的であるのはありがたいです。合計数を出す場合は

それぞれに列の合計を出す場合は

Pythonでは.sumそしてaxis=0が必要ですが、JuliaではsumはFunctionとして使い、axisの代わりにdimsです。このStandard deviationを出す場合

これを仮にPython環境下ですると、np.std(mat_j)ですが結果は7.211となります。これはNumpy.stdの標準が正規分布での最尤推定量をだすのに対し、JuliaではUnbiased estimateだからです。同様の結果はnp.std(mat_j, ddof=1)とすればでます。
np.std(mat_j) == std(mat_j, corrected=false) # unbiased estimator
OR
np.std(mat_j, ddof=1) == std(mat_j) # biased estimator次回はData Scienceに必須のPandasです。
この記事が気に入ったらサポートをしてみませんか?
