
PRIDE にプロテオミクスデータをアップする
この記事では、PRoteomics IDEntifications Database (PRIDE) にプロテオミクスデータをアップする方法をまとめました。
備忘録的な側面が強めですが、ご参考になれば幸いです。
PRIDE とは
そもそも PRoteomics IDEntifications Database (PRIDE) とは、EMBL's European Bioinformatics Institute(EMBL-EBI)が提供する、質量分析(MS)ベースのプロテオミクスデータのデータリポジトリの 1 つです。
PRIDE を使用しようと考えている場合には、まずアカウントを作成しましょう。
用意するファイル
アップロードに入る前に、ファイルが揃っているかどうかを確認します。ここでは、Complete Submission の場合について、述べていきます。
以下は、上記のリンクから抜き出したものです。詳しくは上記のリンクをご確認ください。
Mandatory
Mass spectrometer output files (called ‘RAW’): The RAW files are the native machine data files - Thermo .RAW, ABSCIEX .wiff, .scan, Agilent .d, Waters .raw, Bruker .yep, Bruker .baf - check the full list here. Each RAW file needs to be related with at least one SEARCH file.
mzTab or mzIdentML result files (called ‘RESULT’): The mzTab and mzIdentML are standard file formats provided by most of the analysis software tools check the full list here. The mzIdentML files contains only identification information, whereas the mzTab files can contains both Identification and Quantification results. These files needs to be related with at least one ‘PEAK’ (peak list) file.
PEAK List files (called ‘PEAK’): If mzTab or mzIdentML are provided the corresponding peak list files must also be provided in order to be able to check the MS/MS evidences that support the peptide/protein identifications (check the full list here).
Recommended
Peptide/protein identification files (called ‘SEARCH’): These are the files output by the software used to perform the data analysis - Mascot .dat, ProteomeDiscover .msf - check the full list here. Each SEARCH file needs to be related with at least one RAW file.
Optional
There are specific files tags for: Images of gels generated in the experiment (‘GEL’), search sequence database files (FASTA), spectral libraries (‘SPECTRUM_LIBRARY’) and any other, relevant file types (‘OTHER’).
提出可能なファイルのより詳しい形式一覧についてはこちら。
※ファイル名には、() やスペースなどの特殊文字が入らないようにしなければいけません。しかし、例えば、MGF (PEAK Files)のファイル名を変えたら、mzid(RESULT files)のファイルの中身も対応するように変えないといけないので、注意が必要です。もし、⑤の段階でエラーが発生し、データの大きさが 0 と出た場合には、ファイルの名前を確認してください。
アップロードの手順
基本的には、PRIDE のウェブページ(下記)にすべて書かれているのですが、個人的にはところどころ落とし穴があるように感じたので、メモをしておきます。
①まずは、上記のサイトから、Submission Tool をダウンロードします。※以下は、2024 年 5 月のもので、version 2.7.3 を使っています。
②次に、ダウンロードしたソフトウェアを開き、PRIDE のアカウントでログインします。
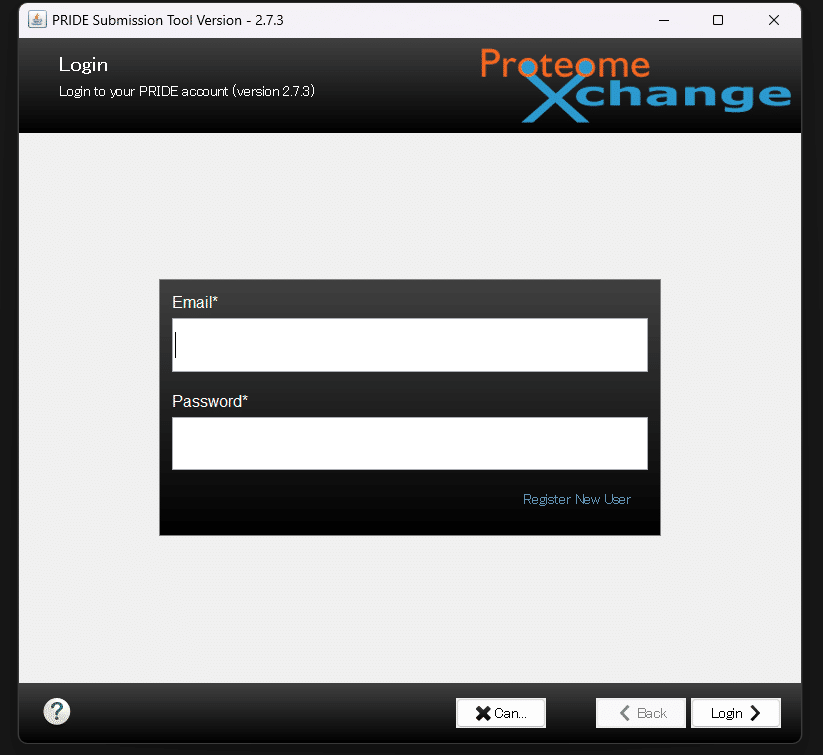
Submission Tool へのログイン
③続いて、Submission の方式を選びます。ここでは Complete Submission を使います。

④指示に従い、データセットの詳細(タイトルやキーワード、プロジェクトの詳細、サンプルの準備のプロセス、データの準備のプロセス、マススぺクトロメトリーの方式)を入力し、次の画面に移ります。
※submit するサンプルのデータ名が、一目では何を表しているかわからない場合、ここの description で annotation しておくことをおすすめします。これ以降のステップで、どのファイルがどのサンプルを示しているのか結びつけられる場所がありません。
⑤続いて準備したファイルを「Add files」から追加します。提供されたファイルの種類に応じて、自動的に各ファイル間の関係とファイルの種類を紐づけられます(例:”RAW” は"RESULT"、"PEAK" は "SEARCH" など)。この時、default で、checksumという txt ファイルが含まれていますが、これはこのままにしておいて、次の画面に移ってください。
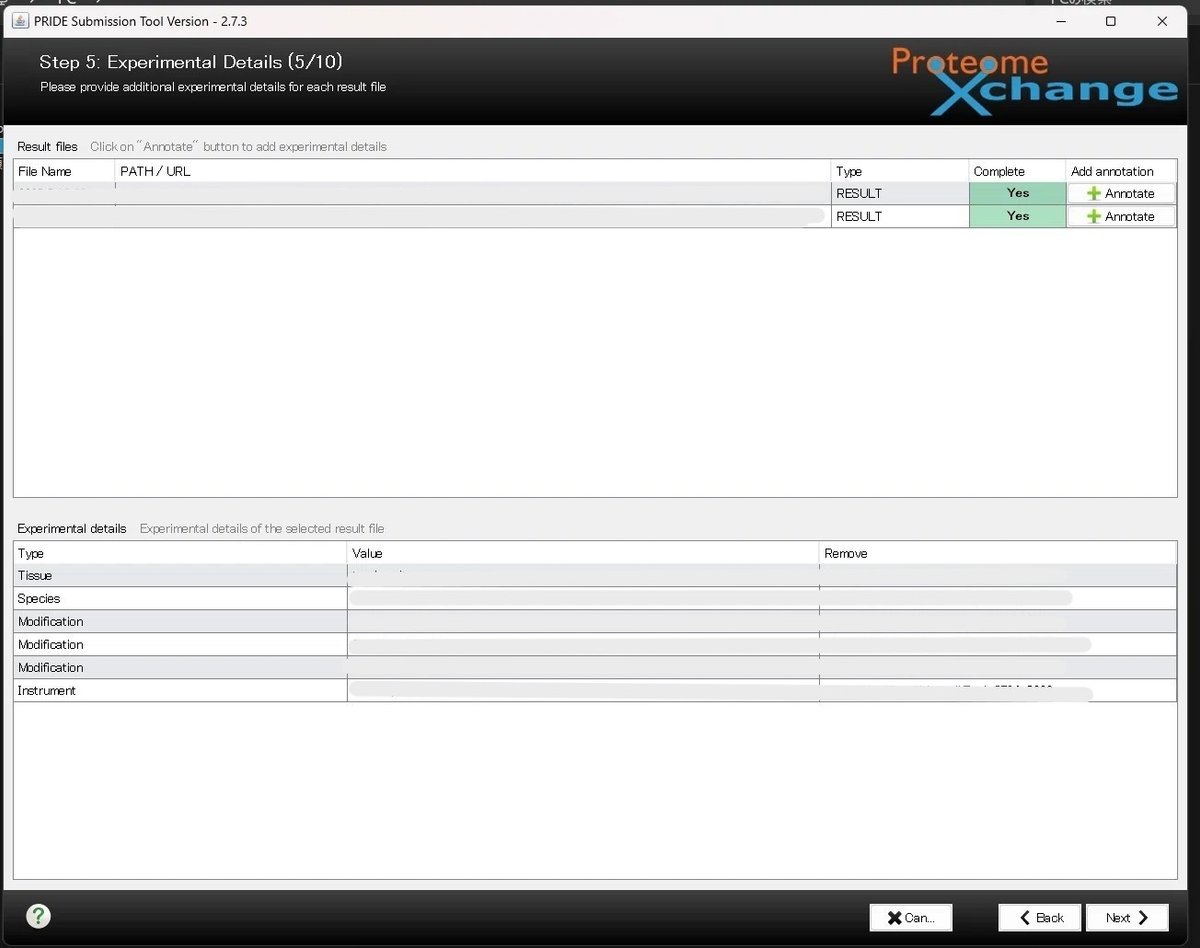
⑥続いて、RESULT として振られたファイルが表示され、その横の Complete の欄が赤で "No" とハイライトされます。その横の、+Annotate を選択し、詳細な情報(生物種、組織、Diseases、MS で detect した modification、instrument(mass spectrometer)、Software 等)を入力します(太字は必須です)。入力すると、Complete の部分が緑の Yes となります。次の画面に移ります。
⑦続いて、checksum の caluculation に移ります。基本的には、caluculation をしてから次に進むことが強く推奨されていますが、ファイルが非常に大きい場合は、チェックサム計算をスキップまたはスルーしても良いそうです。calculation が終わったら手動で次の画面に移ります。
⑧次にラボの head の情報を入力し、次の画面へ。
⑨追加の情報があれば、指示に従って入力し、次の画面へ。
⑨最後に、submission するデータのダブルチェックをします。画面の一番下に、accept の check 欄があるので、ここをチェックし、次の画面へ移行してください。

⑩ここまできたら完了間近です。最後に、フィードバックを行って(顔を選ぶ)完了です。

お疲れ様でした!
何事もなければ、ものの数分で submission が complete し、"Submission Complete" というタイトルのメール(We are happy to inform you that your dataset…)が届きます。このメールには、Project accession、Project DOI、Reviewer 用のアカウントの ID とパスワードがついています。
間違っている部分があればご指摘いただけますと幸いです。
みなさまの研究生活が良いものとなりますように。
この記事が気に入ったらサポートをしてみませんか?
