
Pubmedで論文検索してDeepLで和訳する手間を、全部自動化してラクになりたい人に捧げるNote
医学・生物系分野で研究を行っている研究者は必ずといっていいほどPubmedを利用していると思います。先日、大学病院で研究している方々とお話させてもらったときに、Pubmedの利用方法を聞いていた時に、以下のような不満点があることに気が付きました。
・大量の論文に目を通す必要があるが、Pubmedの画面では何回もクリック、戻る、ページ送りなど、面倒な作業が多い
・掲載されている論文のImpact factorを一つずつ調べる必要があり、手間がかかる
・英語が苦手で、読むのに時間がかかる
この記事を読み終わると以下のことができるようになりPubmedでの論文検索や、論文リストの作成、Impact factorの把握など劇的に効率があがります。
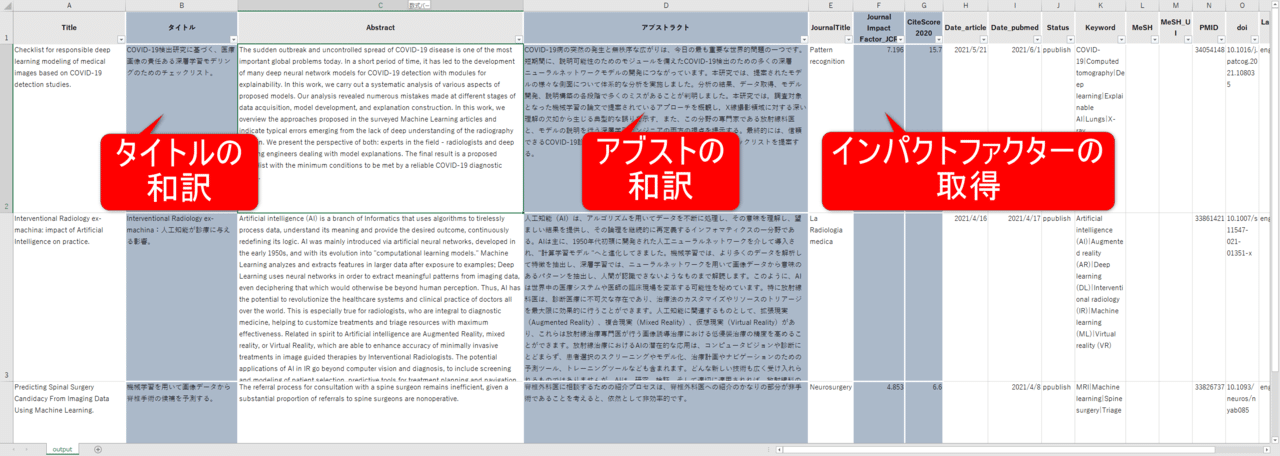
・Pubmedの検索ワードによる検索結果をcsv(エクセルと同等)に出力。
・論文のTitleやAbstractを日本語化。大量の論文に目を通す効率が断然改善。
・それぞれの掲載雑誌のインパクトファクターを取得し、上記のcsvに同時に出力。
csvファイルに出力できるのは非常に重要で、PubmedのHPのように何回もページ送りをしていて、気になる論文がどこで見たかわからなくなることがなくなります。また一つずつインパクトファクターを調べに行く必要がなくなり、インパクトファクターの確認の手間が大幅に削減。またインパクトファクター順にソートしたりすれば、重要な論文から優先的に目を通す、などと行った使い方もできるようにもなります。
本記事ではpythonとGoogle App Scriptを利用したプログラミングを行います。”pythonってなに?プログラミング自分には難しいのでは?”と思う方もいらっしゃるかもしれませんが、あきらめる必要はありません。本記事内に必要なコードはすべて記述していますので、必要なことはコピー&ペーストだけになります。記事に記載している順番通りに進めていけば誰でもできるように工夫しています。もし、プログラミングに興味があるという方は、これを機会に体験してみてはどうでしょうか。
注意
・本記事ではpubmed APIやDeepL APIを利用しているため、その仕様変更によるコードが動かなくなった場合は対応不可能です(2021年6月24日動作確認済)。また各自のPCが原因によるコード不作動にも対応不可です。(ただPythonのコードはWindows、Mac、Linuxいずれも同様に動きますので安心してください。)
・本記事ではプログラミングを行うため、プログラム内を一部分編集する必要がありますが、未経験者でも使えるように非常に丁寧に解説をしているので、そのとおりにすれば問題ありません。またエラーなどはそのままGoogleで検索すればたいてい解決可能です。(それも不可能だと思われる場合は、プログラミングをあまりおすすめしません。ただ、論文検索に費やしている時間のことを考えれば、間違いなく数時間、人によっては数日単位での時間の節約になるのは間違いないと考えています。そのことを考えれば一度トライする価値はあると思います。)
プログラミングは難しいと考えられている方が一定数いると思われますが、本記事内容ではまったくの未経験者でもできるようにワンステップ毎に注意して書いており、むしろプログラミング入門のような内容となってしまっています。プログラミング入門にもいい記事となっていると思います。
前準備
・Anacondaのインストール
今回プログラミングにはpythonを利用します。pythonの環境構築には色々な方法があるのですが、pythonを利用するにはおそらく、Anacondaをインストールするのが一番初心者の方でも間違いがないと思います。もしその他の方法でpythonをインストールできるという方はそちらの方法でやっていただいても問題ありません。以下はAnacondaのインストール方法です。
Anacondaはデータサイエンスおよび機械学習関連アプリケーションのためのPythonおよびRプログラミング言語による開発環境を提供する、オープンソースのディストリビューションです。要は、インストールすることで、pythonを誰でも簡単に利用できる環境を無料で構築することができます。
(Anaconda公式:https://www.anaconda.com/)
個人利用なので、無料のindividual editionをダウンロードします。もし見つからない場合は"Anaconda individual edition”でGoogle検索すれば出てきます。Windowsの場合はWindowsのマークを、Macの場合はリンゴのマーク、Linux系であればペンギンのマークをクリックします。
あとはAnacondaの手順通りにインストールしていくだけですが、注意点は二つあります。
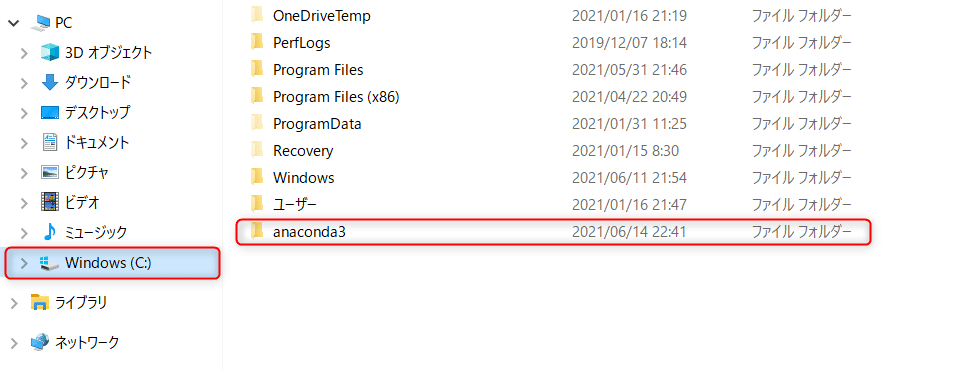
・Anacondaは、日本語などを含むフォルダにはインストールできません。Windowsのアカウント名が日本語になっている場合、デフォルトのインストール先フォルダも日本語になるため、別のフォルダを作成する必要があります。その場合はインストール時、Cドライブ直下にanaconda3というフォルダを作成しておき、インストール先のフォルダをC:¥anaconda3¥としてください。
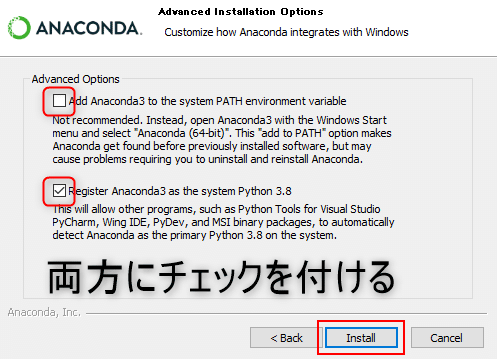
・Advanced Optionsでチェックを付ける。
Add Anaconda 3 to the system PATH environment variable
Register Anaconda3 as the system Python 3.8
の二つにチェックボックスがあるのですが、インターネットで検索すると、いろいろなパターンでインストールされている方がいるようです。私は好みで両方にチェックを付けてインストールしています。
進めていくと、Finishと表示されるのでそれでAnacondaのインストールは完了です。
狙い通りpythonがインストールされているか確認しましょう。
まず、Windowsボタン+Rを同時押しします。
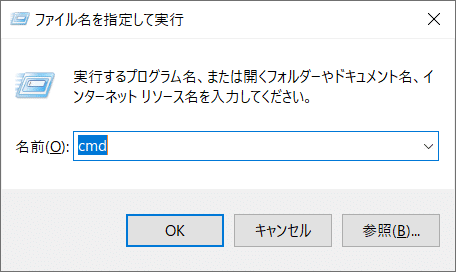
上の画面が表示されるのでcmdと打ってOKをクリックします。コマンドプロンプト(黒い画面)が開きます。
もしくはデスクトップ左下の”ここに入力して検索”欄に"コマンドプロンプト”と入力すれば出てくるのでクリックしてください。黒いウィンドウが開くと思います。
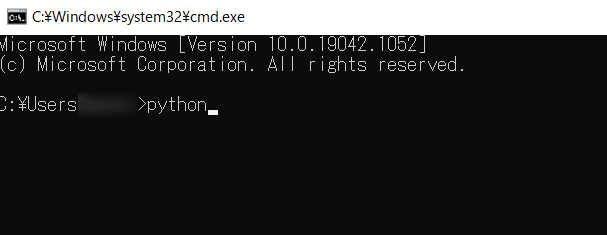
上の黒い画面が表示されるのでpythonと打ち込んでEnterを押しましょう。
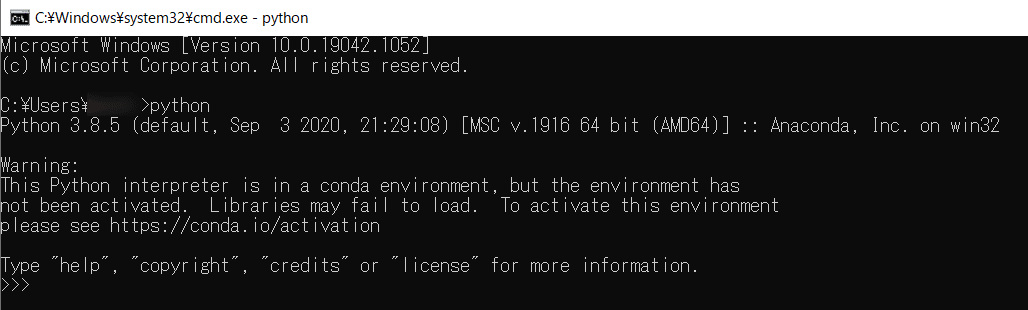
上のように、python 3.8.5のようにバージョンが表示されれば、無事pythonのインストール成功です。ご苦労様でした。ここまで来てしまえば、お使いのPCで好きなようにpythonを使ってプログラミングができるようになります。
Macであれば右上の虫眼鏡をクリック、Spotlight内で”ターミナル”と打ち込めば同様の黒い画面を開くことができます。
pythonでのプログラミングにはpycharmなどのIDEなどをインストールすることで、更に快適な環境を構築することができますが、AnacondaをインストールしておけばJupyter Notebookというデータサイエンスに携わっている人の多くが使っているツールが同時にインストールできています。一つのコードを実行するごとにそのアウトプットを確認することができ、データ分析する際とても便利です。
お疲れさまでした!以上で前準備は完了です。いよいよ、プログラミングしていきましょう!
プログラミングの概要
最小限の機能で実行
ステップ1.Jupyter Notebookを開く
ステップ2.Jupyter Notebookにコードを張り付けて実行
ステップ3.生成されたcsvをgoogle spreadsheetで開く。スクリプトエディタにコードを貼り付けて実行
インパクトファクターやDeepLの和訳を追加
・Journal citation report
・Scopus
・DeepL
進め方としては上記のように、まず最初に最小限の機能で実装します。その後にインパクトファクターや、制度の高いDeepL APIを利用した和訳の実装をしていきたいと思います。
最小限の機能で実行
ステップ1:Jupyter Notebookを開く
まずは作業フォルダを作成します。場所はどこでも大丈夫です。今回はわかりやすいかと思い、Cドライブ直下にpubmedというフォルダ名で作成しました。その作成したフォルダを開きます。
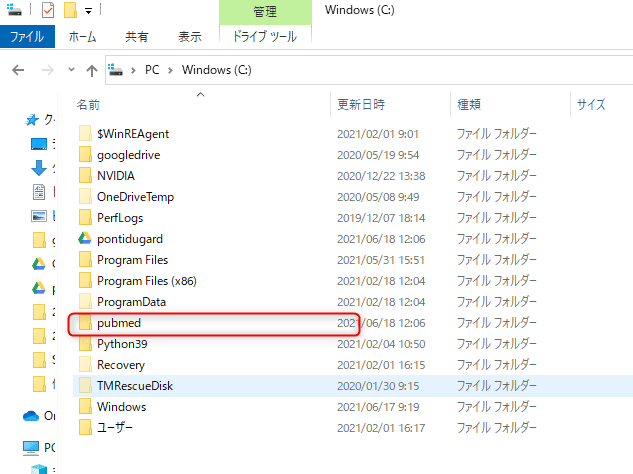
フォルダのパスをコピーします。方法は、フォルダの上のアドレスバーをクリックして、コピーです。
ではコマンドプロンプトを開きます。
黒い画面が表示されれば文字を打つことができるので、pubmedフォルダに移動てください。以下のコードをコピーして貼り付け、Enterを押して実行してください。
(C:\pubmedの部分はそれぞれ各自、先程コピーしたフォルダのパスに置き換えてください。)
cd C:\pubmed以下のようになっていればOKです。

次に以下のコマンドを実行しましょう。少し時間が経つと入力を求められるので”Y”と押して実行しましょう。
pip install numpy pandas matplotlib tqdm完了したら、jupyter notebookを立ち上げましょう。jupyter notebookと打ち、Enterを押してください。
jupyter notebook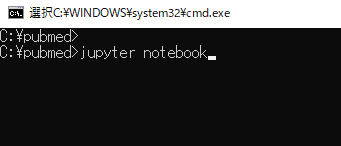
しばらくすると以下の画面が開けばOKです。
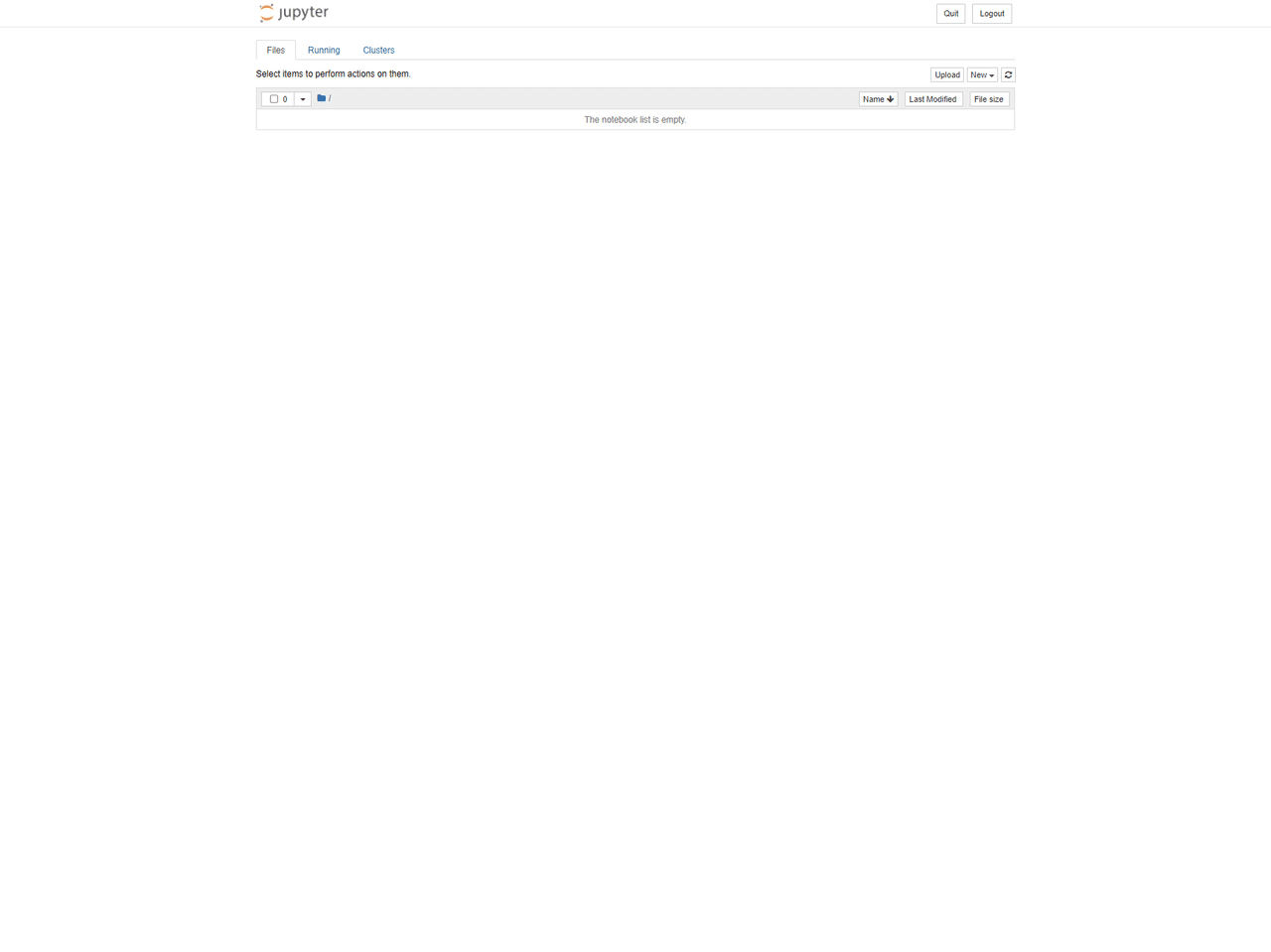
では新しくファイルを作っていきましょう。右上の”New”をクリック、Notebook:の下のPython 3をクリックしてください。
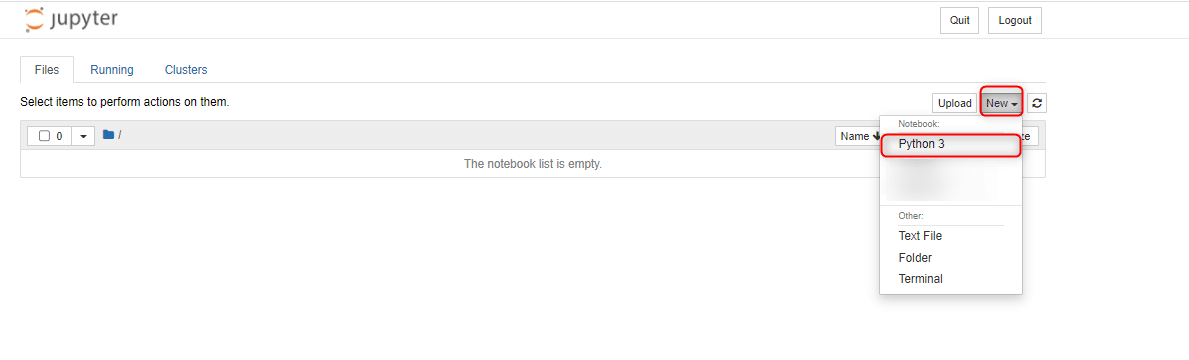
以下の画面になればOKです。
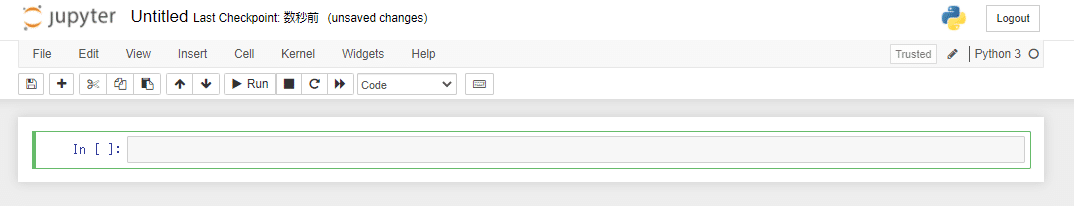
ステップ2.jupyter notebookにコードを張り付けて実行
このボックスの中に以下のコードを貼り付けてください。(Jupyter で出力を確認しながら実行することを前提としたコードなので、冗長的になっていますが、実行する上では問題ありません。)
各自入力が必要な項目は、
TERM:自分がPubmedで検索したいワードを入力してください。Pubmedの検索ボックスに入れるようなワードでOKです。
MIN_DATE、MAX_DATE:それぞれいつからいつまでの日付で論文を取得したいか入力してください。
それ以外はそのままで大丈夫です。
実行はShift+Enterを同時押しです。
import os
import math
import numpy as np
import pandas as pd
import requests
import urllib.parse
import uuid
import xml.etree.ElementTree as ET
from collections import OrderedDict
from tqdm import tqdm_notebook as tqdm
import datetime
###################################################################################################################
# 各自入力が必要な項目
# pubmed search parameters
SOURCE_DB = 'pubmed'
TERM = 'XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX'
DATE_TYPE = 'pdat' # Type of date used to limit a search. The allowed values vary between Entrez databases, but common values are 'mdat' (modification date), 'pdat' (publication date) and 'edat' (Entrez date). Generally an Entrez database will have only two allowed values for datetype.
MIN_DATE = '2021/06/01' # yyyy/mm/dd
MAX_DATE = '2021/12/31' # yyyy/mm/dd
SEP = '|'
BATCH_NUM = 1000
## deepL ##
# API key
deepl_key = "XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX"
# API URL
deepl_path = "https://api-free.deepl.com/v2/translate"
# 有料の場合は
# deepl_path = "https://api.deepl.com/v2/translate"
###################################################################################################################
# pubmed api URL
BASEURL_INFO = 'https://eutils.ncbi.nlm.nih.gov/entrez/eutils/einfo.fcgi'
BASEURL_SRCH = 'https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi'
BASEURL_FTCH = 'https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi'
BASE_PATH = os.getcwd()
def mkquery(base_url, params):
base_url += '?'
for key, value in zip(params.keys(), params.values()):
base_url += '{key}={value}&'.format(key=key, value=value)
url = base_url[0:len(base_url) - 1]
print('request url is: ' + url)
return url
def getXmlFromURL(base_url, params):
response = requests.get(mkquery(base_url, params))
return ET.fromstring(response.text)
def getTextFromNode(root, path, fill='', mode=0, attrib='attribute'):
if (root.find(path) == None):
return fill
else:
if mode == 0:
return root.find(path).text
if mode == 1:
return root.find(path).get(attrib)
# get xml
rootXml = getXmlFromURL(BASEURL_SRCH, {
'db': SOURCE_DB,
'term': TERM,
'usehistory': 'y',
'datetype': DATE_TYPE,
'mindate': MIN_DATE,
'maxdate': MAX_DATE})
# get querykey and webenv
Count = rootXml.find('Count').text
QueryKey = rootXml.find('QueryKey').text
WebEnv = urllib.parse.quote(rootXml.find('WebEnv').text)
print('total Count: ', Count)
print('QueryKey : ', QueryKey)
print('WebEnv : ', WebEnv)
# get all article data
articleDics = []
authorArticleDics = []
authorAffiliationDics = []
def pushData(rootXml):
for article in rootXml.iter('PubmedArticle'):
# get published date
year_p = getTextFromNode(article, 'MedlineCitation/Article/Journal/JournalIssue/PubDate/Year', '')
month_p = getTextFromNode(article, 'MedlineCitation/Article/Journal/JournalIssue/PubDate/Month', '')
date_p = str(year_p) + "/" + month_p
# get article info
year_a = getTextFromNode(article, 'MedlineCitation/Article/ArticleDate/Year', '')
month_a = getTextFromNode(article, 'MedlineCitation/Article/ArticleDate/Month', '')
day_a = getTextFromNode(article, 'MedlineCitation/Article/ArticleDate/Day', '')
try:
date_a = datetime.date(int(year_a), int(month_a), int(day_a)).strftime('%Y/%m/%d')
except:
date_a = ""
year_pm = getTextFromNode(article, 'PubmedData/History/PubMedPubDate[@PubStatus="pubmed"]/Year', ''),
month_pm = getTextFromNode(article, 'PubmedData/History/PubMedPubDate[@PubStatus="pubmed"]/Month', ''),
day_pm = getTextFromNode(article, 'PubmedData/History/PubMedPubDate[@PubStatus="pubmed"]/Day', ''),
try:
date_pm = datetime.date(int(year_pm[0]), int(month_pm[0]), int(day_pm[0])).strftime('%Y/%m/%d')
except:
date_pm = ""
articleDic = {
'Title' : getTextFromNode(article, 'MedlineCitation/Article/ArticleTitle', ''),
'Abstract' : getTextFromNode(article, 'MedlineCitation/Article/Abstract/AbstractText', ''),
'JournalTitle' : getTextFromNode(article, 'MedlineCitation/Article/Journal/Title', ''),
# if you want to get data in flat(denormalized), uncomment below. but it's difficult to use for analytics.
# 'Authors' : SEP.join([author.find('ForeName').text + ' ' + author.find('LastName').text if author.find('CollectiveName') == None else author.find('CollectiveName').text for author in article.findall('MedlineCitation/Article/AuthorList/')]),
# 'AuthorIdentifiers' : SEP.join([getTextFromNode(author, 'Identifier', 'None') for author in article.findall('MedlineCitation/Article/AuthorList/')]),
# 'AuthorIdentifierSources' : SEP.join([getTextFromNode(author, 'Identifier', 'None', 1, 'Source') for author in article.findall('MedlineCitation/Article/AuthorList/')]),
"Date_publish" : date_p,
'Date_article' : date_a,
'Date_pubmed' : date_pm,
'Status' : getTextFromNode(article, './PubmedData/PublicationStatus', ''),
'Keyword' : SEP.join([keyword.text if keyword.text != None else '' for keyword in article.findall('MedlineCitation/KeywordList/')]),
'MeSH' : SEP.join([getTextFromNode(mesh, 'DescriptorName') for mesh in article.findall('MedlineCitation/MeshHeadingList/')]),
'MeSH_UI' : SEP.join([getTextFromNode(mesh, 'DescriptorName', '', 1, 'UI') for mesh in article.findall('MedlineCitation/MeshHeadingList/')]),
'PMID' : getTextFromNode(article, 'MedlineCitation/PMID', ''),
'doi' : getTextFromNode(article, 'MedlineCitation/Article/ELocationID[@EIdType="doi"]', ''),
'Language' : getTextFromNode(article, 'MedlineCitation/Article/Language', ''),
}
articleDics.append(OrderedDict(articleDic))
if article.find('MedlineCitation/MeshHeadingList/MeshHeading/') != None:
tmp = article
# get author info
for author in article.findall('MedlineCitation/Article/AuthorList/'):
# publish author ID
# * It's only random id. not use for identify author. if you want to identify author, you can use identifier.
authorId = str(uuid.uuid4())
# author article
authorArticleDic = {
'authorId' : authorId,
'PMID' : getTextFromNode(article, 'MedlineCitation/PMID', ''),
'name' : getTextFromNode(author, 'ForeName') + ' ' + getTextFromNode(author,'LastName') if author.find('CollectiveName') == None else author.find('CollectiveName').text,
'identifier' : getTextFromNode(author, 'Identifier', '') ,
'identifierSource' : getTextFromNode(author, 'Identifier', '', 1, 'Source')
}
authorArticleDics.append(OrderedDict(authorArticleDic))
# author affiliation(author: affiliation = 1 : n)
if author.find('./AffiliationInfo') != None:
for affiliation in author.findall('./AffiliationInfo'):
authorAffiliationDic = {
'authorId' : authorId,
'affiliation' : getTextFromNode(affiliation, 'Affiliation', '') ,
}
authorAffiliationDics.append(OrderedDict(authorAffiliationDic))
# ceil
iterCount = math.ceil(int(Count) / BATCH_NUM)
# get all data
for i in tqdm(range(iterCount)):
rootXml = getXmlFromURL(BASEURL_FTCH, {
'db': SOURCE_DB,
'query_key': QueryKey,
'WebEnv': WebEnv,
'retstart': i * BATCH_NUM,
'retmax': BATCH_NUM,
'retmode': 'xml'})
pushData(rootXml)
# article
df_article = pd.DataFrame(articleDics)
# df_article.head(10)
# author article
df_author = pd.DataFrame(authorArticleDics)
# df_author.head(10)
# author affiliation
df_affiliation = pd.DataFrame(authorAffiliationDics)
# df_affiliation.head(10)
# save previoud dataframe as previous
today = datetime.date.today()
# save previoud dataframe as previous
try:
os.rename(os.path.join(BASE_PATH,'pubmed_article.csv'),os.path.join(BASE_PATH,'prev_pubmed_article.csv').format(today))
os.rename(os.path.join(BASE_PATH,'pubmed_author.csv'),os.path.join(BASE_PATH,'prev_pubmed_author.csv').format(today))
os.rename(os.path.join(BASE_PATH,'pubmed_affiliation.csv'),os.path.join(BASE_PATH,'prev_pubmed_affiliation.csv').format(today))
except:
pass
# save new dataframe
df_article.to_csv(os.path.join(BASE_PATH,'pubmed_article.csv'), index = False)
df_author.to_csv(os.path.join(BASE_PATH,'pubmed_author.csv'), index = False)
df_affiliation.to_csv(os.path.join(BASE_PATH,'pubmed_affiliation.csv'), index = False)
# get difference between previous and new dataframe
try:
df_article_prev = pd.read_csv(os.path.join(BASE_PATH,'prev_pubmed_article.csv').format(today))
df_diff = df_article[~df_article["PMID"].isin(df_article_prev["PMID"])]
df_diff
except:
pass
## Impact factor
# reload
try:
df_article = pd.read_csv('pubmed_article.csv')
# df_author = pd.read_csv('pubmed_author.csv', index_col=0)
# df_affiliation = pd.read_csv('pubmed_affiliation.csv', index_col=0)
except:
pass
df_article["JournalTitle"].value_counts()
# JCRやScopusデータとmergeするため、論文名を小文字としておく
df_article["JournalTitle_lower"] = df_article["JournalTitle"].str.lower()
#### JCRのデータ処理 ####
# JCRからダウンロードしたファイルをIF_by_JCR.csvという名前に変更しておく
try:
df_JCR_IF = pd.read_csv("IF_by_JCR.csv")
df_JCR_IF = df_JCR_IF.drop_duplicates()
df_JCR_IF["JournalTitle_lower"] = df_JCR_IF["Full Journal Title"].str.lower()
df_JCR_IF = df_JCR_IF.rename(columns={
"Rank": "Rank_JCR",
"TotalCites": "TotalCites_JCR",
"Journal Impact Factor": "Journal Impact Factor_JCR",
"Eigenfactor Score": "Eigenfactor Score_JCR"
})
df_JCR_IF = df_JCR_IF.drop("Full Journal Title", axis =1)
df_article_IF = df_article.merge(df_JCR_IF, on="JournalTitle_lower", how = "left")
df_IF = df_article_IF["Journal Impact Factor_JCR"]
df_article_IF = df_article_IF.drop(["Journal Impact Factor_JCR"], axis =1)
journal_col = df_article_IF_scrps.columns.get_loc("JournalTitle")
df_article_IF.insert(loc = journal_col+1, column = "Journal Impact Factor_JCR", value = df_IF)
df_article_IF
except:
df_article_IF = df_article
#### Scopusのデータ処理 ####
# Scopusからダウンロードしたファイルはxlsbファイルになっているので、
# 一度開き、名前を付けて保存からCiteScore 2011-2020.csvという名前に変更して保存しておく
try:
df_scrps_IF = pd.read_csv("CiteScore 2011-2020.csv")
df_scrps_IF_minimum = df_scrps_IF[["Title", "CiteScore 2020"]]
df_scrps_IF_minimum = df_scrps_IF_minimum.drop_duplicates()
df_scrps_IF_minimum["JournalTitle_lower"] = df_scrps_IF_minimum["Title"].str.lower()
df_scrps_IF_minimum = df_scrps_IF_minimum.drop("Title", axis=1)
df_article_IF_scrps = df_article_IF.merge(df_scrps_IF_minimum, on="JournalTitle_lower", how = "left")
df_IF = df_article_IF_scrps["CiteScore 2020"]
df_article_IF_scrps = df_article_IF_scrps.drop(["CiteScore 2020"], axis =1)
journal_col = df_article_IF_scrps.columns.get_loc("JournalTitle")
df_article_IF_scrps.insert(loc = journal_col+1, column = "CiteScore 2020", value = df_IF)
df_article_IF_scrps
except:
df_article_IF_scrps = df_article_IF
df_article_IF_scrps
def deepl_translate(text):
# URLクエリに仕込むパラメータの辞書を作っておく
params = {
"auth_key": deepl_key,
"text": text,
"source_lang": 'EN', # 入力テキストの言語を日本語に設定(JPではなくJAなので注意)
"target_lang": 'JA' # 出力テキストの言語を英語に設定
}
# パラメータと一緒にPOSTする
request = requests.post(deepl_path, data=params)
result = request.json()
return result["translations"][0]["text"]
# Only when deepl APi is available
try:
title_col = df_article_IF_scrps.columns.get_loc("Title")
df_article_IF_scrps.insert(loc = title_col+1, column = "タイトル_deepl", value = df_article_IF_scrps["Title"].map(deepl_translate))
abstract_col = df_article_IF_scrps.columns.get_loc("Abstract")
df_article_IF_scrps.insert(loc = abstract_col+1, column = "アブストラクト_deepl", value = df_article_IF_scrps["Abstract"].map(deepl_translate))
df_article_IF_scrps
except:
df_article_IF_scrps
df_article_IF_scrps.to_csv("output.csv",index = False, encoding='utf_8_sig')pubmedフォルダ内にoutput.csvが生成されると思いますので、そちらが求めていたものとなります。
ステップ3:生成されたcsvをgoogle spreadsheetで開く。スクリプトエディタにコードを貼り付けて実行
Google翻訳を行います。ここで紹介するGoogle翻訳はspreadsheet内のgoogletranslate関数よりも現状は精度がいい翻訳となるので、利用してみてください。
DeepLの和訳は優秀ですが、無料プランだと1ヶ月50万文字の制限がついています。おそらく、論文にして1000本ぐらいならタイトルとアブストの和訳できるはずです。(もしAPI keyを共有してしまうと自分が使いたいときに制限がかかってしまい、使えなくなるので共有はしないように気をつけてください。)もし制限に引っかかってしまった場合はGoogle翻訳を行います。
まずステップ2で生成されたoutput.csvファイルをgoogleドライブにアップロードし、開いてください。アップロード先はどんな場所でも大丈夫です。
今回はpubmedというフォルダを作り、その中にアップロードしました。アップロードはドラッグ&ドロップで大丈夫です。
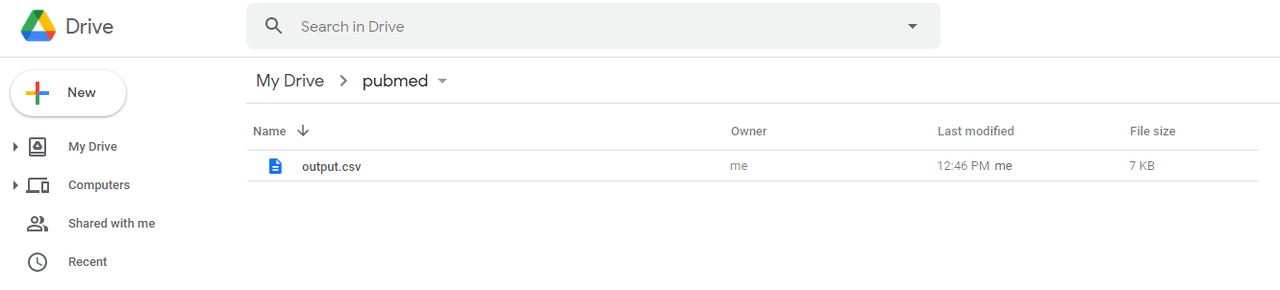
画面の上の”Open with"をクリックして、Google Sheetsをクリックしてください。(日本語環境、英語環境で少し表記が違うかもしれません)
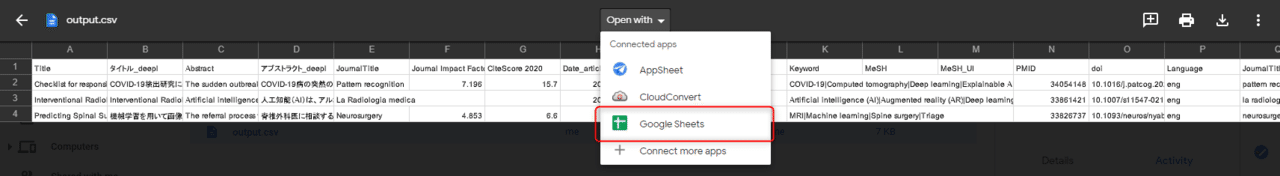
画面上のToolsをクリックし、Script editorをクリックしてください。
以下の画面が開くと思います。
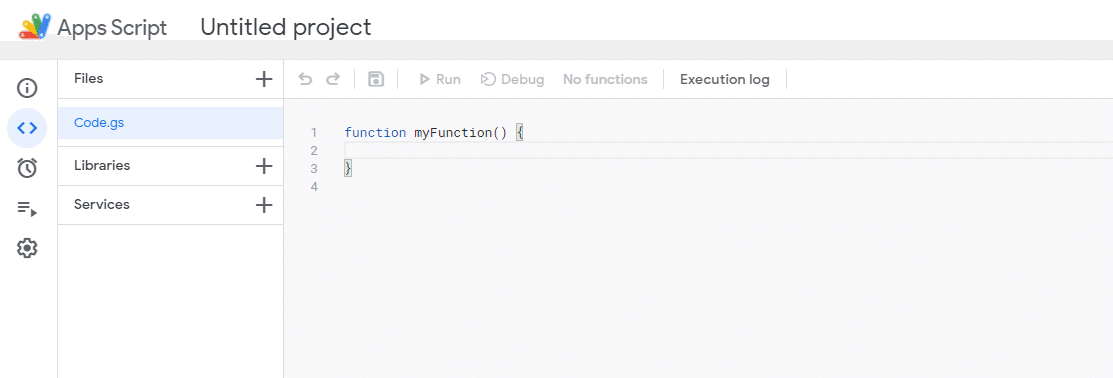
以下のコードを消して、
function myFunction() {
}以下のコードを貼り付けてください。XXXXXXXXXXXの部分はspreadsheetのアドレスのうち、https://docs.google.com/spreadsheets/d/XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX/edit# のXXXXXの部分をコピーして貼り付けてください
function translate() {
var start = new Date()
// https://docs.google.com/spreadsheets/d/XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX/ のXXXXXの部分
var targetSpreadsheet = SpreadsheetApp.openById("XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX")
// sheet名
var targetSheet = targetSpreadsheet.getSheetByName("output")
var lastRow = targetSheet.getLastRow()
var lastColumn = targetSheet.getLastColumn()
var columns = targetSheet.getRange(1,1,1,lastColumn).getValues()[0]
var title_column = columns.indexOf("Title")
var abstract_column = columns.indexOf("Abstract")
for (var i = 1; i <= lastRow; i ++){
Logger.log(i)
var sourceCell = targetSheet.getRange(i, title_column+1, 1, 1).getValue()
var destCell = targetSheet.getRange(i, lastColumn+1, 1, 1)
var translation = LanguageApp.translate(sourceCell,"en","ja")
destCell.setValue(translation)
var sourceCell = targetSheet.getRange(i, abstract_column+1, 1, 1).getValue()
var destCell = targetSheet.getRange(i, lastColumn+2, 1, 1)
var translation = LanguageApp.translate(sourceCell,"en","ja")
destCell.setValue(translation)
}
var end = new Date()
// second
var time_past = (end-start)/1000
Logger.log(time_past)
}貼り付けたら、保存を押してtranslateが実行できる状態にしてください。

最後に"Run"をクリックしてください。するとpermissionを求める画面になります。
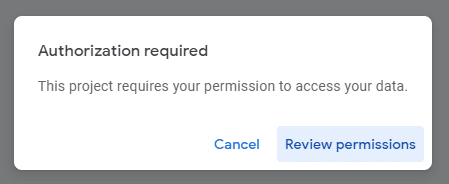
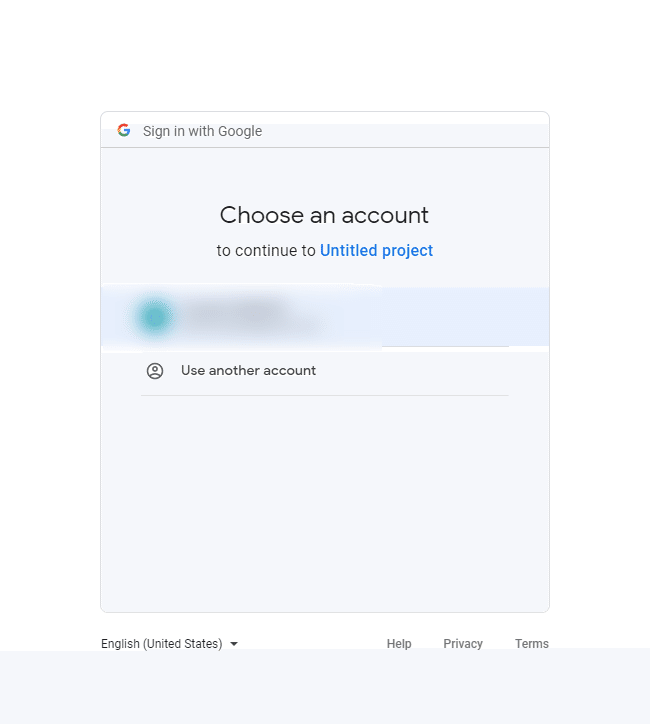
Googleのアカウントを選びます。
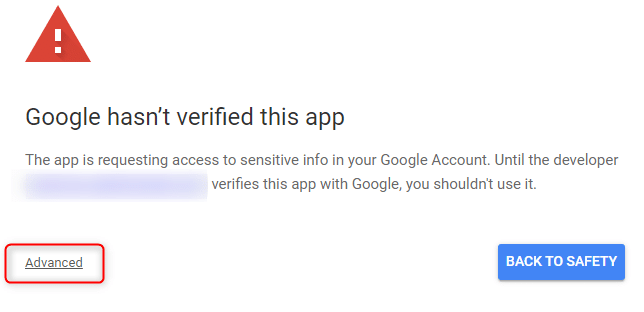
”Advanced"をクリック
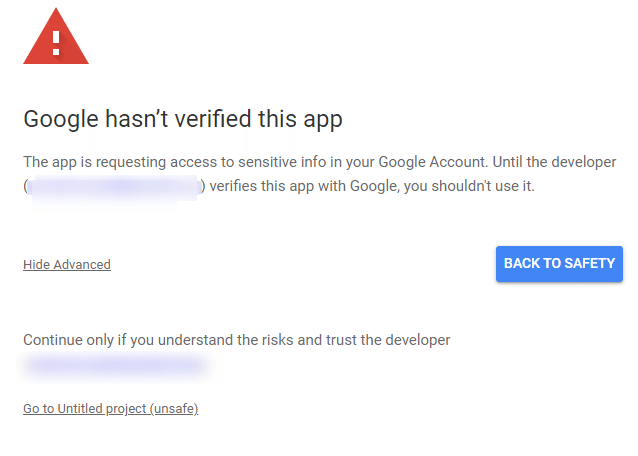
Go to Untitled project(Unsafe)をクリック。
Allowをクリックしてください。
そうするとコードが実行され、Googleの翻訳による和文がセルに現れてきます。
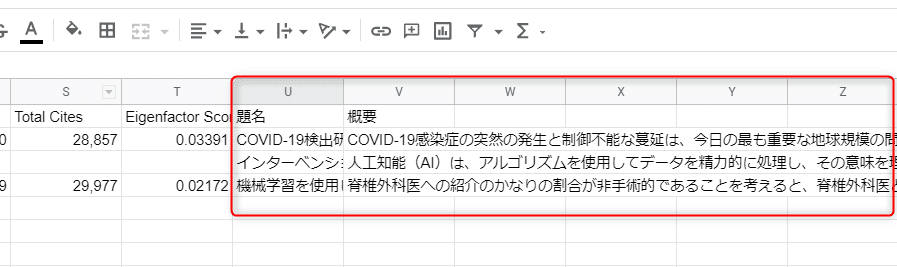
以上が、最低限の機能となります。以降は一つずつ機能を追加する方法について解説しています。完了後、再度同じようにプログラムを実行できるようになっています。
インパクトファクターやDeepLの和訳を追加
・インパクトファクターのデータをダウンロード
インパクトファクターはJournal citation reportとScopusの2つをデータソースとして利用することを想定しています。Journal citation reportは購読している必要がありますが、Scopusはアカウントを作成すれば誰でも取得できます。(もしそれらのデータがない場合、インパクトファクターは出力できませんが、問題なくプログラムは動くようになっています。)それぞれはそれぞれClarivateとElsevierが発表しているデータであり、本記事で配布できないため、各自で準備する必要があります。以下手順です。
Journal citation report
Journal citation reportのHPを開きます。(2021年にレイアウトが変更されているので注意)

右上のProductsをクリックし、そのプルダウンからJournal citation Reports(classic)をクリックします。
"Browse by Journal"をクリックします。
右上のダウンロードボタンをクリックすれば”JournalHomeGrid.csv"というファイルがダウンロードされます。
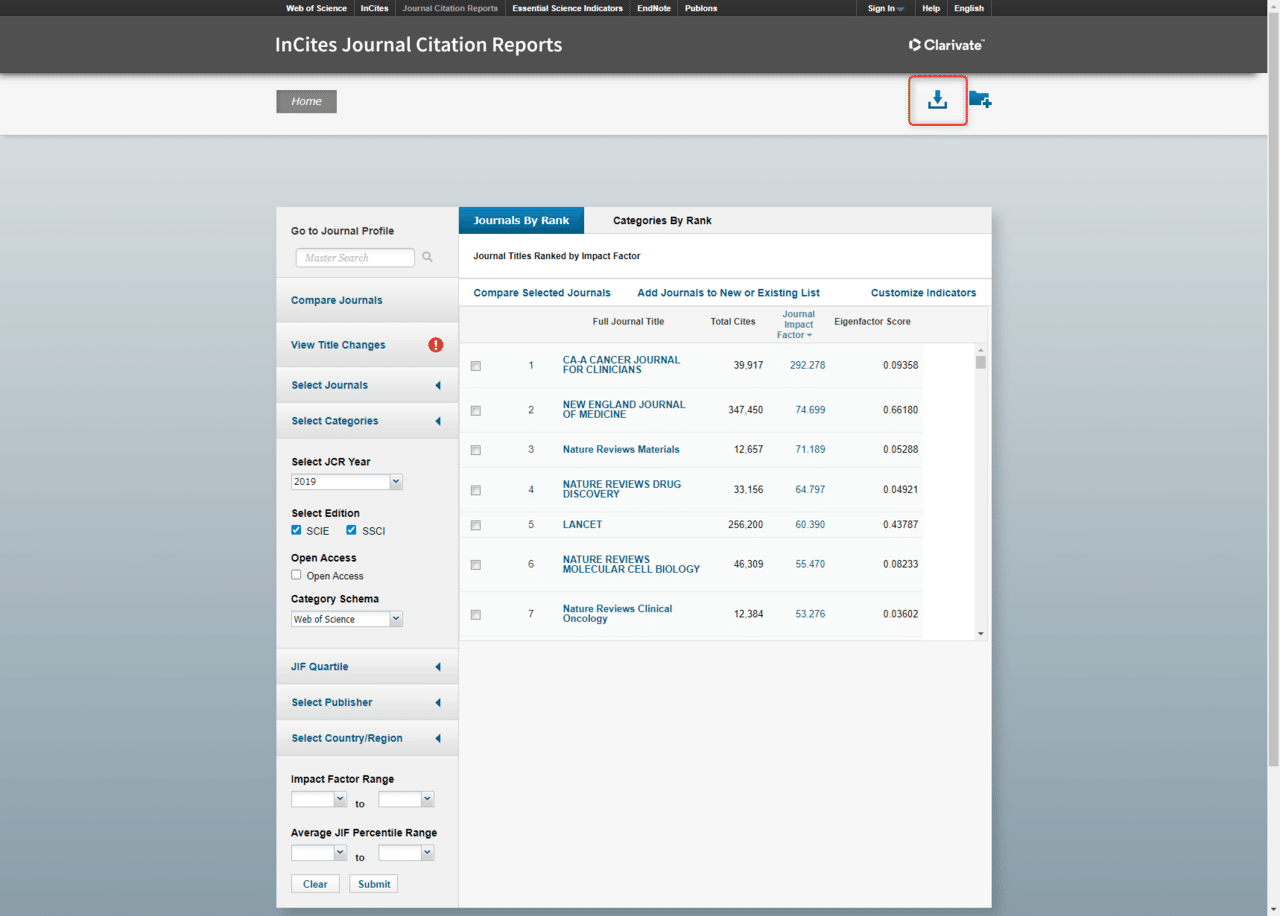
ダウンロードしたcsvファイルに、冒頭と末尾の不必要な行を削除します。
削除をクリックします。

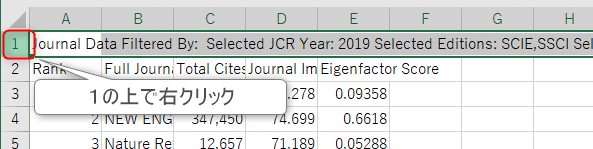
同様に末尾に2行分不必要な行があるので削除します。

削除をクリックします。

冒頭、末尾がそれぞれ以下のようになっていればOKです。

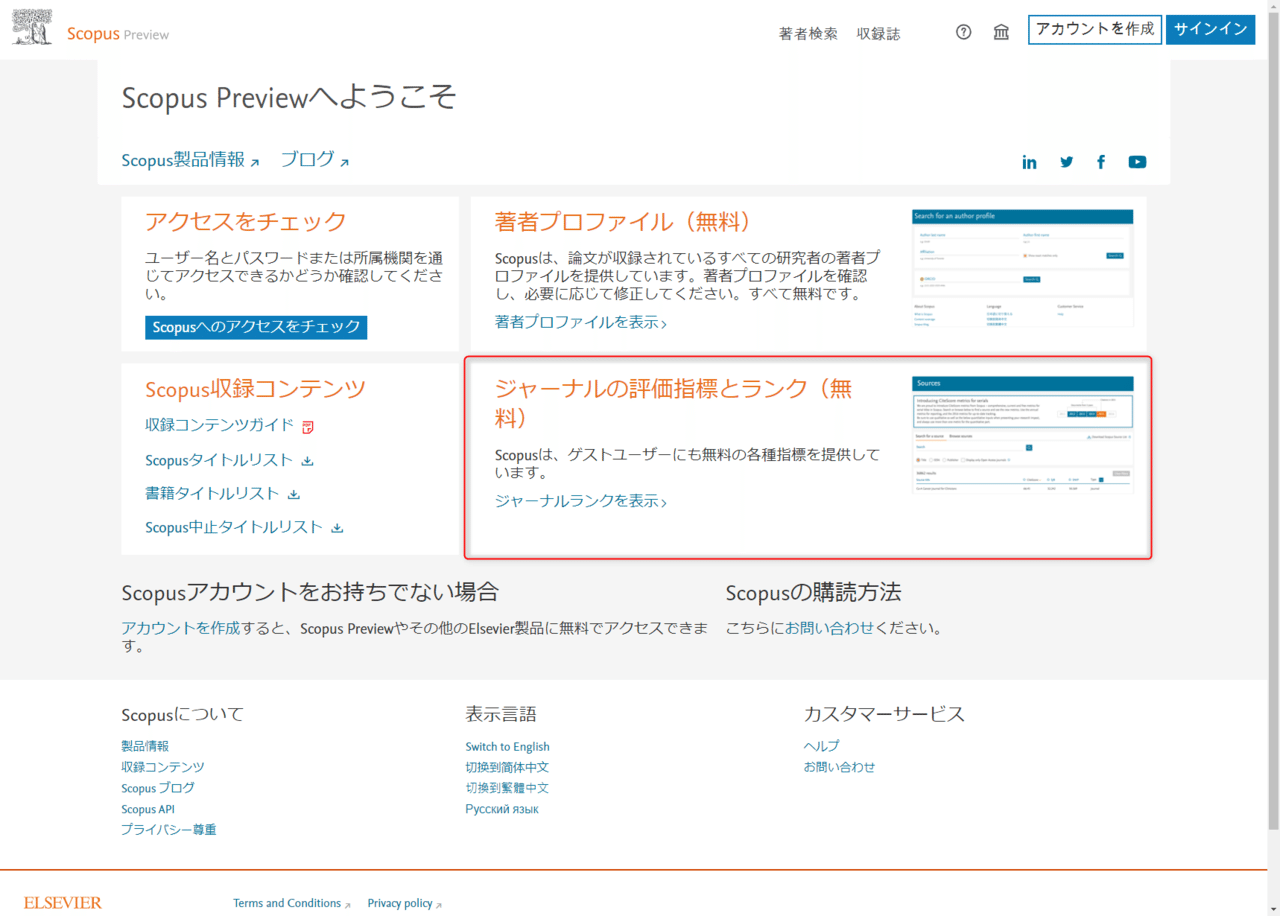
Scopus
ScopusのHPから”ジャーナルの評価指標とランク(無料)”の”ジャーナルの評価指標とランク”をクリックします。
Scopusタイトルリストをダウンロードをクリックします。そうすると”CiteScore-2011-2020-new-methodology-May-2021.xlsb.zip”というファイルがダウンロードされるので解凍してください。
ファイルを回答すると、2つのファイルが生成されるので、”CiteScore 2011- ~”というxlsbファイルを開いてください。
CiteScore 2020のシートをクリックしてください。
ファイルをクリックしてください。
”名前をつけて保存”をクリックします。
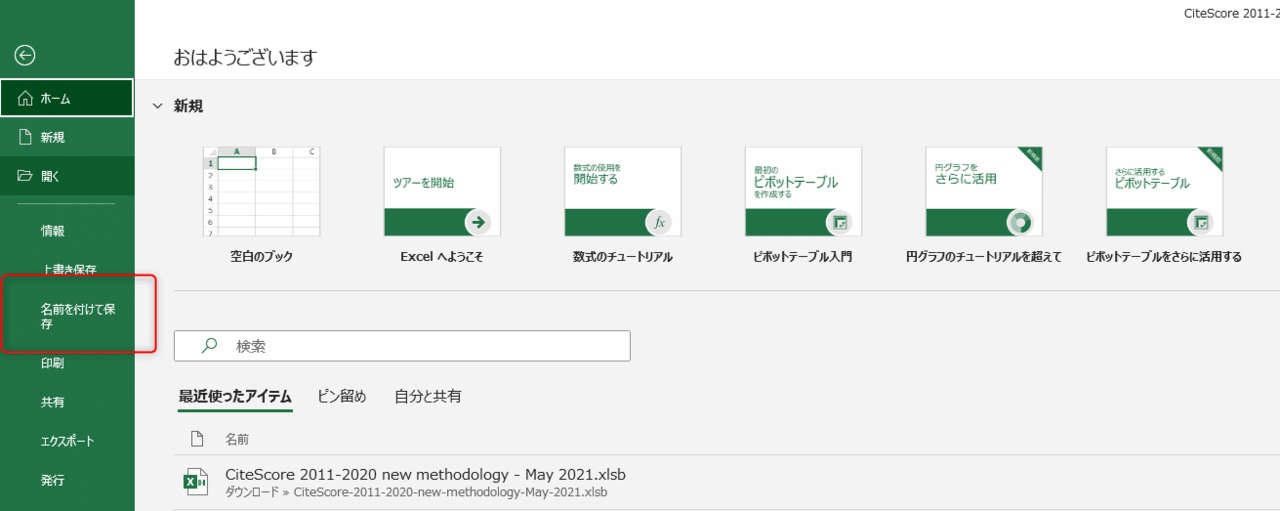
csvファイルで保存し直します。
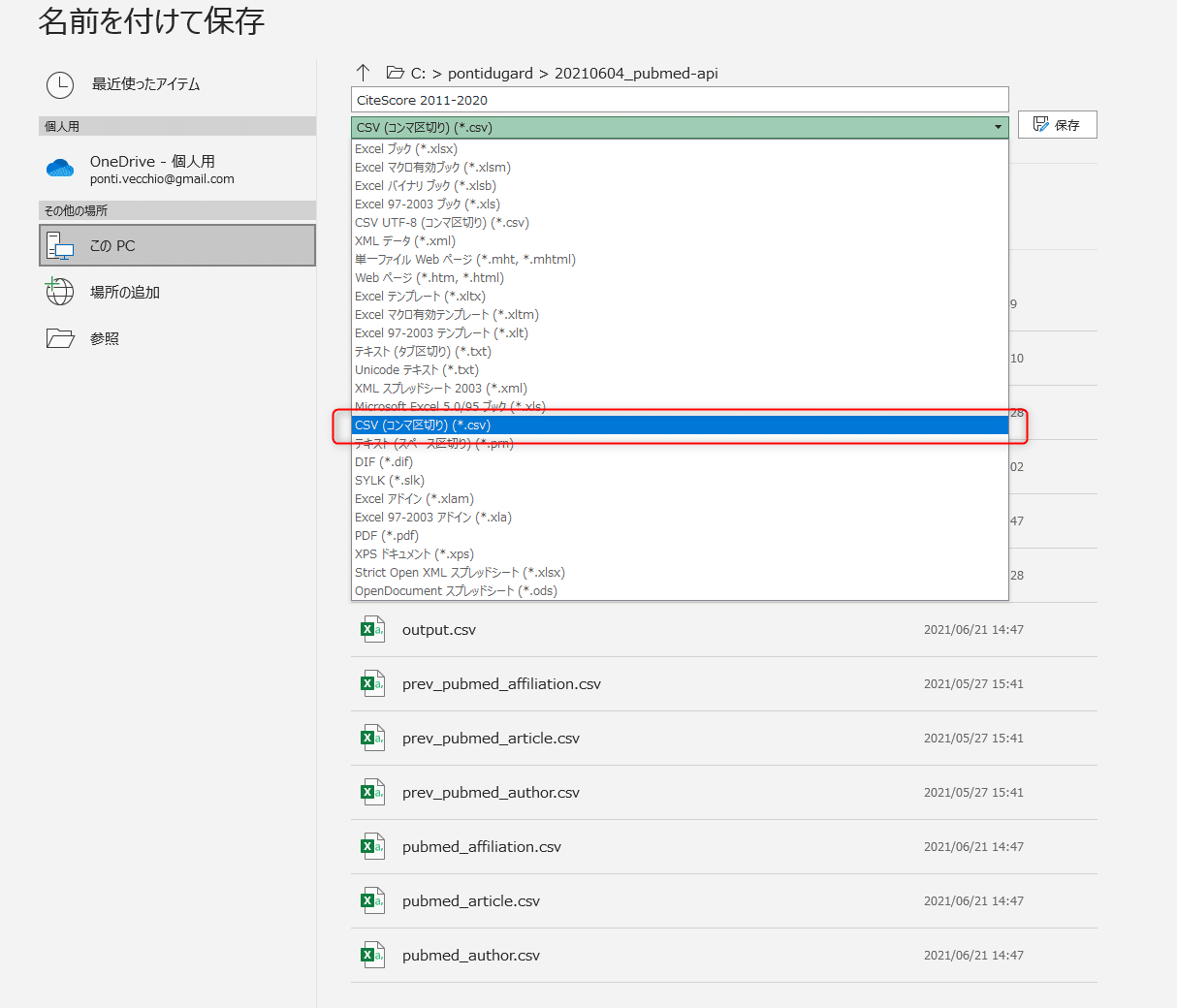
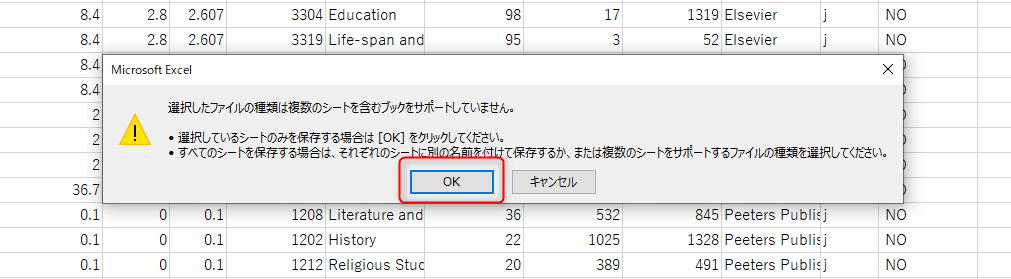
選択したシートのみ保存したいので、OKをクリックします。
新しく.csvファイルが生成されていればOKです。
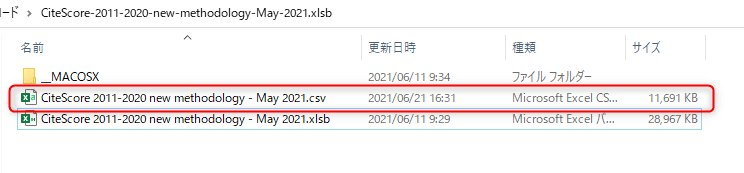
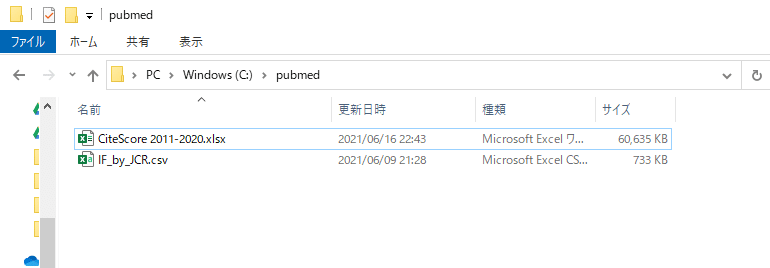
ダウンロードしたJournal citation report のファイルをIF_by_JCR.csv、ScopusのファイルをCiteScore 2011-2020.csvと名前を変更し、pubmedフォルダに入れておいてください。以下の画像のようになっていればOK。
DeepLのAPI取得
DeepLで論文のタイトルとアブストを和訳するために、DeepLのアカウント作成とAPI keyを取得する必要があります。API keyとはDeepLをプログラミングで利用するときに必要なパスワードのようなものです。無料で作成できますので、手順どおりに作成してみてください。
(無料ですが、クレジットカード情報を登録することを求められます。それが嫌な場合は精度は下がってしまいますが、前述のGoogleの翻訳を利用しましょう。)
DeepLのHPから、”開発者向け”をクリック。
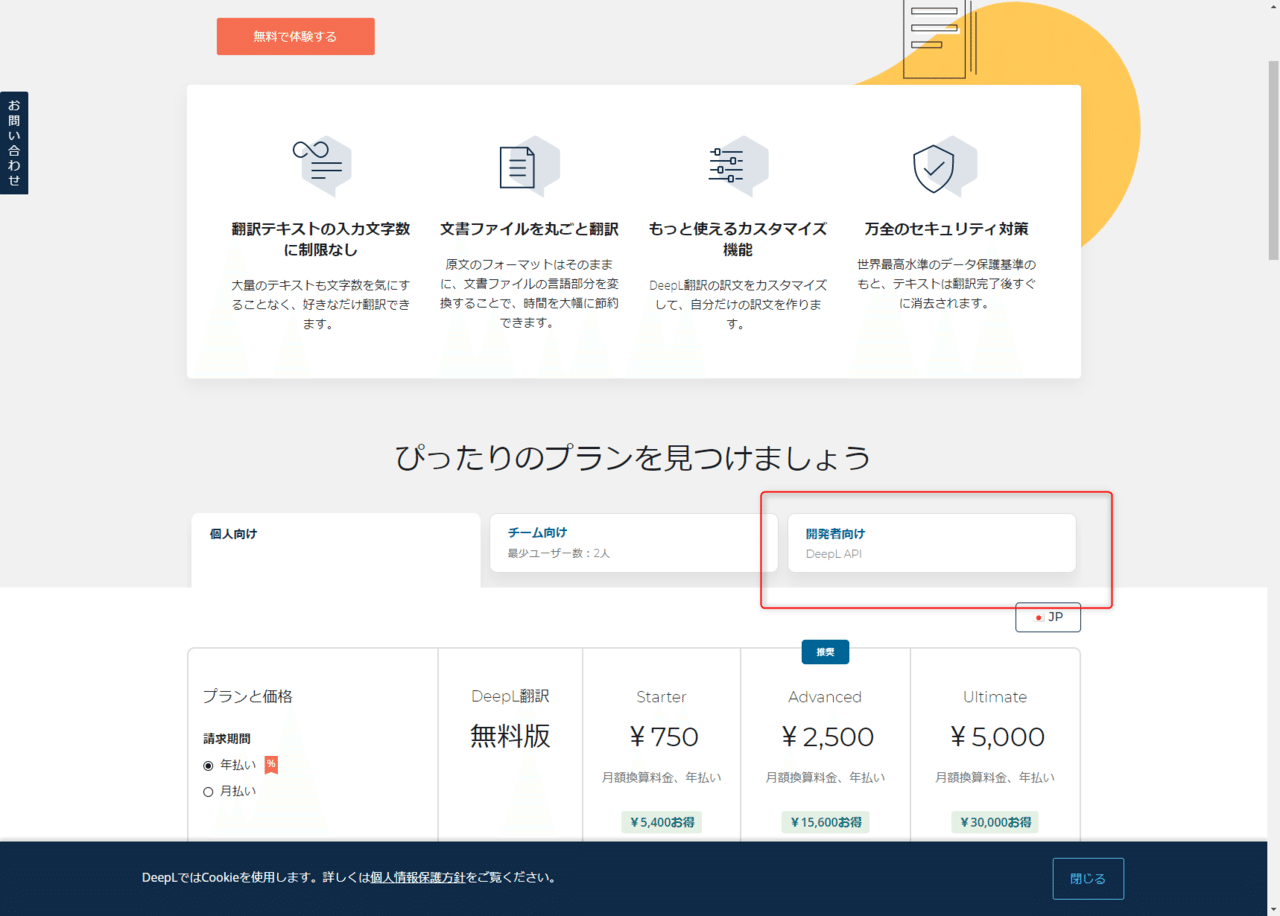
”無料で登録する”をクリック
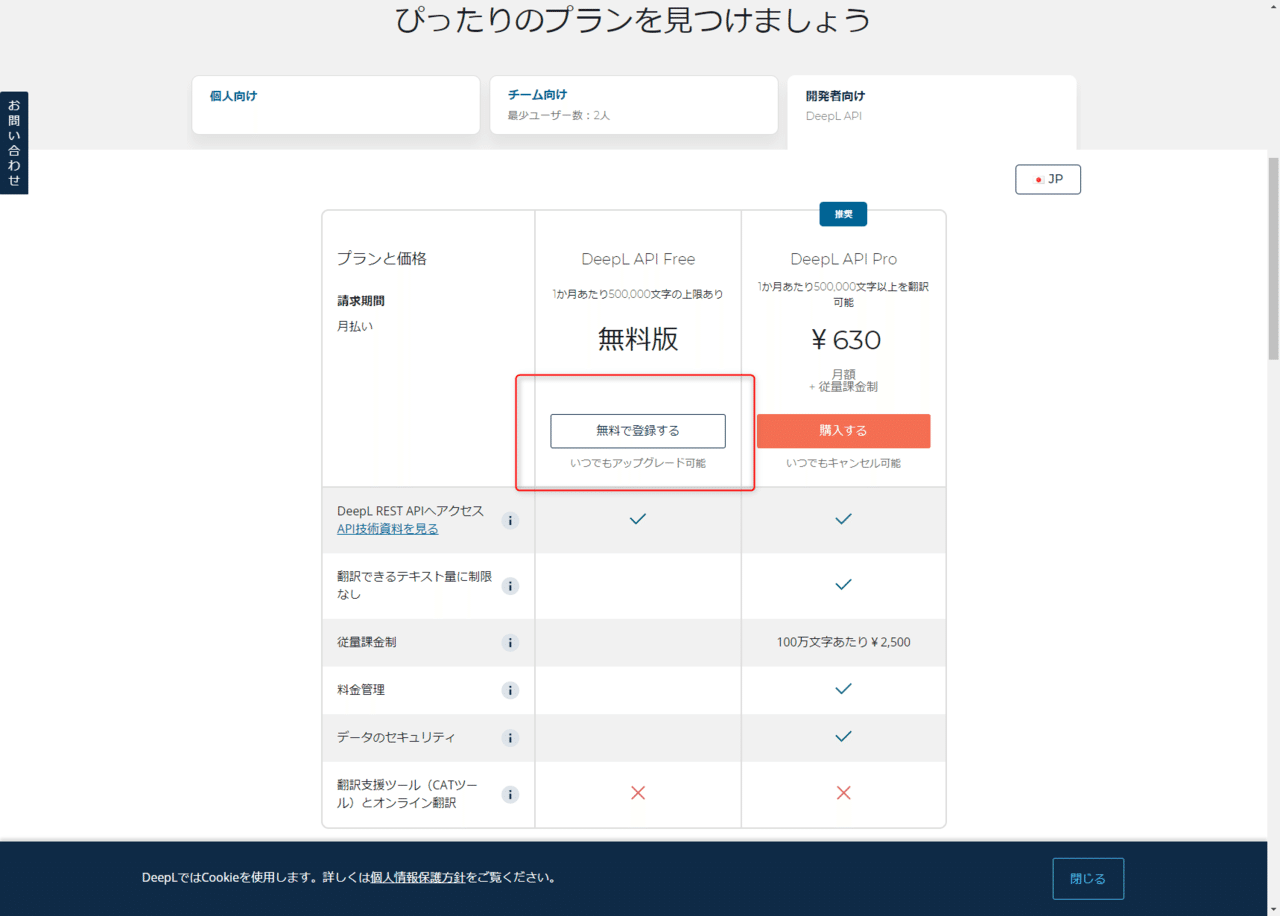
メールアドレスやパスワードの設定を行ってください。
アカウントが作成できたら、アカウントページのプランをクリックします。

そうすると”DeepL APIで使用する認証キー”が表示されていればOKです。
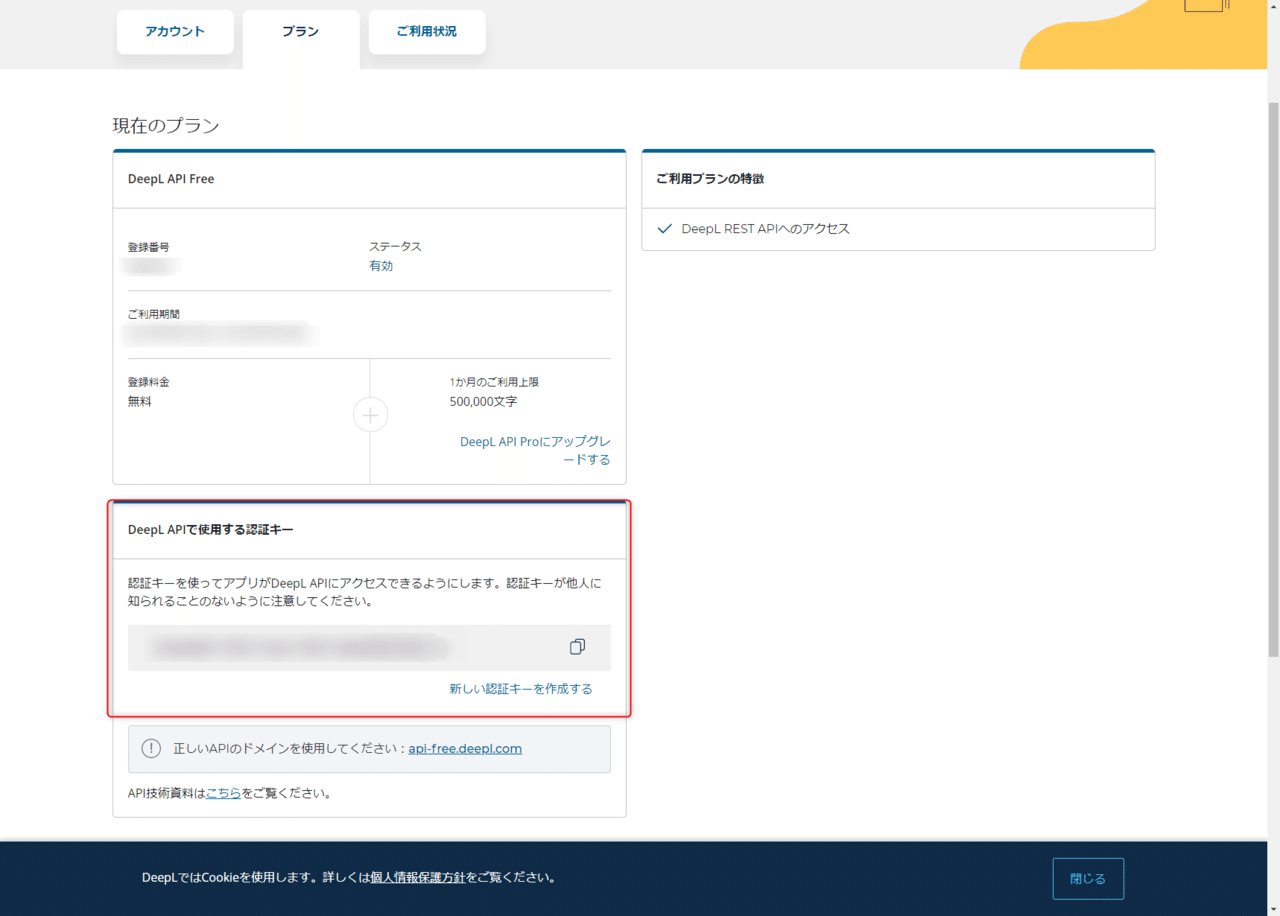
この認証キーをコピーし、Jupyter Notebookの中のdeepl_keyがXXXXXXXとなっているので、そこに貼り付けて置き換えてください。
DeepLのAPIは無料枠だと1ヶ月50万文字数の制限があるので、検索ワードや開始、終了月日などを調節し、ある程度絞っていたほうが良いでしょう。Pubmedでいつものように検索して件数を確認してから実行してもいいと思います。
終わり
いかがでしたでしょうか。できるだけプログラミングが初めてという方にもできるようにどうしたらいいかと考えた結果、Noteにコードを貼り付けておき、それをコピーしてもらうという手法を取りました。
ここでは書きませんでしたが、Jupyter notebookをpythonファイルに出力し、タスクスケジューラーで毎日もしくは毎週1回自動で実行、前回のcsvファイルとの差分だけをメールで送信などすると、日本語に訳された論文アブストなどが手に入ります。今後の論文検索をより快適に過ごせるように役に立てたら幸いです。
この記事が気に入ったらサポートをしてみませんか?
