データサイエンス超入門 第二回
第二回ってか今回使ってるスライドの最後までここに記録する。
1.二乗誤差
被説明変数yを説明変数x=(x_1,x_2,...,x_k)とパラメータ(a_0,a_1,...,a_k)を用いて
y = a_0 + a_1*x_1 + a_2*x_2 + ... +a_k*x_k
で推定する。
実際(x,y)として行列data(各行が一つのベクトルxと値y)が得られた時、二乗誤差を求めるプログラムを書け。dataの行が(1,2,-1,1),(2,1,1,0),(-1,1,3,2),(2,1,0,1),aが(1,-1,1,2)の場合の二乗誤差を求めよ。
data <- rbind( c(1,2,-1,1), c(2,1,1,0), c(-1,1,3,2), c(2,1,0,1) )
a <- c(1,-1,1,2)
b <- c(a,0)
X <- cbind(rep(1,nrow(data)), data)
error2 <- sum((X%*%b-data[,ncol(data)])^2)
print(error2)
こんくらいなら手計算できるんでそっちもおいておく。紙を捨ててよくなるからnoteいいね。
2.確率

箱から果物を取り出す行為を模擬する乱数を10000個発生し、上記問題の確率と比較せよ。
box <- rbinom(n=10000,size=1,p=1/2)
%sum(box)は10000回箱を選んだ時に赤い箱を選ぶ回数
redbox <- rbinom(sum(box), 1, 3/5)
%sum(redbox)はsum(box)回赤い箱から引いた時リンゴを引く回数
bluebox <- rbinom(10000-sum(box), 1, 2/6)
%sum(bluebox)は10000-sum(box)回青い箱から引いた時リンゴを引く回数
red_apple <- sum(redbox)
red_orange <- sum(box)-sum(redbox)
blue_apple <- sum(bluebox)
blue_orange <- (10000-sum(box))-sum(bluebox)
prob_1 <- (sum(box)/10000) * (red_orange/sum(box))
prob_2 <- (prob_1) / (sum(box)/10000)
prob_3 <- prob_1 + ((10000-sum(box))/10000) * (blue_orange / (10000-sum(box)))
prob_4<- prob_1/prob_3最初に思いついたのがこう。解答例では一様乱数を丸めることでこれと同じことをやってた。
3.尤度

p=0.6の場合に、100回引くくじ引きを1000回行い、各回のあたり数の度数グラフを書くプログラムを作成せよ。
raffle <- rbinom(n=1000, size=100, p=0.6)
hist(raffle,breaks=seq(30,80,1),main="Histgram of raffle",xlab="Range",ylab="Frequency",col="gray")知ってる引数をできるだけ使ってヒストグラムを書いた。ググったらくじ引きって英語でraffleって言うらしい。

4.ベイズ統計

プログラムを書いて1の尤度と2の事後確率分布をグラフで表せ。
comb = function(n,k){
result = exp( sum(log(1:n)) - sum(log(1:k)) - sum(log(1:(n-k))) )
return(result)
}
%_nCk_を求める関数combを作成
p = seq(0,1,length=100)
%pは0から1までの値を一様に取りうるとする
LH100 = comb(100,60) * exp(60*log(p) + 40*log(1-p))
%100回引いて60回当たった時の尤度
work = LH100 * dnorm(p,mean=0.3,sd=0.1)
%workは尤度×事前分布。(p=0.01あたりは重みが軽くなる)
plot(p,LH100,xlim=c(0,1),ylim=c(0,0.4),xlab="p",ylab="pdf",type="l",col="black")
%尤度の分布。無情報事前分布だと確率推定値と最尤推定量は一致する
par(new=T) %上書き指定
post100 <- work/sum(work) %和が1になるよう規格化
plot(p,post100,xlim=c(0,1),ylim=c(0,0.4),xlab="",ylab="",type="l",col="blue")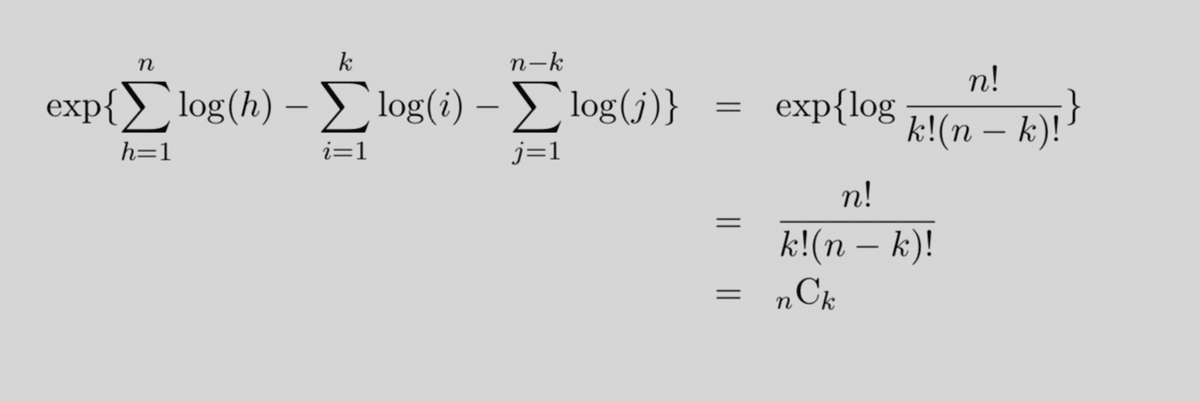

5.回帰分析
単位面積当たりの収量yを施肥量xの関数として表す回帰モデルを考える。
1)以下のデータが得られた時、y=a_1x+a_0というモデルを仮定し、Rの線形回帰関数lm(formula,data)を用いて、回帰分析を行い、x=17の場合のyを推定せよ。
(x,y)=(10,30), (15,41), (22,50)
dataは説明変数のdata.frameでなければならないので、xという列名をもつ1列からなるdata.frameを作る(data.frame(x=c(10,15,22))でできる)。formulaには、yをxで説明するということを記述するという意味の表現y~xで与える(formula=y~xと書く)。lmの出力の一部として推定したパラメータのベクトル(定数項、説明変数の係数の順)で得られる。例えば、lmの出力をestとするとest$coefficientとすると得られる。これからx=17の場合のyの推定値を得る。
次に、 y=a_1x^2+a_0というモデルを仮定し、x=17の場合のyを推定せよ。
x = data.frame(x=c(10,15,22))
%「x=」が大事。xという列名をもつdataframeになる。
y = c(30,41,50)
est = lm(formula=y~x, data=x)
%モデル式y=a_0+a_1*xについて、目的変数をy、説明変数をxとする。
test_data = matrix(c(17),ncol=1)
est_test = cbind(rep(1,nrow(test_data)),test_data)
predict_test = apply(est_test, 1, function(x){
return( sum(x*est$coefficient) )
})
%apply(行列orベクトルor配列, MRGIN(1なら行について。) ,関数)
predict_test
est$coefficient
x2 = c(10,15,22)^2
x = data.frame(x=x2)
y = c(30,41,50)
est = lm(formula=y~x,data=x)
test_data = matrix(c(17*17), ncol=1)
est_test = cbind(rep(1,nrow(test_data)),test_data)
predict_test = apply(est_test, 1, function(x){
return(sum(x*est$coefficient))
})
predict_test
est$coefficient2)上記データ、モデルに対して、前ページの式を直接計算することでパラメータa1、a0を求め、x=17の場合のyを推定し、1)の答えと整合することを確認せよ。
x = c(10, 15, 22)
y = c(30, 41, 50)
F = cbind(rep(1, length(y)), x)
a = solve( t(F)%*%F ) %*% (t(F)) %*% y
%solveで逆行列を求める
test_data = matrix(c(17), ncol=1)
est_test = cbind(rep(1,nrow(test_data)), test_data)
predict_test = apply(est_test, 1, function(x){
return(sum(x*a))
})
predict_test
a
x = c(10, 15, 22)
y = c(30, 41, 50)
F = cbind(rep(1, length(y)), x^x)
a = solve( t(F)%*%F ) %*% (t(F)) %*% y
test_data = matrix(c(17*17), ncol=1)
est_test = cbind(rep(1,nrow(test_data)), test_data)
predict_test = apply(est_test, 1, function(x){
return(sum(x*a))
})
predict_test
a6.ロジスティック回帰

data = iris
%データセットirisは3種類の花、各50サンプルずつで計150サンプル、
%がく片と花びらの幅と長さに関する4つの特徴量からなる
data2 = data[data[,5]!="setosa",]
%dataのうち、5列目の値が"setosa"でない行を抜き出したデータセットをdata2とする
%ロジスティック回帰なので、所属クラスを2つにした
test_id = sample(nrow(data2),10)
%data2の行数の中から行の値を10個無作為抽出してtest_idとおく
test_data = data2[test_id,]
%data2からデータを10個取り出し、test用データとする。残りがパラメータ推定用の訓練データ
train_data = data2[-test_id,]
%パラメータ推定用のデータをtrain_dataとする
est = glm(formula=Species~.%Speciesをそのほかの変数で推定する
,data=train_data, family=binomial)
u = cbind(rep(1,nrow(test_data)),data.matrix(test_data[,-ncol(test_data)]))%*%est$coefficient
%1とtest_dataの"Species"以外の係数を並べた行列にパラメータをかける
result = 1/( 1+exp(-u) )
%ロジスティック変換
cbind(result,test_data[,ncol(test_data)])7.主成分分析
data_iris = iris
id = sample(nrow(data_iris), 10)
%data irisの行数の中から行の値を無作為に10個取り出してidとする
data = data_iris[id,-ncol(data_iris)]
%data irisのうち番号idの行のSpecies以外のデータをdataとする。形はデータフレーム。
pca = prcomp(data)
biplot(pca,choices=1:2)8.K-NN(教師あり)
k-NN(k近傍法)は自分の周りにある近い順にk個のサンプルを抽出し、最も多いクラスに分類する。Rでは、knn(訓練データの説明変数, テストデータのでつ名変数, 訓練データの被説明変数, k)と入力することで、テストデータの分類結果を返す。
irisをテストデータと訓練データに分け、テストデータを分類せよ。
data = iris
test_id = sample(nrow(data), 10)
%テスト用データの番号を乱数発生。
test_data = data[test_id,]
train_data = data[-test_id,]
%dataからテスト用データを抜いたものを訓練用データとする。
library(class) %これ必要??
result = knn(train_data[-ncol(train_data)],%train_dataの被説明変数以外の列を訓練データとして代入
test_data[,-ncol(test_data)], %test_dataの被説明変数以外の列をテストデータとして代入
train_data[,ncol(train_data)], %train_dataの被説明変数を代入
k = 5
)
cbind(test_data[,ncol(data)], result) %なんでここ数値になるん?9.K-MEANS(教師無し、クラス数は既知)
k-MEANSでは、まずデータをランダムにk個のクラスにそれぞれのデータについて各クラスの中心点との距離を計算し、クラス替えを行う、という操作を繰り返すことで分類する。Rでは、kmeans(説明変数, centers)でクラスターに分類する。
irisのSpeciesを推定せよ。
data = iris
x = data[,-5]
%dataからSpiciesの列を抜いてxと置く。
result = kmeans(x,centers = 3)
%クラス3つでK-MEANSを実施
table(data[,5], result$clusters=3)
%tableでdata[,5]とresult$clusterの対応表を作成10.階層クラスタリング(教師なし)〜樹形図の構成〜
階層クラスタリングでは、クラス数は求められないが、任意のクラス数の分類が得られる。まずは、各データを要素1つのクラスタとみなし、そこから代表点の近いクラスタ同士を結合していき、その過程を樹形図として作る。
11.決定木、ランダムフォレスト(教師あり)
決定木とは、あるデータを特徴量に応じて分類することを考えた時に、分類により最も純度が上がるように分類する。
ランダムフォレストとは使用する訓練データや特徴量を様々に変えて決定木の操作を行い、それらを総合化して分類の結論を出す、いわゆるアンサンブル学習。
data = iris
test_id = sample(nrow(data), 10)
%dataから行数を10個抜き出し、テストデータの行番号test_idと置く
test_data = data[test_id,]
train_data = data[-test_id,]
%test_idでない行番号のデータを訓練データtrain_dataと置く。
library(randomForest)
train_result = randomForest(formula=Species~., data=train_data)
%Speciesをそのほかの変数で説明するrandomForestを実行する。
result = predict(train_result, test_data)
%train_resultの結果をもとに、test_dataのSpeciesを推定
cbind(result, test_data[,ncol(test_data)])
12.線形分類器(SVM)(教師あり)
データをある直線で分類することを考える。この直線を平行移動しても分類が変わらないような移動範囲(マージン)が最大化されるような直線を選ぶ。教師あり学習で分類器を構成し、以降分類器でデータを分類できる。Rではksvm(特徴量データ,特徴量データの分類,type=C-svc,kernel="vaniladot")で直線を求める。
irisのSpeciesを2分類で解け。
install.packages("kernlab")
library( "kernlab" )
data = iris
data2 = data[data[,5]!="setosa",]
%dataのうち、Speciesが"setosa"でない行をdata2と置く(2分類にする)
sample_id = sample(nrow(data2),10)
%data2農地、行番号を10個抽出してsample_idと置く。
x = as.matrix(data2[sample_id,c(1,3)])
%行番号がsample_idのデータの1列目、3列目を抜き出して行列化し、xと置く。
%なんで変数を2つに限定したのか忘れたって言ってた
y = data2[sample_id,ncol(data)]
%data2のsample_idの行のSpeciesの列を抜き出してyと置く。
z = data.frame(x,y)
%z = data2[sample_id,c(1,3,5)]と一緒
result = ksvm(x,y,kernel = "vanilladot", type = "C-svc")
%ksvm(x,y,type,kernel)で説明変数xでyを説明するためのsvmを実行する
plot(result,data=x)ランダムフォレストとSVMの長所と短所みたいなもんってあるんだろうか
この記事が気に入ったらサポートをしてみませんか?
