
MATLABとPythonとで作図してみた感想
僕は普段RとMATLABを使っているわけだが,個人的な興味と勉強目的と(8:2)でPythonも使ってみようかなということで,とりあえずMATLABで作ったコードのPythonへの移植を試みた。
内容としては以下の通り。
NIRSデータをcsvファイルから読み込む。
線形トレンドと正弦波ノイズの付与
MRAを使った特定の周波数帯域のデータの抽出
元のデータと抽出したデータに対する周波数解析
各データとそれに対する周波数解析の結果についての作図
(ただし,MRAに関しては同じ処理ができなかった(後述)ので,MATLABとPythonとで結果が異なる)
実行環境は以下の通り。
MacBook Pro (14-inch, M1Max), macOS Monterey (12.6.5)
MATLAB R2022a(大抵のTool Boxは導入済み)
Spyder (Python 3.10.9)
図の作成で比較
まずは作った図を比較してみる。上がMATLABで作成したもの(図1),下がPythonで作成したもの(図2)。

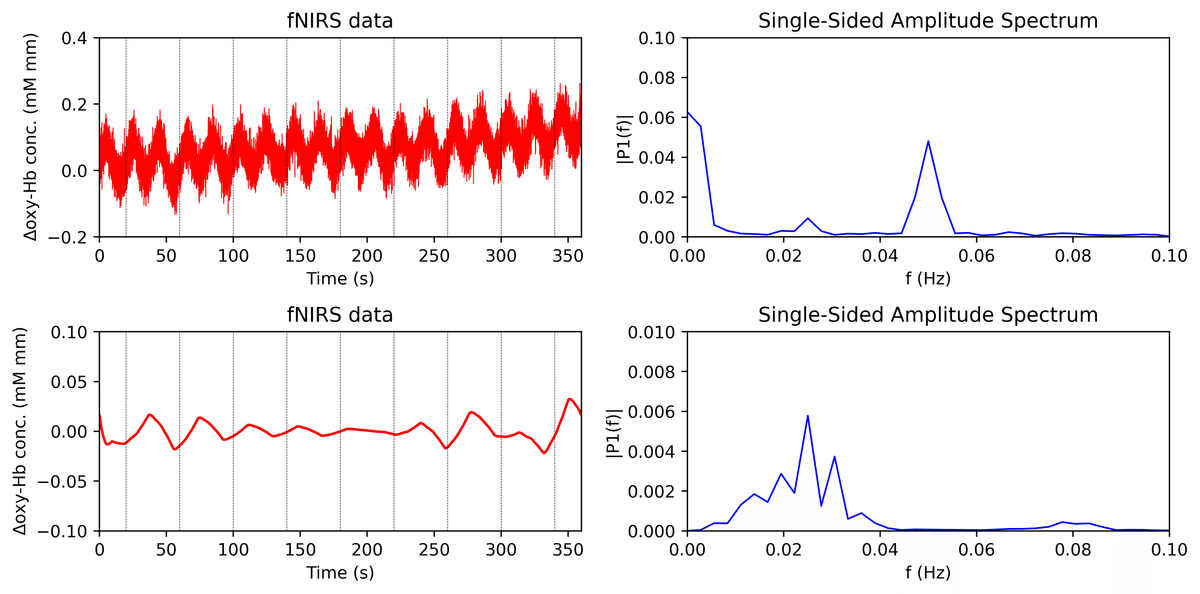
フォントや色合いといった細かい部分の違いはさておき,同じものを作ることができた。
結果としてはいいのだが,図の作成についてはMATLABの方が融通が効くなーという印象を持った。
というのも,MATLABの場合は1つ1つの操作がリアルタイムで図に反映されるが,Pythonの場合は一通りコードを作ってからまとめて実行しないと図に反映されなかった。
具体的には,例えば,MATLABなら上の4つのsubplotを表示した後で特定のsubplotについてラベルだけ変更するといったこともできるのだが,Pythonの場合は図の一部を修正しようとすると,図に関する部分のコードを全て読み込ませないとそれが反映されなかった(つまり図を全て作り直すことになる)。
あと図の画質の問題もある。MATLABは最初からそれなりに高画質の図を表示してくれるのに対して,Pythonはあらかじめ画質を設定しないといけない。これも地味に面倒。
さらに言えば,MATLABの図は目盛りがデフォルトで内向きになっているのも良い(今回はPython側ではその設定をしていないから外向きになっているけど)。
MATLABばかり褒めているけど,今回の作図でPythonの方が優れていると思った点は,subplotで図を複数作成した際の個々の図間をいい感じに調整できるところ。MATLABだと余白が大きいと感じることもあるので(もちろん調整できるが結構面倒)。
結論として,やり方や環境や慣れの問題かもしれないが,現時点では作図ならMATLABの方が使いやすいと感じている。
PythonでのMRAについて
僕はMATLABではWaveletToolBoxに含まれている関数"modwt"と"modwtmra"を使ってMRAを行っている。
Pythonでもこれと同じことができないかと探してみたところ,全く同じことができるようにと作成されたものを見つけた(https://github.com/pistonly/modwtpy)。
しかしながら,説明通りにやってみても"modwt"を実行したときの結果がMATLABでのそれと違っているし,"modwtmra"を実行すると結果が全て0になってしまった。
MRAを実行するまでのデータはMATLABでのデータと一致しているし,配布されているサンプルデータとサンプルコードを使ってみても"modwtmra"の結果は全て0になったため,配布されているコードに問題があると思われた。
そのため,今回は"pywt"というモジュールに含まれている関数"mra"を使ったが,やはり計算方法が異なると思われ,当然だが出力された結果はMATLABのものとは一致しなかった(図1, 2)。
ちなみに,これまた当然だが,マザーウェーブレットも分解レベルも第何成分を抽出したのかもMATLAB側とPython側とで同じだし,両方ともDWTである。
まあ,全く違う結果になっているわけではないのだが,やはり気になる…
コードの比較
コードも比較してみよう。
ただ,どこで何をしているのかをいちいち説明するのは面倒なので,興味がある人は考えながら読んでいってほしい(MATLABとPythonとで処理する順序は統一してある)。
(なお,実際には必要な部分を必要に応じて調整しながら実行しているため,このコードをそのまま実行しているわけではない)
まずはMATLABから。
workDir = pwd;
dataDir = '/XXX/XXX/XXX';
cd(dataDir);
raw = readmatrix('XXX.csv');
time = raw(:,1);
[on, ~] = trigPoint(raw(:,2), 9); %自作関数
Fs = 1/raw(2,1);
data = raw(:, 3*22+[2:3]);
dat = data(:, 1);
%-------- add linear trend --------
tmp = [1:length(dat)];
tmp = tmp*0.000005;
tmp2 = tmp';
dat = dat+tmp2;
%-------- add optional wave --------
Hz = 0.05;
Mgn = 0.05;
y = sin(2*pi*Hz*time)*Mgn;
dat = dat+y;
%-------- MRA --------
wv = 'sym4';
level = 12;
mra = modwtmra(modwt(dat, wv, level), wv);
exdat = mra(11, :);
exdat = exdat';
%-------- FFT --------
x = dat'; %FFTしたいデータをxに代入する
L = length(x);
W = hamming(L)';
xh = x.*W;
Y = fft(xh);
acf = 1/(sum(W)/L);
P2 = abs(Y/L);
P1 = P2(1:L/2+1);
P1(2:end-1) = 2*P1(2:end-1);
f = Fs*(0:(L/2))/L;
%-------- plot --------
%(今回は2つ分のみ記載)
subplot(1, 2, 1)
plot(time, dat, 'r');
axis tight
ylim([-0.2 0.4])
xlabel('Time (s)'); ylabel('\Deltaoxy-Hb conc. (mM mm)');
title('fNIRS data');
hold on
yl = ylim;
for jj = 1:length(on)
plot(time(on(jj))*[1,1], yl, 'k:');
end
hold off
subplot(1, 2, 2)
plot(f, P1*acf, 'b', 'LineWidth', 2)
title('Single-Sided Amplitude Spectrum')
xlabel('f (Hz)')
ylabel('|P1(f)|')
xlim([0 0.1])
ylim([0 0.1])
続いてPython。
import os
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import pywt
oldDir = os.getcwd()
dataDir = '/XXX/XXX/XXX'
os.chdir(dataDir)
raw = pd.read_csv('XXX.csv')
time = raw.iloc[:, 0]
#-------- 自作関数"trigPoint"に相当する部分 --------
nBlock = 9
on = np.zeros(nBlock, dtype = int)
j = 0
mark = raw.iloc[:, 1]
for i in range(1, len(mark)):
if (mark.iloc[i-1] == 0) and (mark.iloc[i] == 1):
j += 1
on[j-1] = i
#------------------------------------------------
Fs = 1/raw.iloc[1, 0]
data = raw.iloc[:, 3*22+np.array([2, 3])-1]
dat = data.iloc[:, 0]
#-------- add linear trend --------
tmp = np.arange(1, len(dat)+1, 1)
tmp = tmp * 0.000005
dat += tmp
#-------- add optional wave --------
Hz = 0.05
Mgn = 0.05
y = np.sin(2 * np.pi * Hz * time) * Mgn
y = y.to_numpy()
dat += y
#-------- MRA --------
wv = 'sym4';
level = 12;
mra = pywt.mra(dat, wv, level = level, transform = 'dwt')
exdat = mra[2]
#-------- FFT --------
x = dat #FFTしたいデータをxに代入する
L = len(x)
W = np.hamming(L)
xh = x * W
Y = np.fft.fft(xh)
acf = 1/(sum(W)/L)
P2 = np.abs(Y/L)
P1 = P2[0:int(L/2)+1]
P1[1:] = 2 * P1[1:]
f = Fs * (np.arange(0, int(L/2)+1, 1)/L)
#-------- plot --------
#(今回は2つ分のみ記載)
fig, axs = plt.subplots(1, 2, figsize = (10, 5), dpi = 500)
axs[0].plot(time, dat, 'r', linewidth = 0.5)
axs[0].set(xlim = (0, 360), ylim = (-0.1, 0.1), xlabel = 'Time (s)',
ylabel = '$\u0394$oxy-Hb conc. (mM mm)', title = 'fNIRS data')
for i in on:
axs[0].axvline(x = time[i], color = 'k', linestyle = 'dashed', linewidth = 0.3)
axs[1].plot(f, P1*acf, 'b', linewidth = 1)
axs[1].set(xlim = (0, 0.1), ylim = (0, 0.1), xlabel = 'f (Hz)',
ylabel = '|P1(f)|', title = 'Single-Sided Amplitude Spectrum')
plt.tight_layout()とまあこんな感じ。
変数名の設定とか値渡し云々など,記述方法についての批判は受け付ける気はないので悪しからず(例えば,変数名をアルファベット1つだけにしているとか,ループで"i"を使うとか,結構ツッコミどころは多いと思うけど)。
Rから始まってMATLABに馴染んでいる僕としては,データ型の問題とか,配列の1番目を指定する際には1ではなく0だとか,範囲の指定の際の終点はその1つ手前までだとか,とても面倒に感じた。
あと"for"とか"if"とかの終わりに"end"を付けないことが気持ち悪い。
ただまあ,Pythonについてはちまちま触ったことはあるが,作図やらデータの読み込みやらまともにいじったのは今回が初めてで,なんならモジュールもnumpyしか知らなかったわけだから(前回の記事の内容も序盤も序盤でつまづいているので),それを考えたら上出来なのではないだろうか。
おわりに
Pythonはわかりにくいと感じたが,これでもまだ易しい方で,RやMATLABはPythonに限らず他の言語と比べてもずっと簡単なんだろうなー。
この記事が気に入ったらサポートをしてみませんか?
