
【統計ソフトR】多群比較のための分散分析
今回は、3群以上の比較をする時に大切な分散分析(ANOVA)をRでやっていきます。
分散分析をするには、まず確認しておかないといけないことがあるので、ちょっとややこしいです。具体的には、正規性の検定と分散等質性の検定を行なわなければなりません。それを全部Rでやります。
0. データの読み込み
この記事では、データはRにデフォルトで入っている練習用セット「iris」を使います。このデータはアヤメ(花)の3品種各50サンプルについて、がくの長さ、幅、花弁の長さ、幅を計測した数値です。
まずはデータを読み込んでください。
data(iris)
なお、今回は関係ないですがExcelのCSVファイルを読み込むには、「ファイル」タブから作業ディレクトリを使いたいファイルが入っている場所に変更してから、
iris<-read.csv("ファイル名.csv")
と入力します。
どんな変数が入ったデータなのかわからないので、列の一番目だけを表示して変数名を見ます。
colnames(iris)

「Sepal.Length」「Sepal.Width」「Petal.Length」「Petal.Width」「Species」という5つの変数があることがわかりました。
それでは、例として「Sepal.Width」が「Species」という群間でどのような差が出ているかについて分散分析をします。
1. 前提① 正規性の検定
分散分析をするには、「Sepal.Width」に正規性がないといけません。つまり「Sepal.Width」の母集団が正規分布である必要があるのです。正規性がない時はノンパラメトリック手法をとるのですが、それについては割愛します。
では、正規性があるかどうか確認していきます。
次のコードを実行し、「ggpubr」パッケージを利用して正規性に関するプロット図を出してみます。
install.packages("ggpubr") #パッケージのインストール
library(ggpubr) #パッケージの呼び出し
qqnorm(iris$Sepal.Width) #プロット
qqline(iris$Sepal.Width) #線引き
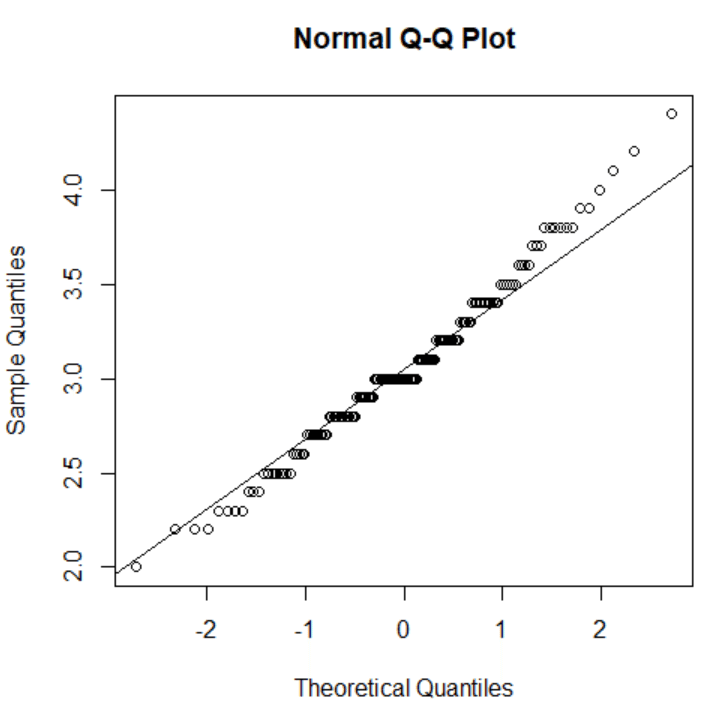
y=xの直線上にプロットが集まっているほど正規性があることを意味します。これはどうも「Sepal.Width」に正規性がありそうですね。
プロットだけで判断しにくい場合はShapiro-Wilk検定という方法で正規性に関する検定ができます。
shapiro.test(t(iris$Sepal.Width))
実行すると末尾に「p-value=0.1012」と表示されます。このpが0.05より大きい場合は正規性を持つといえるので、やはり「Sepal.Width」には正規性がありますね。これで次の段階に進めます。
2. 前提② 分散等質性の検定
分散分析をするためにもう一つ確めなければいけないのが分散等質性です。「Species」3群の間で「Sepal.Width」の分散が等しくなければ分散分析が使えません。(分散等質性がない時にはWelchの分散分析を使いますが、ここでは割愛します。)
分散等質性を検定するにはBartlett検定を使います。
bartlett.test(iris$Sepal.Width~iris$Species)
実行すると末尾に「p-value=0.3515」と表示されます。このpが0.05より大きい場合に分散が等しいとされるので、無事分散等質性が確認できました。これでやっと分散分析に入れます。
3. 分散分析
「Species」3群の間で「Sepal.Width」に差はあるのでしょうか。それを知りたい時に行なうのが分散分析です。
今回は「rstatix」パッケージを用いた方法を紹介します。次のようなコードを書きます。
install.packages("rstatix") #パッケージのインストール
library(rstatix) #パッケージの呼び出し
anova_test(data=iris,Sepal.Width~Species)
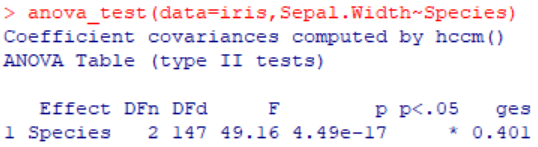
このような結果が返ってきます。今回はp=4.49e-17でp<0.05なので、「Species」3群の間で「Sepal.Width」に差があることがわかりました。
4. 事後テスト
分散分析だけでは「Species」3群のどれに違いがあるかということまではわかりません。「Sepal.Width」に有意な差があることまではわかったので、具体的にどのペアに違いがあるかを事後テストで確認してみます。
やり方はいろいろあるのですが、今回は一般的なTukeyの多重比較検定をしてみます。
tukey_hsd(iris,Sepal.Width~Species)

このような結果が返ってきます。ペア毎に検定をしてくれています。
p値が全て0.05未満なので、全ての「Species」のペアで「Sepal.Width」が有意に異なることがわかりました。また、p値が一番小さいのは「setosa」×「versicolor」のペアなので、このペアに一番大きな差があることもわかりました。
では、今回はここまで。他の記事で基本統計量の算出、相関と検定、様々な図グラフの描き方なども解説しています。
この記事が気に入ったらサポートをしてみませんか?
