
【W5】化合物クラスタリング_02_Step1
【W5の目的】
化合物をグループ化する方法と、多様性のある化合物セットを選ぶ方法
を学びます。Python版TeachOpenCADDのT5が対応しますが、こちらはより発展的です。
【Step1: 階層型クラスタリング】
W5の本体であるメタノード「5. Compound clustering」

のStep1で階層型クラスタリングを体験します。
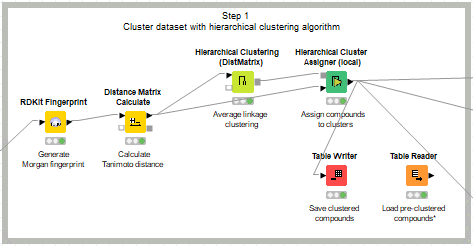
W4と同様にMorganフィンガープリントを算出したのち、タニモト距離を用いて階層型クラスタリングを実施します。
【RDKit Fingerprint】

デモデータの入力はW4と同じく4511化合物。算出するフィンガープリントは2048ビット半径2のMorganですので、記事を紹介して説明に代えさせていただきます。
設定:

MFP2というカラム名でデータ出力しています。
【Distance Matric Calculate】

日本語化ディスクリプションより
入力テーブルの行のすべてのペアの距離値を計算します。結果は、距離ベクトル値を含む単一の列として入力テーブルに追加されます。
設定:
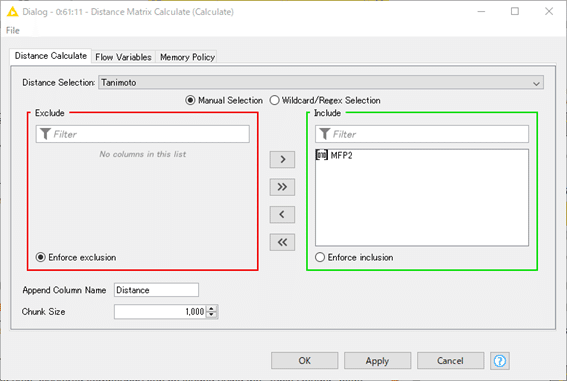
タニモト距離を算出します。
類似度の評価にはタニモト係数を使いましたが、クラスタリングなどでは類似度よりも距離を用いることの方が多いです。
距離は類似度の補数、すなわち(1 - 類似度)です。
結果:
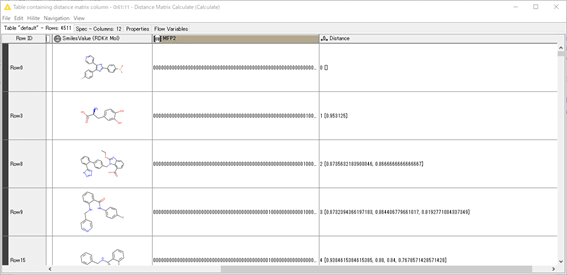
Row0から順に総当たりでタニモト距離がDistance vector型というデータ型で出力されています。
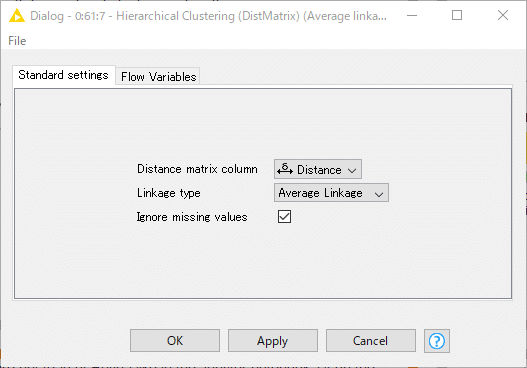
【Hierarchical Clustering (DistMatrix)】設定確認

日本語化ディスクリプションより
距離行列を使用して入力データを階層的にクラスタリングします
設定を確認していきましょう。
【凝集型クラスタリング】
本ノードのクラスタリングの条件ですが、ディスクリプションに
This algorithm works agglomerative.
とありますので凝集型です。詳しくはこちらをどうぞ。
「単純に最も似ているもの同士をくっつければいいのでは?」という人間的な直感をそのままクラスタリング手法に当てはめたアルゴリズムがこの「凝集型クラスタリング」です。
一般的には凝集した状態を上、ばらばらの状態を下とみなすので、ボトムアップクラスタリング(bottom-up clustering)とも呼ばれています。
逆の分割型もあるそうですし、分割最適型もありますが今回のノードは対応していないですし割愛します。
<参考>
【距離定義いろいろ】
また距離は本ノードでは3種選べるのですが、今回は”Average Linkage”を選んでいます。平均距離法ともいうようです。
平均リンケージ:2つのクラスターc1とc2の間の距離を、c1とc2のすべての点の間の平均距離として定義します。
とのことです。
ここも実際にはいろいろと手法があります。
最近隣法:クラスター同士で、最も近いサンプル同士の距離
最遠隣法:クラスター同士で、最も遠いサンプル同士の距離
重心法:クラスター内のサンプルの重心(平均) の間の距離
平均距離法:クラスター同士で、すべてのサンプル間の距離の平均
に加えて一番よくつかわれる
ウォード法では、クラスター内のサンプルの重心(平均)と、そのクラスター内のサンプルとの距離の二乗和に基づいて、2つのクラスターを結合するかどうか決めます。
計算量は多いですが、妥当なクラスタリングの結果を得られやすい、と言われています。
とのことで、”Average Linkage”設定でいいのかなとも思いますが、今回は体験が主目的なのでここまでとします。KNIMEを利用したクラスタリングを掘り下げてくださっているt-kahiさんの記事を紹介しておきます。
KNIMEの「Hieralchical Clustering」ノードを使うのは非常に簡単なのですが,距離に関する手法がRやPythonと同じように色々と選べるわけではないのでその点だけ注意してください.
【Hierarchical Clustering (DistMatrix)】実行時の注意
注:このノードは3次の複雑さがあるため、小さなデータセットでのみ機能します。
とディスクリプションにある通り、デモデータの4511化合物のクラスタリングに数時間以上かかるでしょう。W1-8の全工程で最も計算が重いステップのようです。
もし自分でも再計算などしたい場合はご注意ください。
このノードだけが実行完了しても、どの化合物がどのクラスターに分類されるかは次のHierarchical Cluster Assigner (local)も実行しないとみることができません。
【Hierarchical Cluster Assigner (local)】
日本語化ディスクリプションより
階層的クラスタリングに基づいてクラスターを行に割り当てます。
クラスターの固定数を選択するか、距離のしきい値を入力することができます。
今回はしきい値を0.5としています。
設定:

結果:
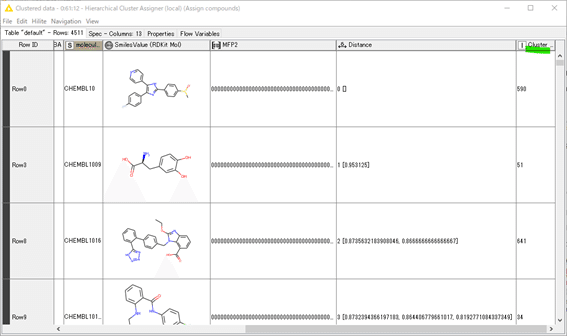
Cluster numberというカラムに
クラスターのID番号がついています。Step2以降で詳しく見ていきましょう。
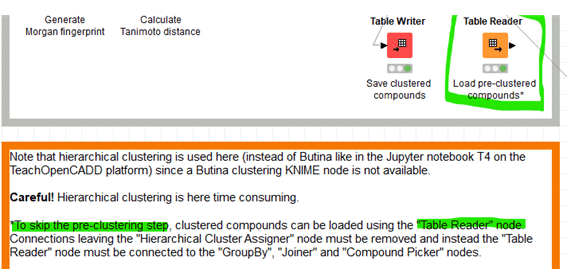
【Table Writer】

初出ですね。便利なので意外です。
まっきーさんは使いすぎに注意とおっしゃっています。
tableというKNIME独自のTableをやり取りするNodeです。KNIME独自のファイルなので、読み込み・書き出し処理が格段に早いです。
KNIMEのWorkflow内でTableをやり取りしたい場合や、中間処理テーブルとして使いたい場合などに有効です。ただし使いすぎるとWorkflowが訳分からなくなるので、多用するのはオススメしません。
私が思うに、一番ありがたいのはKNIMEで利用可能なすべてのデータ型に対応してデータファイルを作成できることでしょう。
今回のデータテーブルはRDKit型、フィンガープリント型、距離ベクトル型など多様なデータ種があるので、csvなどに保存するのは難しいです。
設定:
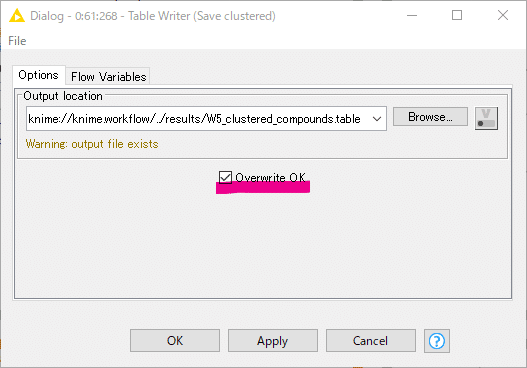
Overwrite OKにしてあるので、実行するたびに上書き更新されてしまうのは要注意です。
結果:
W5_clustered_compounds.table
というtable型のファイルが生成します。
【Table Reader】
Step1の下部を見ると、実行結果がtable形式で別途保存されています。
計算をスキップしてStep2以降を見たいならworkflowを繋ぎかえて使ってくださいとの親切設計です。
Step1は以上です。お疲れさまでした。Step2へ進めます。
記事を読んでいただきありがとうございます。 先人の智慧をお借りしつつ、みなさんに役立つ情報が届けられたらと願っています。 もしサポートいただけるなら、そのお金はKNIMEの無料勉強会の開催資金に充てようと思います。
