AlphaFold2の非専門家向け活用法 第4回前半「AlphaFoldが現状カバーできていない構造情報」
AlphaFold2による予測構造の良し悪しの判断についての第1回および第1.5回記事、予測構造に基づき実践的にメカニズムをいろいろ考察してみた第2回記事、タンパク質発現や構造解析へのAlphaFold2の活用についての第3回記事、などなどをこれまで述べてきました
これまでの記事では主に「AlphaFold2の有用性」に基づき、それを研究にどう活かすかという話でした。それらとは逆に、今回の第4回記事(おそらく最終回)では「AlphaFold2が現状カバーしていないこと、苦手なこと」に基づき、研究をどう進めていくかということについて前後編で二回に分けて述べてみたいと思います。
AlphaFold2がカバーできていないもしくは苦手な構造情報
1. 各種リガンド、イオン、薬剤等との結合認識
2. コンフォメーション変化
3. 複合体(当たり外れが多め)
あたりが現状のAlphaFoldが直接的にはカバーできていない、もしくはあまりカバーできていない構造情報でしょうか。
また、創薬研究者の「創薬ちゃん」さんは、これらに加え「点変異の影響の予測」「翻訳後修飾」等をあげています。
今後のハードルとしては
— 叢雲くすり(創薬ちゃん) (@souyakuchan) July 22, 2021
コンフォメーションの制御
<< 点変異の影響の予測、
単体では構造が完成しない分子の予測
<<< 翻訳後修飾や非典型アミノ酸の取り扱い
<<<<< 任意の化合物との相互作用予測
まあまだ敷居が高いことは色々あるだろう。#AlphaFold #AlphaFold2
ただ、点変異に関しては、安定性の評価にAlphaFoldが使えるのではないかという話もあり、この点についての続報が気になるところです。
One final alphafold thing :) - @Ally_Dunham compared deep mutational scanning data with predicted changes in stability for mutations on the alphafold models (with FoldX). The correlations observed are typically as good or better than with experimentally derived structures pic.twitter.com/qcDmCMALaV
— Pedro Beltrao (@pedrobeltrao) July 26, 2021
AlphaFoldが現状サポートしていない点については、AlphaFoldの公式サイトのFAQにも解説があります。ただ、「今のところサポートしていない」という言い方からも明らかのように「今後チャレンジしていくべき/現在進行形でチャレンジしている」ところでもありそうで、そちらも今後の進展に期待です。
それでは各トピックについて述べていきましょう。
1. 各種リガンド、イオン、薬剤等との結合認識
まず、リガンド等の認識についてですが、現状のAlphaFoldの予測構造にはリガンド等、タンパク以外の部分の構造は含まれていません。つまり、例えば「イオンチャネルがどのようにイオンを認識しているのか?」「受容体がそのリガンドをどのように認識しているのか?」というタンパク質の作動機構の中で極めて大切なところ、面白いところ、その要が含まれていないということです。第2回記事で紹介したNIPA1予測構造について、そのあたりの話をしなかったのもそういう事情があります。
では、「AlphaFoldの予測構造では基質認識はわからないのか?」というと必ずしもそうではありません。いくつか例をみていきましょう。
下図はヒト由来SLC41A1というマグネシウム輸送体の予測構造です。一部のN末端領域を除くと膜貫通ドメイン全域にわたって高い信頼度が確保されているのがわかります。

ヒト由来SLC41A1のAlphaFold予測
SLC41ファミリーの原核生物ホモログはMgtEといいまして、私が過去15年ほど取り組んでいるタンパク質です。そして、その結晶構造、特にマグネシウム結合状態の構造は以前から明らかになっています。ですので、既知構造の情報を元にSLC41A1のマグネシウムイオン認識について考察することは十分可能です。

MgtE実験構造におけるマグネシウムイオン結合部位の拡大図
上図は実験構造におけるマグネシウムイオン結合部位ですが、構造の類似性から対応するSLC41A1予測構造での相当部位は下図のようになります(MgtEプロトマーが5回の膜貫通へリックスを持ち機能的ダイマーを形成するのに対して、SLC41は単体で10回の膜貫通へリックスを持つ)。

SLC41A1予測構造でのMgtEマグネシウムイオン結合箇所の相当部位
また、これらを重ね合わせると下図のようになり、よく一致していることがわかります。
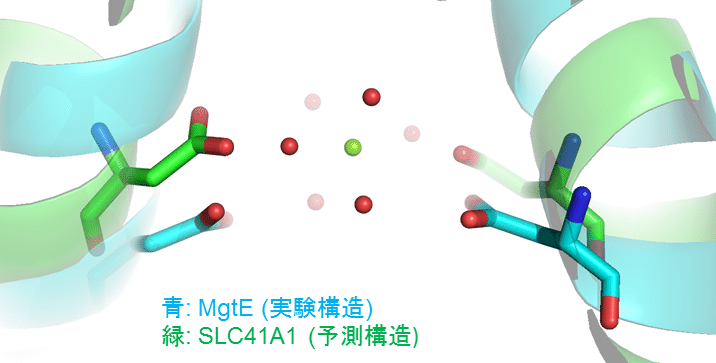
MgtE実験構造とSLC41A1予測構造を重ね合わせた上でのマグネシウムイオン結合部位の拡大図
以上、これらの比較から「SLC41A1ではAsp263とAsp487により水和状態のマグネシウムイオンが認識されおり、イオン透過に重要な役割を果たしている」と考察できると思われます。イオンチャネルにおけるイオン水和状態の認識の議論については、カリウムチャネルの構造解析が2003年のノーベル化学賞にもつながっており、長く続く非常に重要なトピックです。そして、この考察のさらなる検証には、例えば「Asp263とAsp487の変異体解析」「予測構造に対し当該の部位に水和状態のマグネシウムイオンをドッキングしての分子動力学シミュレーション」などが考えらえれるでしょう。
さて、「ここまでわかるならSLC41A1の実験構造の解析をする必要はもうないのではないか?」という声もあるかもしれません。ですが、「MgtEがマグネシウムイオンを受動輸送するイオンチャネルであるのに対し、SLC41A1はナトリウムイオンを輸送駆動力とするトランスポーター」であると考えられているのですが、今回の予測構造を眺めてみた限りでは、ナトリウムイオンの結合部位について推測するのは難しそうな感じでした。
また、薬剤等の化合物との結合予測についても、同様にAlphaFoldはサポートしておらず、現状では技術的なハードルも高いようです。タンパク質と薬剤もしくはその候補化合物の複合体構造情報というのは、創薬において非常に重要な情報です。
2. コンフォメーション変化
リガンド認識と同様に現状のAlphaFoldがサポートしていないのが、タンパク質のダイナミクス、コンフォメーション変化です。つまり、イオンチャネルの開閉におけるダイナミクス等の情報を直接得ることはできません。
AlphaFoldの特徴として、類似の実験構造があるようなターゲットについては、予測構造にリガンド等が含まれていないにも関わらず、「リガンド結合型の実験構造に引き寄せられたコンフォメーションの予測構造になりがち」のようです。AlphaFold2のWebsiteでもその旨、注意書きがあります。実際、SLC41A1の予測構造にマグネシウムイオンが含まれていないにも関わらず、マグネシウムイオン結合型のMgtEの実験構造によく似た構造となっていたのは既に示した通りです。それを含めて、その他の例をみていきましょう。
下図は、ATPをリガンドとして活性化されるリガンド依存性イオンチャネルであるP2X受容体、特にそのヒト由来P2X4受容体のAlphaFold予測構造です。P2X4受容体は神経障害性疼痛等に関与しており、創薬ターゲットとしても注目されています。SLC41/MgtEに続き、手前味噌ではありますが、こちらも私が過去10年ほど取り組んでいるタンパク質で、解説がしやすいということもあり、P2Xを例に話を進めたいと思います。ちなみに、下図では、P2X受容体は3量体を形成するため、GoogleColab版AlphaFold2を使って3量体の予測構造を得ています。
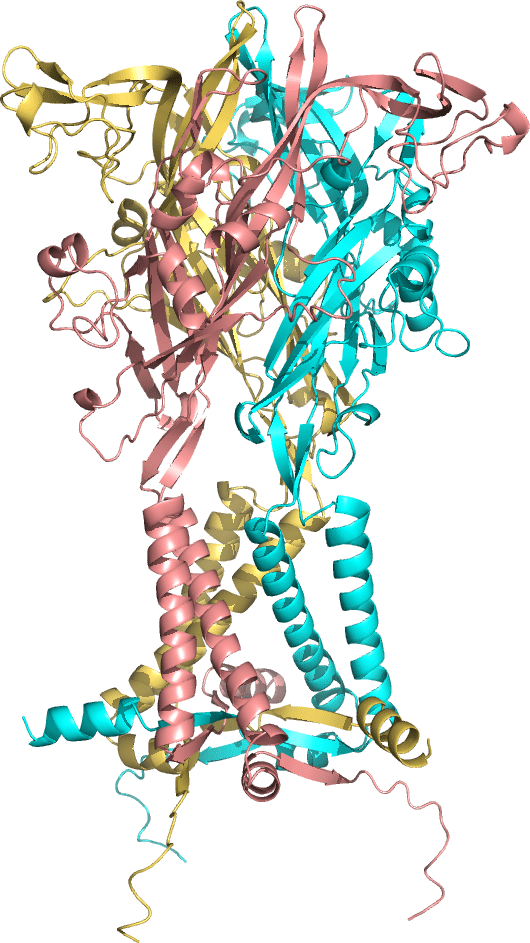
ヒト由来P2X4受容体ホモ3量体のAlhaFold2予測構造
P2X受容体については、7つあるサブタイプのうち、P2X3、P2X7、P2X7等いくつかのサブタイプについて様々なコンフォメーションの実験構造(Xray/Cryo-EM)が既に報告されています。そのため、予測構造の信頼性についても高いものが得られています。
ただし、予測構造には、「リガンドであるATPは含まれていない」「チャネルの開閉におけるどの機能状態(コンフォメーション)に対応する構造なのか不明」という問題があります。これらについて検討するために、既に得られているP2X受容体の実験構造と重ね合わせた結果が、下図です。用いたのはゼブラフィッシュ由来P2X4受容体のアポ型(ATP非結合型)とATP結合型の結晶構造になります。

ヒトP2X4受容体のAlphaFold予測構造とゼブラフィッシュP2X4受容体実験構造の重ね合わせ図
AlphaFoldの仕様上、ATP結合型の結晶構造に近い予測構造が出てくると予想していたのですが、重ね合わせの結果を見る限り、興味深いことにATP結合型ではなくアポ型の結晶構造に近い予測結果となっていました。
AlphaFoldの予測構造そのものは、コンフォメーション変化についての情報を与えてくれませんが、この考察(ヒト由来P2X4受容体の予測構造はアポ型におそらく相当する)に基づき、既知の類似実験構造のATP結合型、アポ型との比較に基づき、ヒト由来P2X4受容体のチャネル開閉について研究(変異体解析、MDシミュレーション)等を行うことは十分可能でしょう。
気になる点としては、AlphaFoldの予測構造では、リガンド結合型のコンフォメーションに近い予測構造が出やすい傾向という一方、、やはりケースバイケースのようです。この点も予測構造を利用する場合に注意が必要な点でしょう。例えば、化合物のバーチャルライブラリに対してこの予測構造を用いてin silico screeningをする場合、アポ型の不活性化状態に対応する予測構造が出発点となるため、たとえばアンタゴニスト(阻害剤)をスクリーニングしたい場合には向いているかもしれませんが、アゴニストをスクリーニングしたい場合にはおそらく理想的ではないのではないかと推測します。
まとめ
1. AlphaFold2の予測構造には、タンパク部分以外の結合因子(各種リガンド、イオン、化合物)は含まれていない。そのため、既知の類似実験構造を参考にそれらの結合部位や結合様式を参考に考察することは有用だが、その有用性と限界を念頭におくことが重要である
2. AlphaFold2の予測構造には、タンパク部分以外の結合因子は含まれていないが、得られるコンフォメーションは結合状態に相当することが多い(例: SLC41A1)。が、それもケースバイケースであり、リガンド非結合状態に近いものが得られることもある(例:P2X4受容体)。そのため、コンフォメーション状態の解釈についても既知の類似実験構造を参考にすべし。
3. 逆に言えば、1,2ともにリガンド結合やコンフォメーションの解釈の参考に、既知の実験構造を用いているため、それらの情報がないケース(類似構造がない場合や化合物との結合様式等)では、それらの情報考察は途端にかなり難しくなる。
といった感じでしょうか。後編では、残る「複合体構造予測」について主に述べるとともに、これまで述べてきたAlphaFoldが現在カバーしている領域/カバーしていない領域の比較から自ずとみえてくるであろう「After AlphaFoldの構造生物学研究での進め方の個人的所見」も述べてみたいと思います。
この記事が気に入ったらサポートをしてみませんか?
