AlphaFold2の非専門家向け活用法 第1.5回「予測の良し悪しの判断(補足と情報更新)」
前回の第1回記事の公開後、友人からいくつか追記点、修正点についてアドバイスをいくつか受けまして、その点の補足と、AlphaFold2の使い方のうち「3. EMBL版のAlphaFoldデータベースを使う(一番簡単)」に加えて、「2. Google Colab版のAlphaFold2を使う(配列をコピペ入力するだけ)」についての説明を第1.5回記事として追記します(Confidence score (pLDDT)表示のためのPyMoLプラグインの項は完全に勘違いしていたので削除。失礼しました…)。
ドメイン間の関係についての予測指標「Predicted aligned error」について
Confidence score (pLDDT)が重要という話を前回記事に書きましたが、EMBL版AlphaFold構造のページにも出ているもう一つのメジャーな指標の「Predicted aligned error」については完全スルーでした。こちらについて簡単に補足します。これは複数ドメインから構成されるタンパク質構造について、ドメイン間の関係の予測の確からしさの参考になる話です。
下記は、EMBL版AlphaFold予測構造サイトにおいて、「Predicted aligned error」のInstructionの例に出てくるBifunctional UDP-N-acetylglucosamine 2-epimerase/N-acetylmannosamine kinaseの予測構造を例に説明します。

このタンパクは主に二つのドメインから構成されており、いずれのドメイン構造のConfidence scoreは全領域で「Very High」となっており、信頼してよさそうです。
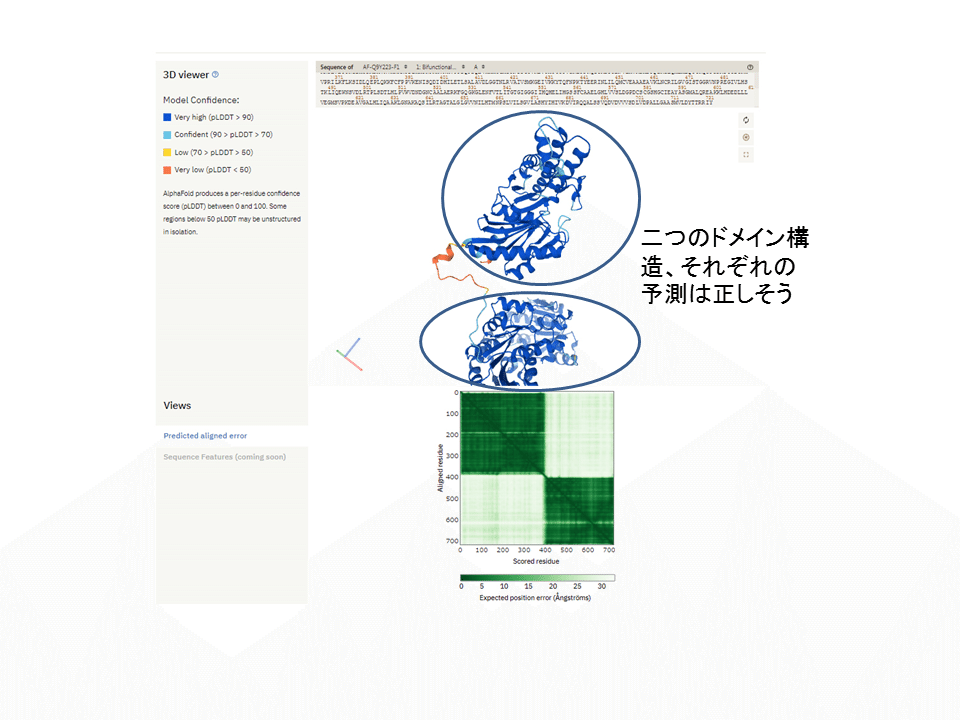
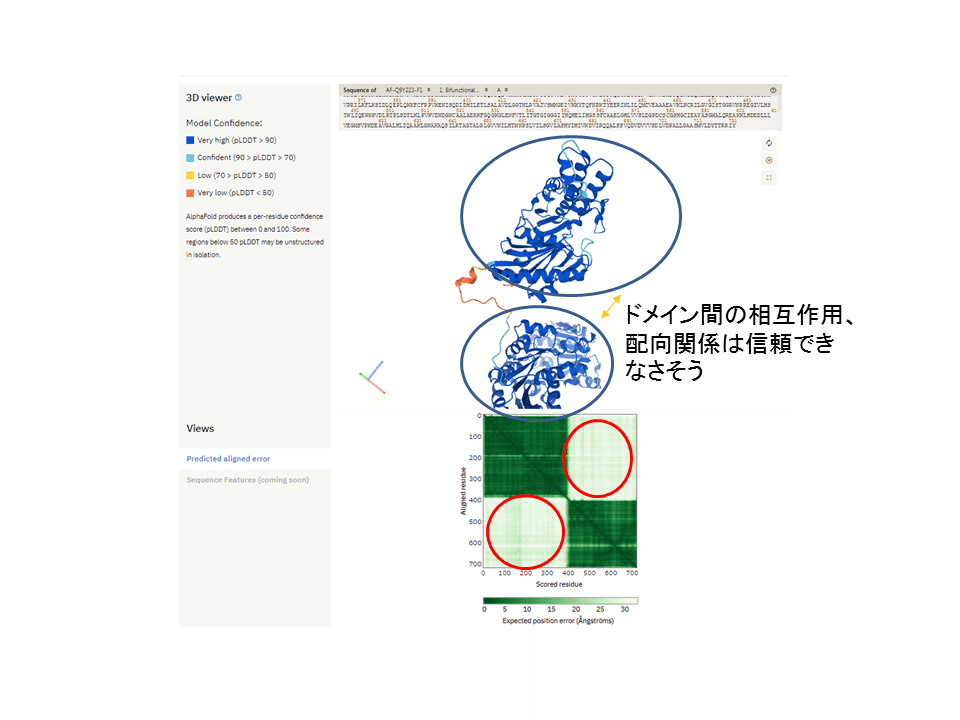
その一方、その下に表示されている「Predicted aligned error」については、信頼性が高い領域(深緑)と信頼性が低い領域(白っぽい)にはっきりとわかれています。「Predicted aligned error」は、おおざっぱにいって各残基"間"の関係についての予測の信頼性を示すものといえますが、Confidence scoreの結果と一致する形で、各ドメイン内では概ね信頼できそうな結果(深緑)となっています。

それに対して、各ドメイン間の「Predicted aligned error」は非常に低いスコアとなっています(白っぽい)。つまり、この予測構造を使う際、個々のドメイン構造それぞれの情報は参考しても大丈夫であろうが、AlphaFoldからの予測構造から得られる情報のうち、ドメイン間の相互作用領域、配向等についての予測情報についてはあまり参考にしないほうが良いということになるでしょう。
Google Colab版のAlphaFold2の紹介
最後に、任意の配列を投げることで得られるGoogle Colab版のAlphaFold2の紹介をしておきます。前回記事執筆後に各種情報のアップデートがあり、当初よりさらに有用になっているようです。
Protein structure prediction with AlphaFold2 and MMseqs2
リリース直後に各種アップデートがあり、使う上で重要な点として、ホモオリゴマー予測に対応するようになりました。EMBLデータベースに登録されているものはオリゴマーではなくモノマー構造なので、機能的に多量体を形成するタンパクについてはこちらがお勧めです。
ヘテロ複合体予測版。どうも開発者側も予測していなかった状況のようですが、なぜかヘテロ複合体の予測に有用なケースもかなりあるようで、ヘテロ複合体構造予測対応バージョンも出ています。謝辞にAg_smithさんの名前も。
以上になります。近日中に当初の第二回記事として「予測構造をみて、自分の研究に役立てたいけど、どうしたらいいの?」という話を構造生物学を専門としない生命科学者向けに述べてみたいと思います。
この記事が気に入ったらサポートをしてみませんか?
