タンパク質立体構造予測プログラムAlphaFold2の非専門家向け活用法 第1回「予測の良し悪しの判断」
構造生物学界隈のみならず、生命科学研究者やAI研究者の界隈すら超え、一般のニュースにもなっているタンパク質立体構造予測プログラム「AlphaFold2」について、構造生物学を専門としない生命科学研究者を主な対象として、note記事を3回くらいに分けて書いてみたいと思います。
生体高分子の立体構造データベース「Protein Data Bank」に登録されている実験構造は今や18万を超えています(私が15年くらい前にこの分野の研究を始めた際は2万弱でした)。にも関わらず近年急速に増えるゲノム情報に対しては、実験構造の数は圧倒的に不足しているのが実情だと思います。そのような状況の中、AlphaFold2はこの分野における「福音」だと私は感じてます。実際、構造生物学者の私自身も、ここ数日AlphaFold2で遊んでいますが、今までの構造予測と比べて非常にレベルが高く、研究のアイディアがバンバン浮かんでくるので、遊んでいて非常に楽しいです。
Highly accurate protein structure prediction with AlphaFold (Nature, Published: 15 July 2021)
「6年解けなかった構造があっさり」──タンパク質の“形”を予測する「AlphaFold2」の衝撃 GitHubで公開、誰でも利用可能に (ITmedia News)
わたしの記事の主眼としては、「AlphaFold2ってのがスゴイらしいけど、自分の扱っているタンパク質の予測構造をみてみたい。でも、構造生物学全然ワカラン」「予測構造に正しくなさそうなところがあっても自分では判断が付かない」「予測構造をみて、自分の研究に役立てたいけど、どうしたらいいの?」みたいなノリの、構造生物学を専門としない生命科学研究者に役立ててもらえばと思っています。
その理由としては、「 AlphaFold2」以後の時代においては、構造生物学の研究における「構造決定をする」「立体構造を元にBiological insightsを得る」のうち、いわゆる構造生物学研究者ではない、生命科学の広い分野の研究者にとって、後者の能力が今までより俄然必要になってくるのではないかと思うからです。
つまり、DeepLといったようなAIベースの翻訳ツールを使う際に「翻訳が間違っているとわかるだけの英語力」が必要なのと同様に、AlphaFold2を適切に使うには、構造生物学の基本的な素養を身に着けている必要があるであろうということです。
また、構造生物学、特に私のような構造解析を主な専門とする研究者がAlphaFold2をどう役立てることができるのかについても、第三回目あたりに書いてみる予定です。私自身はAIについて全く詳しくありませんが、今受けている印象としては、AlphaFold2(や今後出てくるその派生形)と構造生物学者の関係は、AIベースの将棋ソフトと将棋棋士のような関係になってくるのではないかと感じています。つまり、現在の将棋棋士にとって将棋ソフトの活用が重要なのと同じで、構造生物学者にとっても構造予測の活用が必須となってくると思われます。
AlphaFold2による予測構造を得る方法
現状大まかに分けて、3つの方法があります。
1. 自分でインストールして使う (上級者向け)
2. Google Colab版のAlphaFold2を使う(配列をコピペ入力するだけ)
3. EMBL版のAlphaFoldデータベースを使う(一番簡単)
1.はほとんどの人にとって敷居が高いと思います。私も計算リソースの関係で試していません。日本語では、@Ag_smithさんによるインストールマニュアルがqiitaにでています。2.についても同じqitta記事に説明があります。こちらはかなり簡単で、私がここ数日遊んでいたのはこのGoogle Colab版です。
AlphaFold (ver.2) インストール by Ag_smith
3. のEMBL版のAlphaFoldデータベースはちょうど今日発表されたものですが、UniProtと連動しており、アミノ酸配列を探す感覚で高精度の予測構造を得ることができます。現状、メジャーなモデル生物のゲノム中のタンパク質構造のみのカバーに限られてはいますが、多くの生命科学研究者の需要を既に満たせているのではないかと思います。また、今後さらに拡張予定とのことです。
AlphaFold Protein Structure Database, Developed by DeepMind and EMBL-EBI
このnote記事では3. EMBL版のAlphaFoldデータベースについて主に紹介したいと思います。
予測構造の良し悪しをどう判断する?
さて、上記のAlphaFold Protein Structure Databaseから立体構造のpdbファイルをダウンロードしたとしましょう。1958年に最初のタンパク質の立体構造として解析されたのは、Max Perutz先生の重原子同形置換法を用いたJohn Kendrew 先生によるミオグロビンですが、下記はヒト由来のミオグロビンについてのAlphaFold Protein Structure Databaseからのスクリーンショットです。
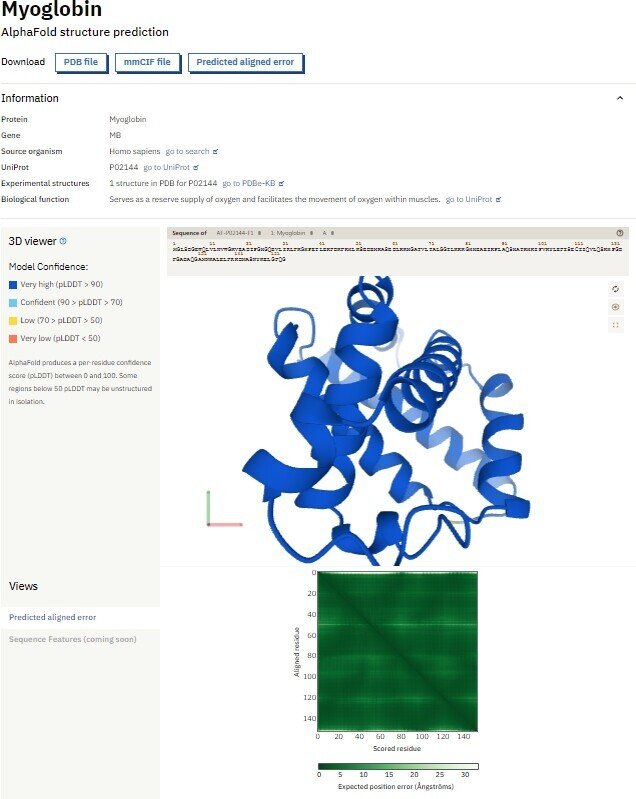
ダウンロードした立体構造のPDBファイルの分子ビューワープログラムによる見方については、下記のToGoTV(ライフサイエンス統合データベース)による分子ビューワーの使い方をご参照ください。
PDBjを使ってタンパク質の立体構造を調べる(TOGOTV)
まず、構造の良し悪しについて、真っ先にみるべきは、「Model Confidence」の項でしょう。全領域にわたって、真っ青(Very high)なものについては基本的に信頼できることが多いと考えてして良いでしょう。既に実験構造が存在するヒト由来ミオグロビンについては全域が「Very high」となっています。
その一方、Model Confidenceが「Low」や「Very Low」の領域ついては、立体構造の解釈に用いるのはあまりオススメできません。「Confident」の領域については正直ケースバイケースですが、多数のVery highに隣接して一部存在しているような「Confident」についてはかなり正しい印象があるものの、「Confident」領域から主に構成されているようなケースでは要注意です。
うまくいっている予測構造例(とその注意点)
上記のような説明では「よくわからん」という方も多いと思いますので、予測がうまくいっていそうなケース、うまくいってなさそうなケースについて個別の事例紹介をすることで、「なるほど」と思っていただけたらと思います。
下記は、2012年のノーベル化学賞にもつながったGPCRの最初の立体構造のうち、そのbeta2アドレナリン受容体のAlfaFold2による予測構造(2021年7月23日リンク先訂正。本文や下図はbeta2なのにリンクとして別のを間違えて貼ってました)です。
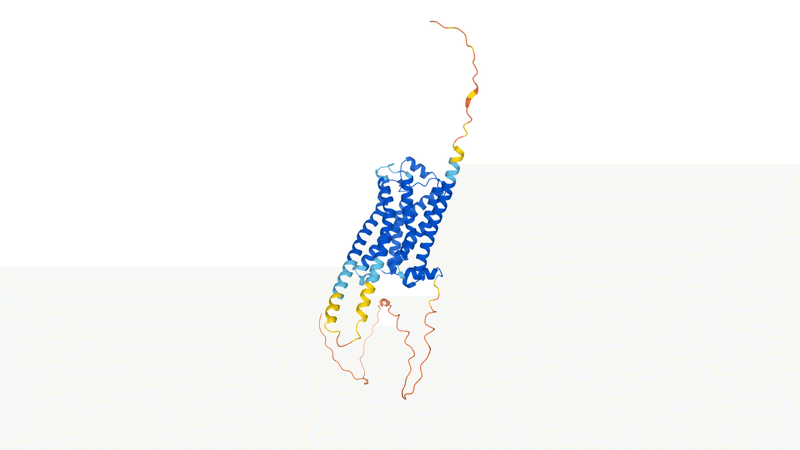
この構造では、GPCRのコアとなる7回の膜貫通へリックス領域において概ね「Model Confidence」の項が真っ青(Very high)になっています。これらは構造の解釈の際、概ね信頼して使ってよい区域といえるでしょう。
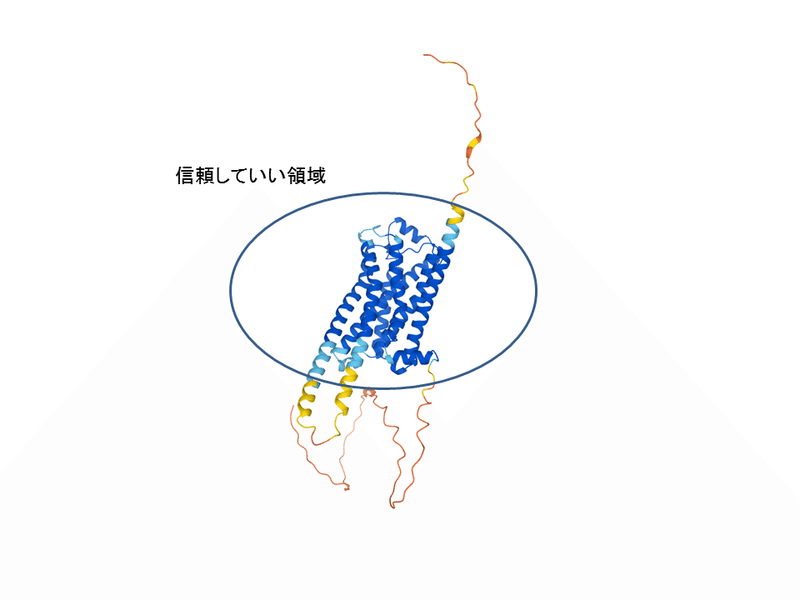
それに対して、N末端側や細胞質側ループの一部(通称: ICL3)は、「Model Confidence」が低くなっています。また、二次構造とほとんど形成しておらず、「ぶらぶらとしたループ領域」となっていることがパッと見でも感じられます。このような領域の現状の予測構造については、構造の解釈の際、重視すべきではないと思います。
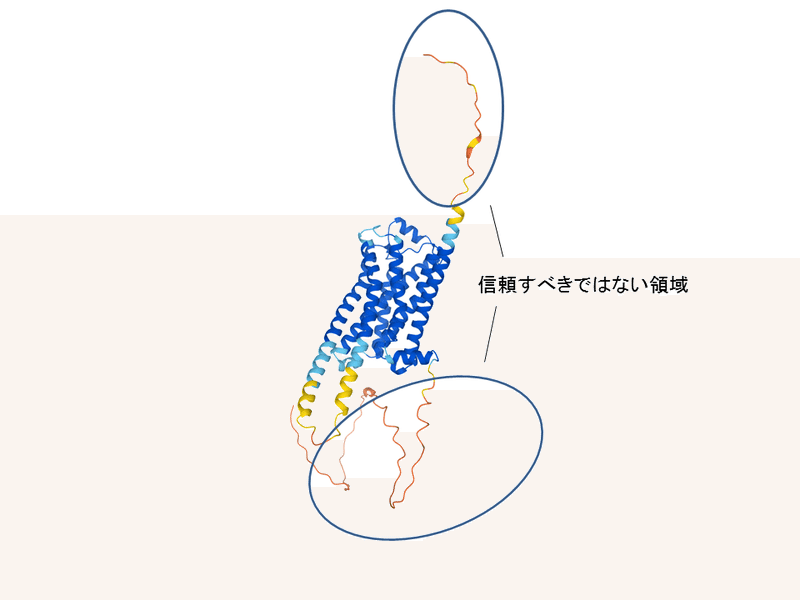
実際、これらの領域は、実験的なX線結晶構造決定の際は、disorder(立体構造がオーダーしていない)領域として、結晶化の際のタンパク質発現コンストラクトからは除かれる等しています。
ちなみに、このbeta2アドレナリン受容体のように実験構造があるケースにおいては、実験構造のほうを参考にされることをまずオススメします。理由としては、現状のAlphaFold2の場合、「リガンド等の基質は予測構造には含まれていない」「オリゴマー状態を反映せず、モノマー状態の予測構造」「コンフォメーション(構造上の機能状態)が複数の状態が混じったような奇妙な構造になっていることもある」等の理由からです。
予測がうまくいってなさそうな例
さて、さきほどは基本的にうまくいっている例ですが、AlphaFold2にもまだ苦手と思われる対象はいくつかあるようです。特に「天然変性( Intrinsic Disordered」 な領域が多いタンパク質については、そもそもdisorderしている領域なため、一定の構造をアサインすることはできません。
Pack up your labs, SWI/SNF biologists. chromatin is solved. pic.twitter.com/uyENfQxtE1
— Emma J Chory (@chorye) July 22, 2021
i see the signal transduction field is kaput as well pic.twitter.com/soUVuimyyj
— Yaakov (@TheYCluster) July 23, 2021
また、私自身が専門とする膜タンパク質について、未公表構造をみてみましたが、AlphaFold2が非常にうまくいっている場合(実験構造に対しRMSDが0.4-0.6Å以内)のような予測精度はなく、2Å弱とあまり芳しいものではありませんでした(それでも従来の構造予測よりは良いのですが。)。
以上、従来法より大幅にレベルアップしたAlphaFold2の構造予測ではありますが、まだ得意不得意はあり、予測構造を使う上で「どこが信頼できるか」を念頭に置くのは大切かと思います。
それを踏まえた上で、次回記事では「予測構造をみて、自分の研究に役立てたいけど、どうしたらいいの?」という話について、構造生物学を専門としない生命科学者向けに述べてみたいと思います。
この記事が気に入ったらサポートをしてみませんか?
