
データサイエンスによる日韓のコロナ対策比較
ベイジアンモデリングという手法を使って日本と韓国のコロナ対策の有効性を考えます。
SIRDモデル
まず背景にある数学的モデルを説明します。理論的な話に興味のない方は飛ばしてください。
ベースになっているのはよく知られているSIRDモデル(susceptible, infected, recovered and death model)です。これはこのように考えます。S[t]を時点tで未感染の人の数、I[t]を今感染している人の数(アクティブ)、R[t]を回復した人、D[t]を死亡した人の数とします。感染している人は各時点である確率βで未感染の人に病気をうつします。今感染している人はある確率aで回復し、確率dで死亡します。一旦回復すると免疫がついてもう病気にはかからなくなります。これを式にまとめてみましょう。Pは人口です。

新型コロナウイルス の感染症で難しいのは、かなりの割合の人に症状がない、ということです。また、検査能力も限られているため、完全に感染者数を把握しているとは考えられません。また、感染を封じ込める一定の政策が取られるはずです。
そこでターチンのモデルにならって検出率qを導入します。C0(t)を確認された感染者の総数、R0(t)をそのうちで回復した人、D0(t)をなくなった人の数とします。すると

とかけます。ここでC0(t) - R0(t) - D0(t)は現在の感染者です。
また、βやqは時間と共に変化すると考えます。変化はsigmoid曲線に従うと仮定しいます。つまり、

検出率は最初は0とします。(しないモデルも考えられますが、うまくデータを説明できませんでした)
すこしこのモデルの理論的性質について触れておきます。このモデルはill-conditonedです。ill-conditionedとは入力のわずかな差で答えが大きく変わる問題のことです。今回のモデルの場合、qを具体的に定めることは容易ではありません。理由を考えるために、感染の初期で感染者が指数関数的に増加している状況を考えましょう。また、qは定数とします。すると、新たに検出される感染者の数はq * N * exp(β t)で近似できます。Nはt=0の感染者の数です。見ていただくとわかるように、qとNは個別に決定することができません。真の感染者数はN exp(β t)ですから真の感染者数を推定することもできないことになります。
私たちのモデルはqはt=0でq=0から出発してsigmoid曲線により増加すると仮定しています。よって、検出される感染者の数は指数関数から外れます。この差を使ってq1を計算することが可能になるかもしれません。実際今回使ったツールはq1等のパラメータを比較的確信をもって推定しています。ただ、具体的な数値よりも全体の傾向を掴む手段だと考えたほうが良さそうです。
パラメーターはベイズ推定により決定しました。ベイズ推定は推定値の誤差を求めることができ、また事前知識をモデルに組み込むことができます。
日本の結果
日本の分析結果を述べます。まずモデルが予想するアクティブケース(感染が確認された人でまだ回復していない人)の人数と実際の値をグラフにしてみます。

よく一致していることがわかります。推定された検出率の推移は次のようになります。
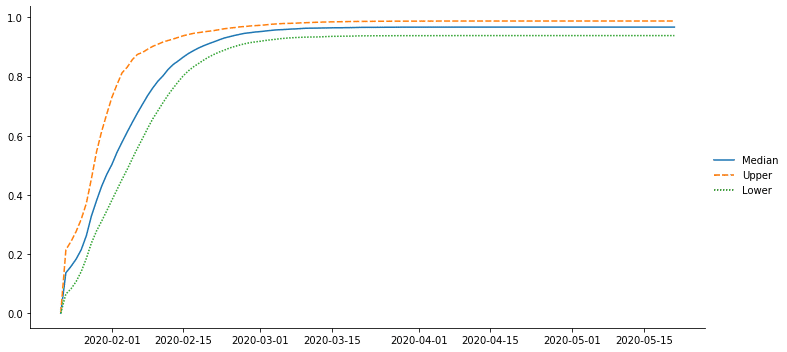
意外なことにかなり1に近い値を示しています。最後に増殖率βです。大きいほど感染拡大の速度が速いことを示しています。

4月の初めに急激に増加していることがわかります。詳しい人向けにツール(Stan)の出力を載せます。
Inference for Stan model: anon_model_7ba6d17563748454d8034e538402909e.
4 chains, each with iter=100000; warmup=50000; thin=1;
post-warmup draws per chain=50000, total post-warmup draws=200000.
mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
init_inf 21.37 0.02 3.22 15.91 19.04 21.1 23.41 28.36 43469 1.0
b0 0.08 1.1e-5 2.3e-3 0.08 0.08 0.08 0.08 0.08 42313 1.0
b1 0.13 5.1e-6 2.0e-3 0.13 0.13 0.13 0.13 0.13 151129 1.0
theta_b 5.8 3.7e-3 1.61 3.33 4.66 5.57 6.7 9.59 189395 1.0
b_date 70.6 3.2e-4 0.14 70.32 70.5 70.6 70.69 70.85 185513 1.0
q1 0.96 9.8e-5 0.04 0.85 0.94 0.97 0.99 1.0 175728 1.0
theta_q 0.45 4.0e-3 0.69 0.1 0.14 0.17 0.33 2.53 29798 1.0
q_date 8.42 0.02 3.88 1.65 5.22 8.2 11.45 15.84 30806 1.0
a 0.01 1.4e-6 4.8e-4 0.01 0.01 0.01 0.01 0.01 115203 1.0
d 1.9e-3 5.1e-7 1.8e-4 1.6e-3 1.8e-3 1.9e-3 2.0e-3 2.3e-3 126505 1.0
lp__ 3.1e4 0.01 2.49 3.1e4 3.1e4 3.1e4 3.1e4 3.1e4 46532 1.0
init_infは最初の感染者数、b0、b1は増殖率、b_dateは増殖率が変化した日、q1は最終的な検出率、aは回復率、dは死亡率です。b_dateから増殖率が急増したのが4月1日であることがわかります。
検出率がほぼ1に近づいている、増殖率が4月1日に急増しているなどやや不自然な点がありますが、Stanの診断出力は推定値がかなり信頼できるものであることを示しています。
韓国の結果
韓国に同じモデルを適用した結果を示します。まずモデルが予想するアクティブケース(感染が確認された人でまだ回復していない人)の人数と実際の値です。

ややずれがありますが、比較的よく一致していると言えるでしょう。検出率の推定値のグラフは次のとおりです。

ゆるやかに8割程度まで上昇していることがわかります。推定された増殖率は次のようになります。

Stanの出力結果は次のとおりです。
Inference for Stan model: anon_model_7ba6d17563748454d8034e538402909e.
4 chains, each with iter=100000; warmup=50000; thin=1;
post-warmup draws per chain=50000, total post-warmup draws=200000.
mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
init_inf 5.06 7.4e-3 2.19 1.74 3.47 4.75 6.32 10.19 86911 1.0
b0 0.29 4.6e-5 0.01 0.27 0.28 0.29 0.3 0.32 80552 1.0
b1 6.1e-3 4.0e-6 1.0e-3 4.3e-3 5.4e-3 6.0e-3 6.8e-3 8.3e-3 66270 1.0
theta_b 0.42 3.6e-5 0.01 0.4 0.42 0.42 0.43 0.45 100615 1.0
b_date 40.41 9.2e-4 0.26 39.9 40.24 40.42 40.59 40.92 81206 1.0
q1 0.84 3.5e-4 0.14 0.49 0.76 0.87 0.94 1.0 159922 1.0
theta_q 0.14 3.0e-5 9.1e-3 0.12 0.13 0.14 0.14 0.15 90374 1.0
q_date 47.63 6.3e-3 1.81 43.72 46.5 47.75 48.89 50.82 82683 1.0
a 0.03 7.9e-7 3.6e-4 0.03 0.03 0.03 0.03 0.03 204455 1.0
d 9.2e-4 1.4e-7 6.2e-5 8.0e-4 8.7e-4 9.2e-4 9.6e-4 1.0e-3 209800 1.0
lp__ 7.9e4 8.4e-3 2.31 7.9e4 7.9e4 7.9e4 7.9e4 7.9e4 75020 1.0
診断結果は推定がうまく行っていることを示しています。
日韓の比較
日本と韓国を比較すると、韓国はコロナの制圧に成功した一方、日本は失敗しつつあるように思われます。このモデルは予測を目指したものではありませんが、このままの状況が続くと爆発的に感染が増えることが予想されます。
なお、日韓それぞれのデータの挙動の具体的な理由については検討することができませんでした。また、ターチンが行った韓国の分析とは推定値の振る舞いがやや異なっています。ターチンは最小二乗法で推定を行っており推定手法が違う他、仮定している関数形も違うように見えます。今後引き続き検討したいと思います。
モデルおよび計算に用いたコードは公開しています。ご自由にお使いください。
利用したStanモデル: https://github.com/yoriyuki/COVID-19/blob/master/prediction/turzin.stan
計算のプロセス(日本) : https://github.com/yoriyuki/COVID-19/blob/master/prediction/Turzin.ipynb
計算のプロセス(韓国) : https://github.com/yoriyuki/COVID-19/blob/master/prediction/Turzin-Korea.ipynb
なおディレクトリ名にpredictionとありますがモデルは将来予測を想定して作られたものではありません。
この記事が気に入ったらサポートをしてみませんか?
