
ITK-SNAPを使った3D DICOMイメージのセグメンテーション(誰トク?)
誰に向かっての記事なのか分かりませんが、基本コンセプトである「自分のための健忘録」として、とても有名なフリーソフトであるITK-SNAPを使った腎臓のセグメンテーションのやり方を書いておきます。結構簡単にできるので、特定の医療研究に関係する人にはそれなりの需要はあると思うのですが…w。
0.ITK-SNAPのインストール
まずはこちらのページにアクセスしてください。右上にあるダウンロードのリンクをクリックして自分の環境・PCにあったものをダウンロードしてください。

ユーザー登録をしてください、というお願いがありますが、右下でスキップすることも可能です。
ダウンロードが開始されるとSOURCE FORGEのサイトに移動してマックの場合は.dmgファイルがダウンロードされます。
1.画像を読み込む
「File>Open Main Image…」から画像を読み込みます。フォルダ内の一つの画像しか選択できないのですが、フォルダ内の一つの画像を選んで「Open」ボタンを押せばフォルダ内のすべての画像が読み込まれます。


画像が読み込み終わると、以下の様に3断面の画像が表示されます。(初回は画像の並び順などの設定が聞かれますが、自分が慣れた表示方法にする世にしてください。)

2.画像の表示条件を変更する
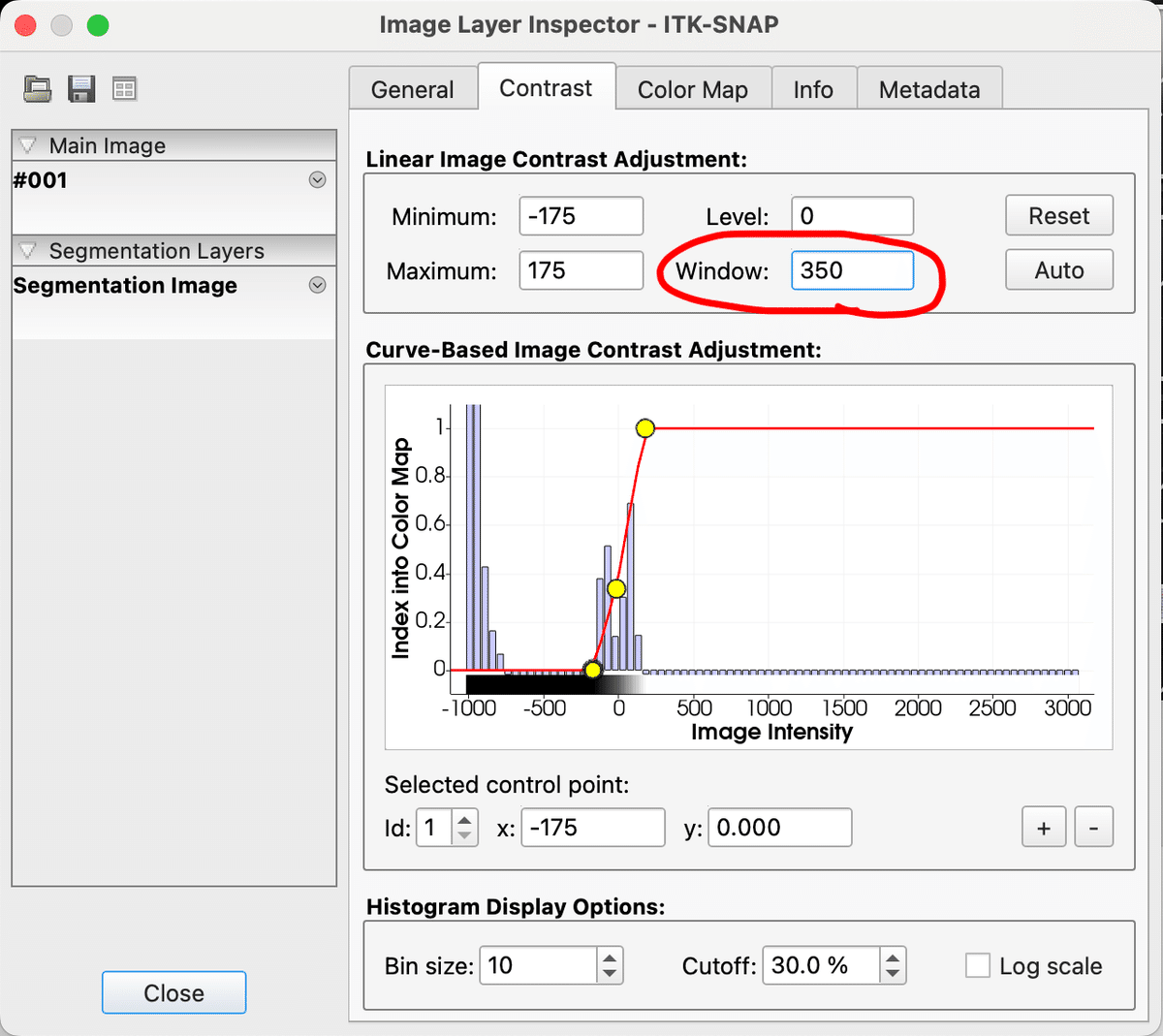
最初画像を読み込んでみたら、画面が真っ白だったり、おかしく表示されたりします。その場合は「Tools > Image Contrast > Contrast Adjustment…」を選んで、適切な表示条件にします。上記の画像はCT画像なのですが、CT画像は2000階調くらいのデータを持っていて、それを256階調のモニタに如何に表示するかを決めてあげます。

表示条件には、1)Window幅とレベル・2)上限と下限の2つの方法があり、CTやMRIでは幅とレベル、核医学では上限と下限を使う事が多いです。今回はCTの画像なのでWindow(幅)を350に変更してみます。
3.矩形で目的物を取り囲む
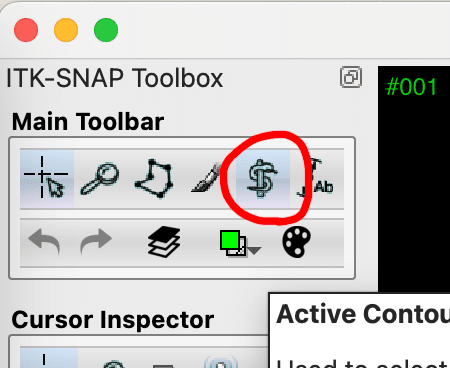
「自動セグメンテーション」は蛇とペンのアイコンを選ぶことから始まります。
今回は左の腎臓をセグメンテーションしてみます。まずは大まかに左の腎臓を点線の四角の枠で囲んでみます。最初は四角が最大になっているので、どこにあるか分かりにくいです。まずは下図の「Reset ROI」を押してあげると、右側の赤い点線の四角のようになります。
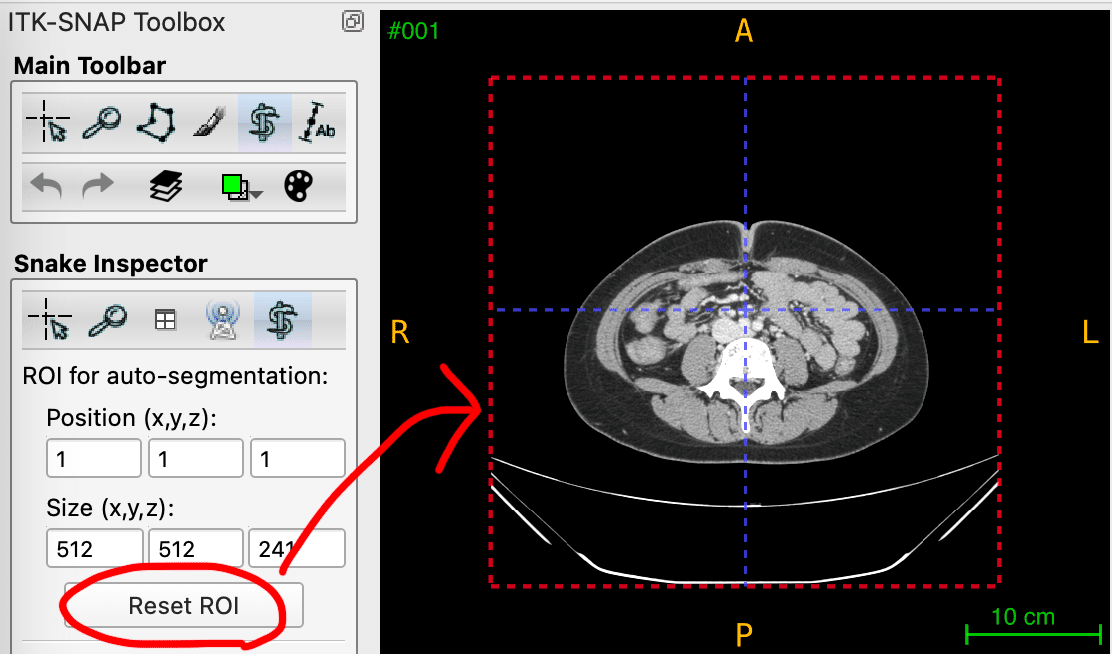
この四角をドラックして調整してあげます。3断面の画像のそれぞれで左の腎臓よりやや大きくなるようにしてみてください。
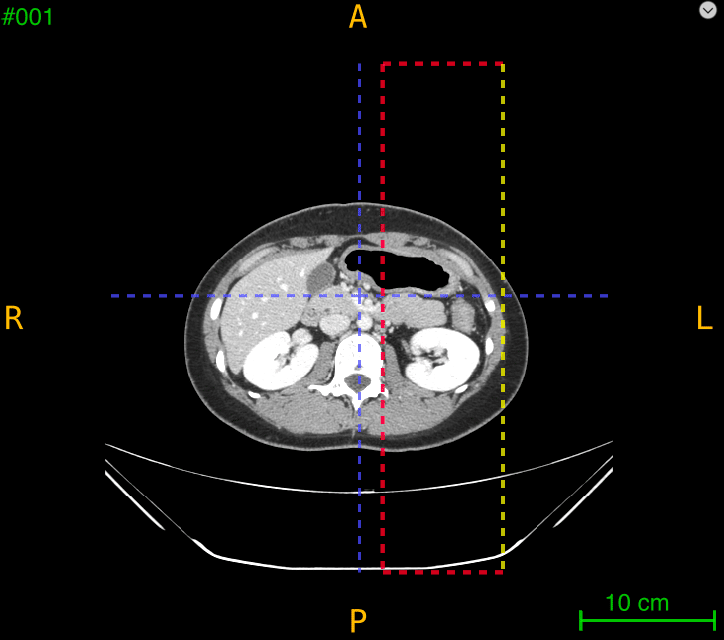
このような形で3断面で左の腎臓を囲んで見てください。(下の図はやや拡大して表示しています。)

4.(半)自動セグメンテーションのウィザード
「Segment 3D」というボタンを押してみてください。画像の右側に自動セグメンテーションのウィザード部品(縦長)が開きます。
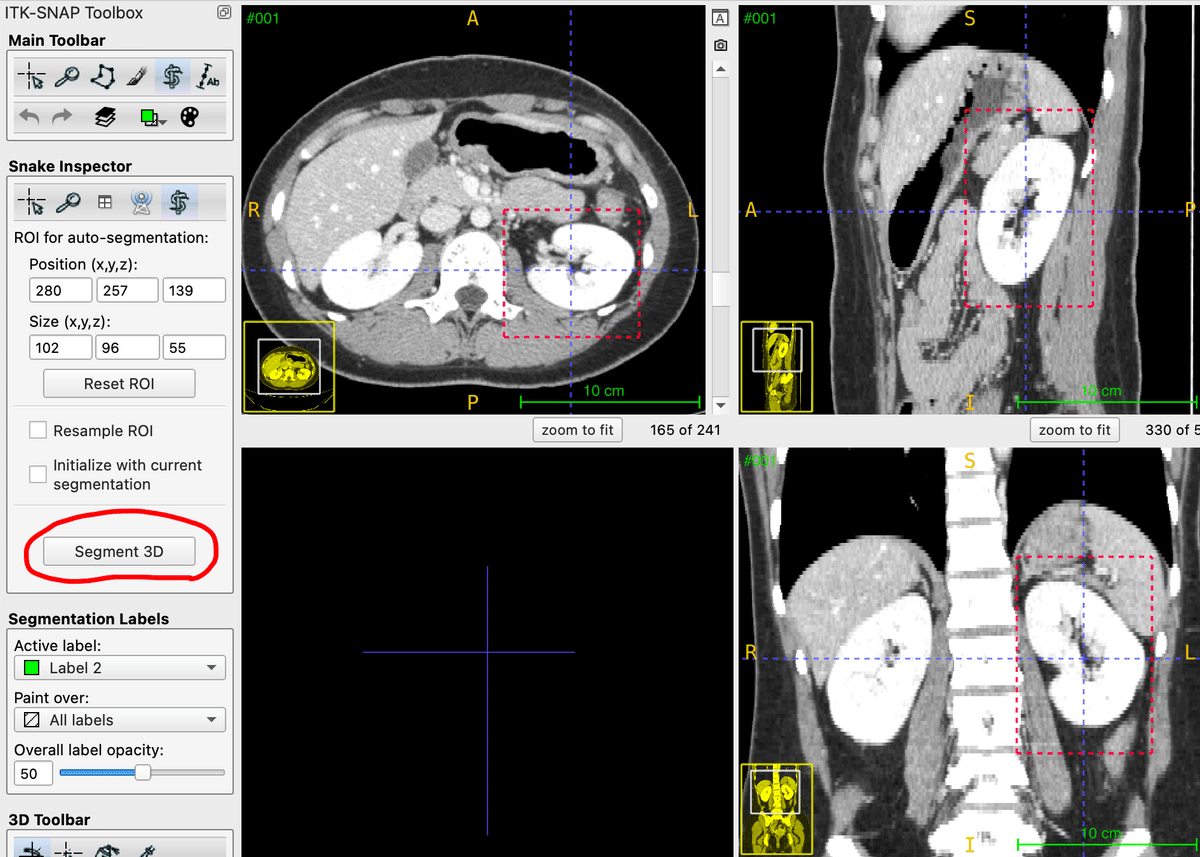
1) Step 1:閾値設定
Stepは1/3〜3/3までの3ステップになります。まずはthreshold(閾値)の設定になります。Threshold mode:というところでは3つのタイプがあります。その内容はアイコンの通りです。下の図では左のアイコンが選ばれていますが、これは上限と下限を設けて選択するというモードになります。
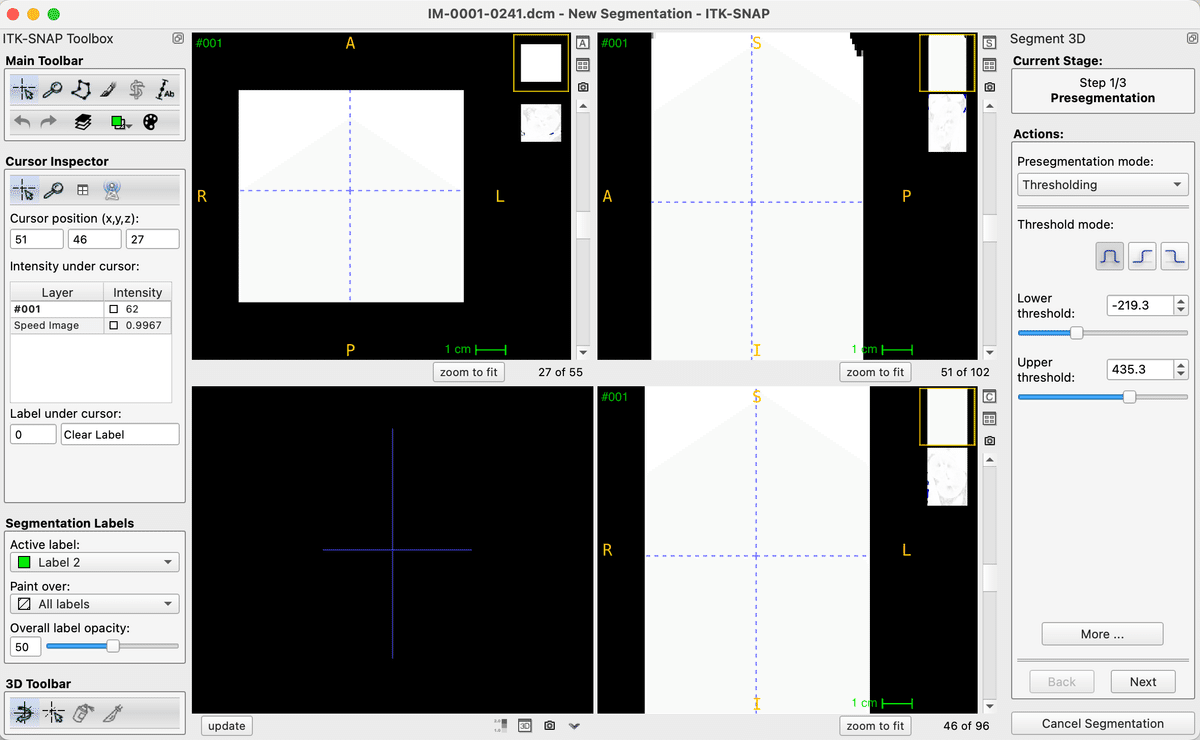
今回は下限のみを指定して腎臓の周りの脂肪が選択されないようにしました。※青で示されている部分は関心領域以外という意味になります。

閾値の設定が終わったら、右下にある「Next」のボタンを押してStep2/3に移動します。
2) Step 2: Seedを決める
この後、半自動で腎臓をセグメンテーションしていくのですが、その種(シード)となる点を加えていきます。(画面内の緑の丸)
画像の中で、腎臓の領域内に青の十字を移動させてから、「Add Bubble at Cursor」というボタンを押します。すると、青の十字を中心に緑の丸が塗りつぶされます。
まるが多い方が、ITK-SNAPが推測しやすくなるのだと思いますが、あまりSeedを増やさなくてもシンプルな構造であれば少なくても大丈夫です。
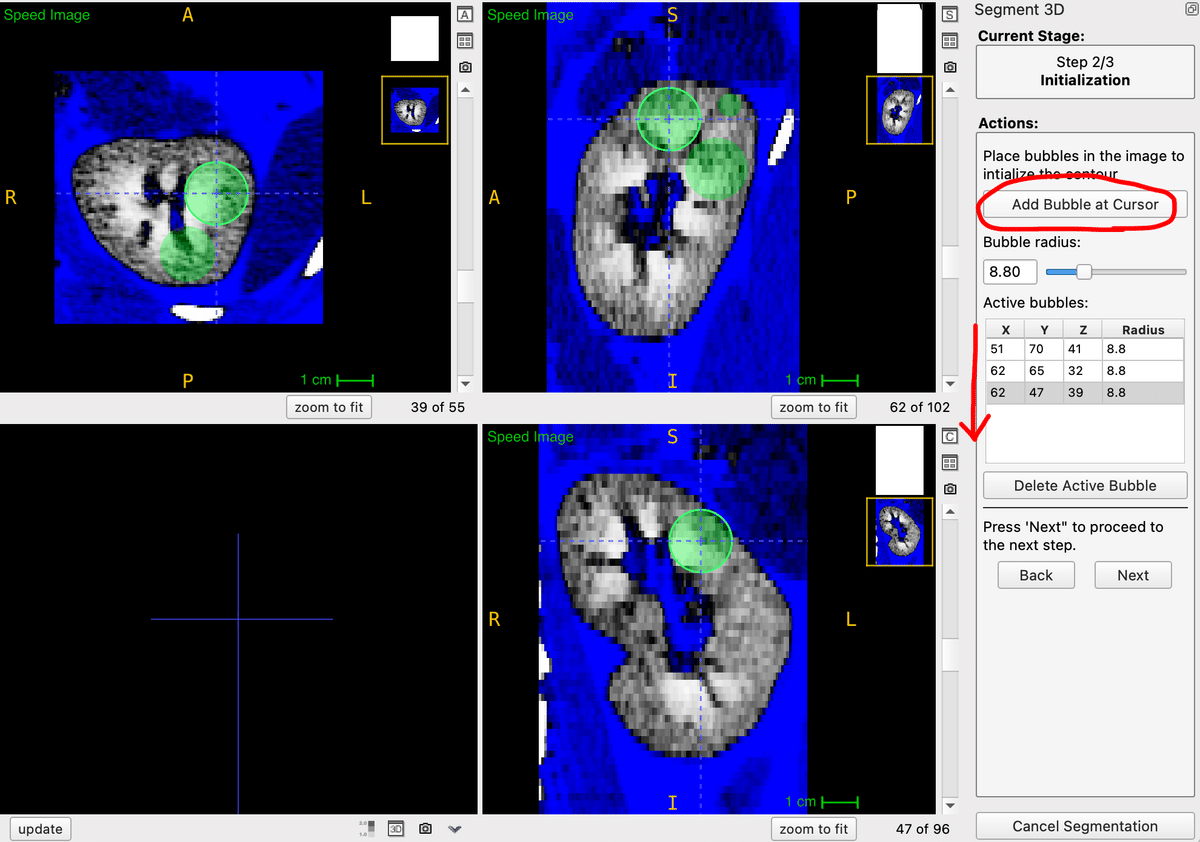
適当なSeedを加えたら、右下の「Next」ボタンを押してStep 3/3の最後のステップに移動します。
3)Step 3:イタレーション(逐次近似)によるセグメンテーション
Step 1で設定した閾値・Step 2で設定したSeedを元にITK-SNAPでは逐次近似的に腎臓の領域をセグメンテーションします。
「▶」のボタンを押すことで、逐次近似が開始されます。逐次近似が進むとIteration(逐次近似)回数がどんどん上昇していき、それと共に赤で塗りつぶされる領域が腎臓全体に広がっていきます。
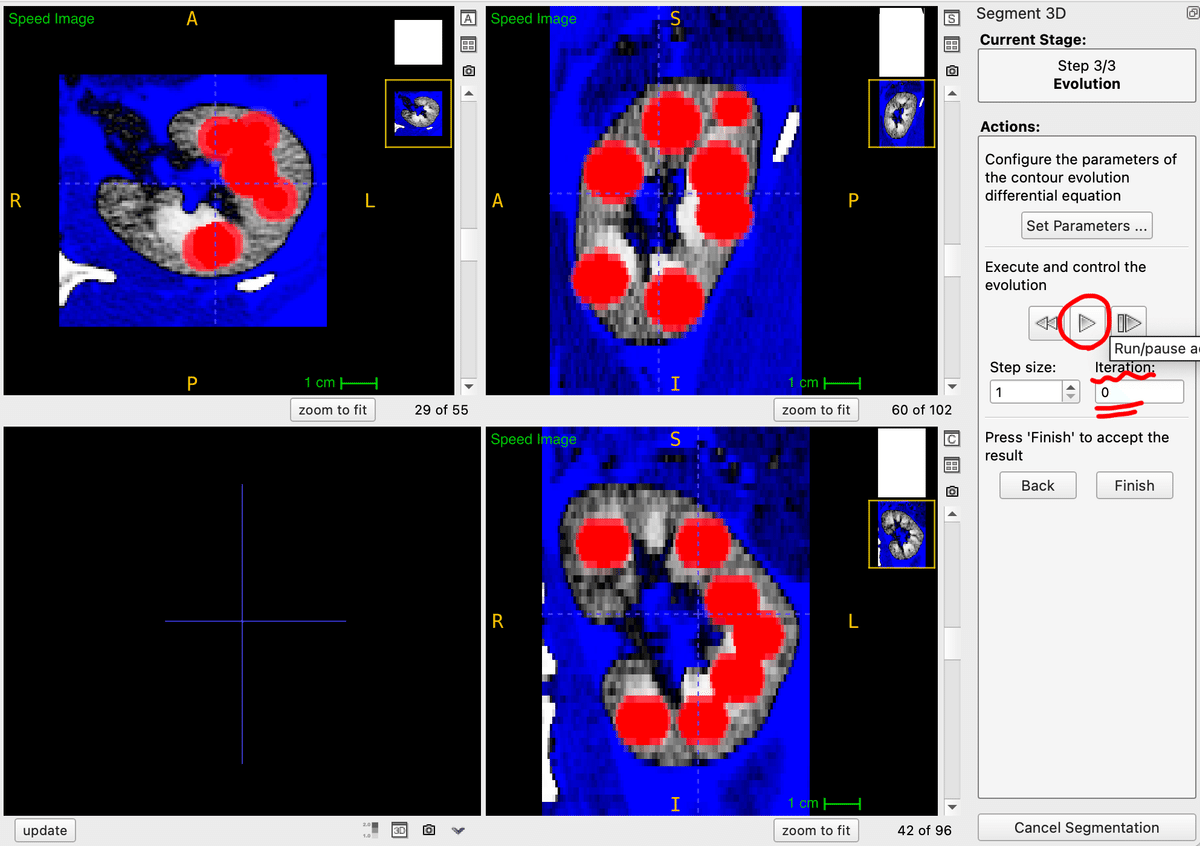
良いなと思ったところで、「▶」のボタンを再度押して、一時停止します。
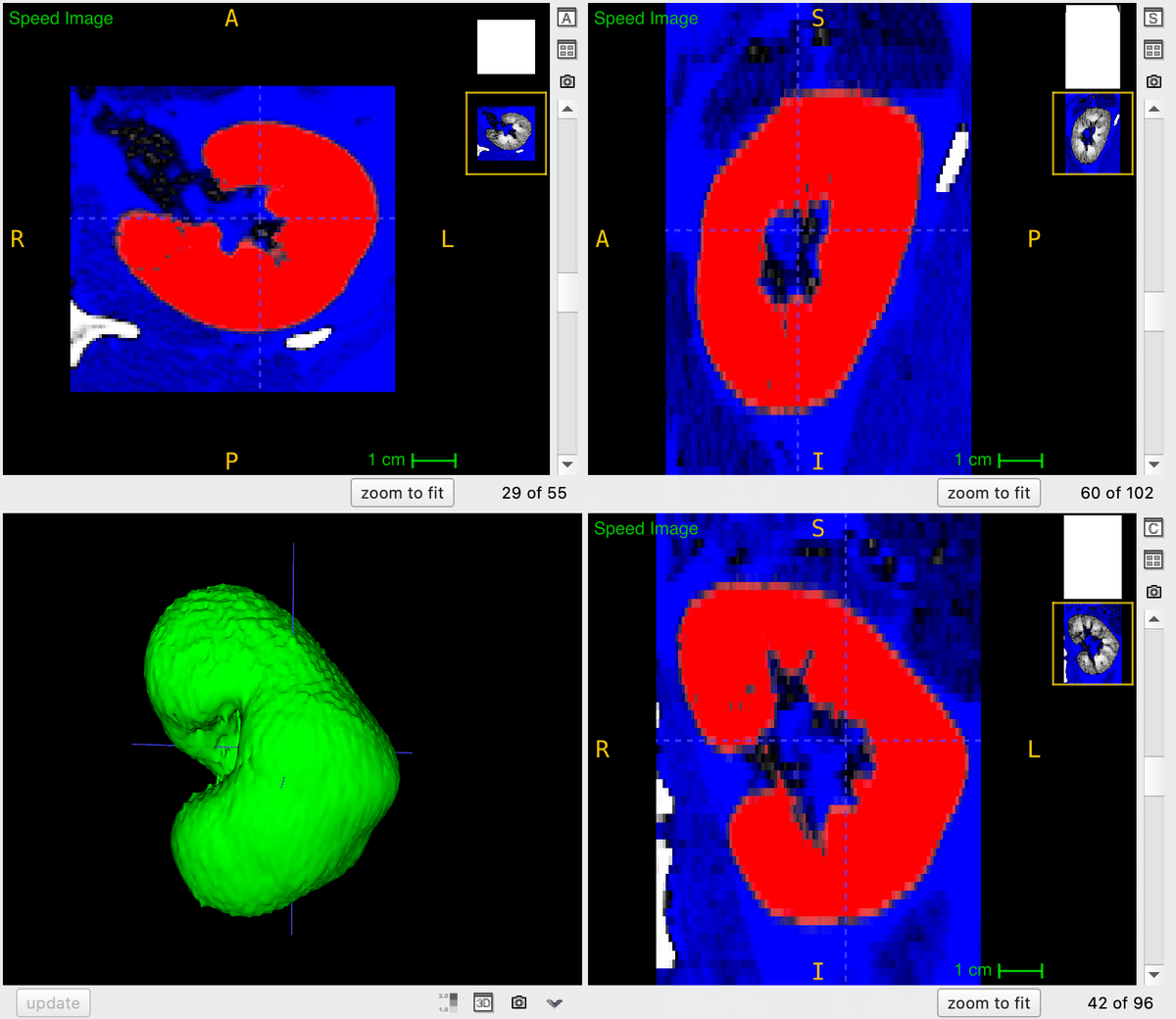
その後に左下にある「update」のボタンを押すと左下のウインドウに腎臓の3D画像が表示されます!

updateを押すと、以下の様に腎臓が緑色で3次元表示されます。
まとめ
いかがだったでしょうか?
けっこう、簡単に腎臓の3次元データを取り出すことができました。
一度、この記事にあるようなことをやっておくと、自分で癌をセグメンテーションしたい時に役立つと思います。
機械学習などの際には、労力の9割以上が教師用のラベル作成ですので、如何に簡単にセグメンテーションできるかが、研究が遂行できるかのカギになります。
自分的にはITK-SNAPのこのような使い方を教えてもらえるとうれしいのですが、皆さんいかがでしょうか?笑
この記事が気に入ったらサポートをしてみませんか?
