
グラフ作成練習: コロナ感染者数推移
#pandas #seaborn #matplotlib #python #Jupyter_Notebook #グラフ #コロナ
*あくまで練習の題材です。科学的な何かを導き出そうとしているわけではないです。データの正確性の保証などもできません。
データ
厚生労働省と東京都がオープンデータとして公開してた!いい時代になったなぁ(;ω;)感動した・・
読み込み
read_csvにリンクを渡せば良い。utf-8だったりsjisだったりすることに注意。日によって変わっている気がするのだが気の所為か…?
In : import pandas as pd
import seaborn as sns
import numpy as np
import matplotlib.pyplot as plt
In : pcr_positive_daily: pd.DataFrame = pd.read_csv("https://www.mhlw.go.jp/content/pcr_positive_daily.csv", encoding='shift_jis')
pcr_positive_daily["日付"] = pcr_positive_daily["日付"].astype(np.datetime64)
pcr_positive_daily.head()
Out: 日付 PCR 検査陽性者数(単日)
0 2020-01-16 1
1 2020-01-17 0
2 2020-01-18 0
3 2020-01-19 0
4 2020-01-20 0
In : cases_total: pd.DataFrame = pd.read_csv("https://www.mhlw.go.jp/content/cases_total.csv", encoding='shift_jis')
cases_total["日付"] = cases_total["日付"].astype(np.datetime64)
cases_total.head()
Out: 日付 入院治療を要する者
0 2020-02-04 15
1 2020-02-05 16
2 2020-02-06 12
3 2020-02-07 12
4 2020-02-08 7
In : recovery_total: pd.DataFrame = pd.read_csv("https://www.mhlw.go.jp/content/recovery_total.csv", encoding='shift_jis')
recovery_total["日付"] = recovery_total["日付"].astype(np.datetime64)
recovery_total.head()
Out: 日付 退院、療養解除となった者
0 2020-01-29 1
1 2020-01-30 1
2 2020-01-31 1
3 2020-02-01 1
4 2020-02-02 1
In : death_total: pd.DataFrame = pd.read_csv("https://www.mhlw.go.jp/content/death_total.csv", encoding='shift_jis')
death_total["日付"] = death_total["日付"].astype(np.datetime64)
death_total.head()
Out: 日付 死亡者数
0 2020-02-14 1
1 2020-02-15 1
2 2020-02-16 1
3 2020-02-17 1
4 2020-02-18 1
In : pcr_case_daily: pd.DataFrame = pd.read_csv("https://www.mhlw.go.jp/content/pcr_case_daily.csv")
pcr_case_daily["日付"] = pcr_case_daily["日付"].astype(np.datetime64)
pcr_case_daily.head()
Out: 日付 国立感染症研究所 検疫所 地方衛生研究所・保健所 民間検査会社 大学等 医療機関
0 2020-02-18 472.0 75.0 398.0 0.0 79.0 NaN
1 2020-02-19 15.0 68.0 609.0 0.0 0.0 NaN
2 2020-02-20 20.0 15.0 758.0 0.0 0.0 NaN
3 2020-02-21 261.0 188.0 902.0 132.0 108.0 NaN
4 2020-02-22 341.0 127.0 677.0 2.0 19.0 NaN「PCR検査実施人数」と「PCR検査の実施件数」の違いがよくわからない。
こっちの数値を見ると実施件数の方を実施人数としているようにも見える。
とりあえず実施件数の方を使う。
整形
それぞれのcsvで形式が違うので合成して扱いやすくする。
In: merged = pd.concat([
# column名を英単語へ
pcr_positive_daily.rename(columns={"PCR 検査陽性者数(単日)": "positive", "日付": "day"}).set_index("day"),
cases_total.rename(columns={"入院治療を要する者": "cases", "日付": "day"}).set_index("day"),
recovery_total.rename(columns={"退院、療養解除となった者": "recovery", "日付": "day"}).set_index("day"),
death_total.rename(columns={"死亡者数": "death", "日付": "day"}).set_index("day"),
# 各所での検査数を合計して新しいcolumnにする
pd.DataFrame(pcr_case_daily.rename(columns={"日付": "day"}).set_index("day").sum(axis=1).rename("pcr_case"))
], axis='columns').reset_index()
merged
Out: day positive cases recovery death pcr_case
0 2020-01-16 1 NaN NaN NaN NaN
1 2020-01-17 0 NaN NaN NaN NaN
2 2020-01-18 0 NaN NaN NaN NaN
3 2020-01-19 0 NaN NaN NaN NaN
4 2020-01-20 0 NaN NaN NaN NaN
... ... ... ... ... ... ...
168 2020-07-02 194 1105.0 16654.0 975.0 8749.0
169 2020-07-03 249 1224.0 16747.0 976.0 9873.0
170 2020-07-04 268 1386.0 16838.0 976.0 9917.0
171 2020-07-05 195 1550.0 16912.0 976.0 4456.0
172 2020-07-06 172 1671.0 16981.0 977.0 NaNこれを例えば陽性者数のグラフにしてみる。
In: plt.figure(figsize=(20, 20))
sns.barplot(x="day", y="positive", data=merged)
テレビとかで見るグラフっぽい感じに。増えてきているな。。
次に陽性者数と検査数の2軸グラフに。と思ったけど2ケースを一気にプロットしづらい。。種類のグラフにしづらい。
↓だとおかしくなる。yを2つは指定できないよね…?2軸グラフにしようとしても何故かエラーに。
In : sns.barplot(x="day", y="positive", data=merged)
sns.lineplot(x="day", y="pcr_case", data=merged)
ValueError: view limit minimum -36881.020000000004 is less than 1 and is an invalid Matplotlib date value. This often happens if you pass a non-datetime value to an axis that has datetime unitsmatplotlib側からすればどうとでもなるのだろうけど、seabornでいい感じに表示するにはdataframeを"index,key,value"の形式にする必要がありそう。
pandasクックブックによると「整然化」と呼ばれる処理らしい。
In : stacked = merged.set_index("day").stack().reset_index().rename(columns={"level_1": "key", 0: "value"})
stacked.head()
Out: day key value
0 2020-01-16 positive 1.0
1 2020-01-17 positive 0.0
2 2020-01-18 positive 0.0
3 2020-01-19 positive 0.0
4 2020-01-20 positive 0.0グラフ化(全国)
とりあえず全部表示。
In : plt.figure(figsize=(20, 20))
sns.lineplot(x="day", y="value", hue="key", data=stack)
それっぽい図。桁が違うのがあるのと、合計(単調増加)と日ごとの値が混じっていて見づらい。
とりあえず前回と同じ要領で2軸へ。
In : plt.figure(figsize=(20, 20))
sns.lineplot(x="day", y="value", color="blue", data=stacked[stacked["key"].isin(["positive"])])
ax1 = plt.gca()
ax2 = ax1.twinx()
sns.lineplot(x="day", y="value", color="red", data=stacked[stacked["key"].isin(["pcr_case"])])
最初のピークのときより陽性率は確かに下がってそう。
陽性率を出してみる。positive*100 / pcr_case。整然化済みのdataframeから作る方法がわからなかったのでmergedから。
In : merged["pcr_positive_per"] = merged.apply(lambda row: row["positive"]*100 / row["pcr_case"], axis=1)
stacked = merged.set_index("day").stack().reset_index().rename(columns={"level_1": "key", 0: "value"})
plt.figure(figsize=(20, 20))
sns.lineplot(x="day", y="value", color="blue", data=stacked[stacked["key"].isin(["pcr_case"])])
ax1 = plt.gca()
ax2 = ax1.twinx()
sns.lineplot(x="day", y="value", color="red", data=stacked[stacked["key"].isin(["pcr_positive_per"])])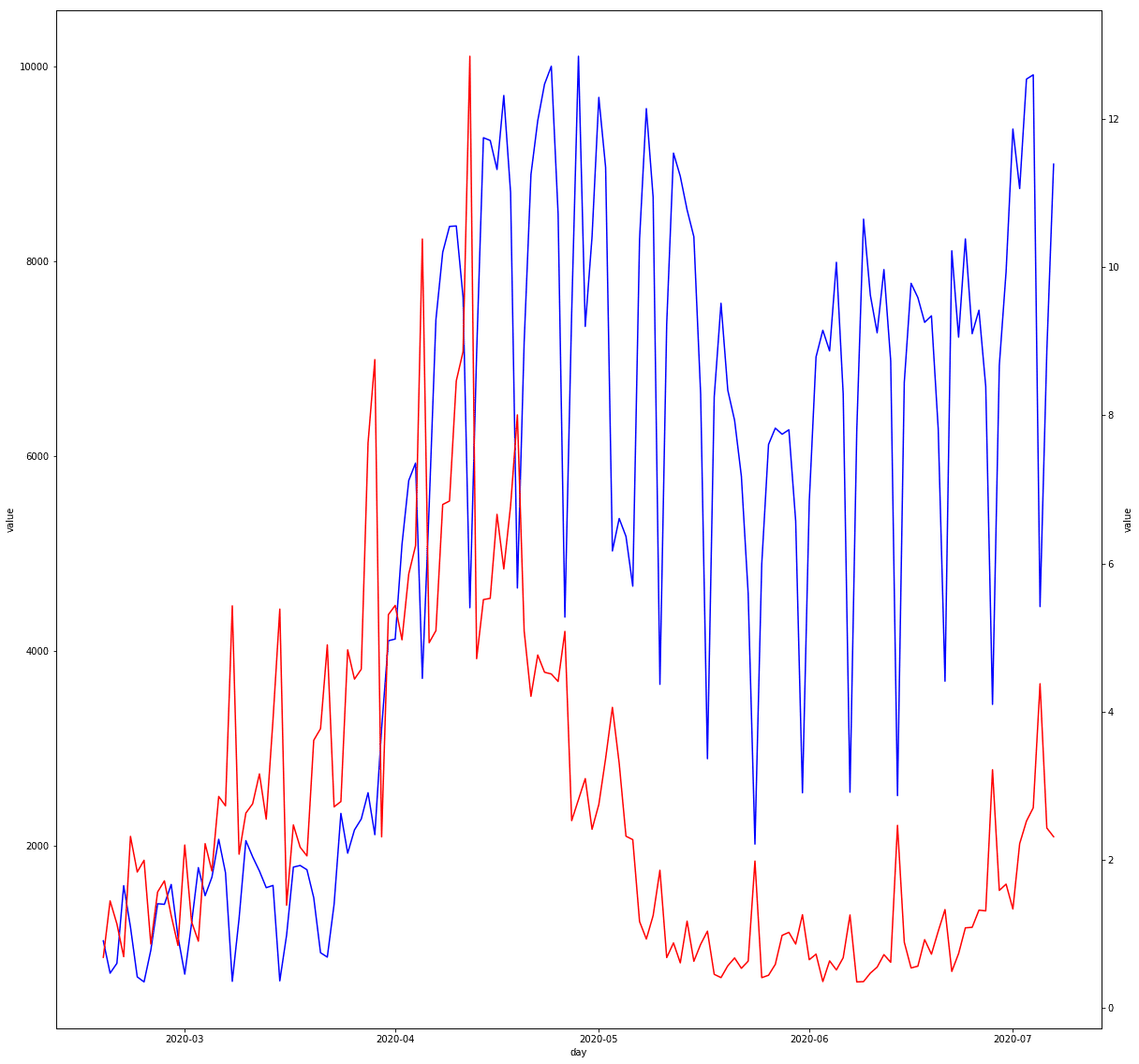
検査数が青(左軸)、陽性率が赤(右軸)。最悪期は陽性率が12%程度だった。でもそれでも10回検査して1回程度?それとも検査して判明するまで時間がかかるという話だったから比較すべき値がずれている…?
今は陽性率は4%くらい。とはいえ落ち着いていた頃より上がり気味。
陽性者数と死者数の推移
In : plt.figure(figsize=(20, 20))
sns.barplot(x="day", y="value", hue="key", data=stacked[stacked["key"].isin(["positive", "death"])])
グラフ化(東京)
東京のデータは統計値ではなく感染者ごと。まとめる必要がある。とりあえず日付だけ・年代別・性別別くらいを見てみる
In : tokyo_covid19_patients: pd.DataFrame = pd.read_csv("https://stopcovid19.metro.tokyo.lg.jp/data/130001_tokyo_covid19_patients.csv")
tokyo_covid19_patients["公表_年月日"] = tokyo_covid19_patients["公表_年月日"].astype(np.datetime64)
tokyo_covid19_patients.head()
Out: No 全国地方公共団体コード 都道府県名 市区町村名 公表_年月日 曜日 発症_年月日 患者_居住地 患者_年代 患者_性別 患者_属性 患者_状態 患者_症状 患者_渡航歴の有無フラグ 備考 退院済フラグ
0 1 130001 東京都 NaN 2020-01-24 金 NaN 湖北省武漢市 40代 男性 NaN NaN NaN NaN NaN 1.0
1 2 130001 東京都 NaN 2020-01-25 土 NaN 湖北省武漢市 30代 女性 NaN NaN NaN NaN NaN 1.0
2 3 130001 東京都 NaN 2020-01-30 木 NaN 湖南省長沙市 30代 女性 NaN NaN NaN NaN NaN 1.0
3 4 130001 東京都 NaN 2020-02-13 木 NaN 都内 70代 男性 NaN NaN NaN NaN NaN 1.0
4 5 130001 東京都 NaN 2020-02-14 金 NaN 都内 50代 女性 NaN NaN NaN NaN NaN 1.0まずは日付だけ。
In : tokyo = tokyo_covid19_patients.groupby(["公表_年月日"]).count()["No"].to_frame().reset_index().rename(columns={"公表_年月日": "day", "No": "value"})
In : tokyo.head()
Out: day value
0 2020-01-24 1
1 2020-01-25 1
2 2020-01-30 1
3 2020-02-13 1
4 2020-02-14 2
In : plt.figure(figsize=(20, 20))
sns.lineplot(x="day", y="value", data=tokyo)
次に年代別。
In : tokyo_old = tokyo_covid19_patients.groupby(["公表_年月日", "患者_年代"]).count()["No"].to_frame().reset_index().rename(columns={"公表_年月日": "day", "患者_年代": "old", "No": "value"})
In : tokyo_old.head()
Out: day old value
0 2020-01-24 40代 1
1 2020-01-25 30代 1
2 2020-01-30 30代 1
3 2020-02-13 70代 1
4 2020-02-14 50代 1
In : plt.figure(figsize=(20, 20))
sns.lineplot(x="day", y="value", hue="old", data=tokyo_old)
20代 > 30代 >> 40代、って感じ?4月のときは全般的に増加していたけど、今回は働く世代直撃って感じなのかな?
最後に性別別。
In : tokyo_sex = tokyo_covid19_patients.groupby(["公表_年月日", "患者_性別"]).count()["No"].to_frame().reset_index().rename(columns={"公表_年月日": "day", "患者_性別": "sex", "No": "value"})
In : tokyo_sex.head()
Out: day sex value
0 2020-01-24 男性 1
1 2020-01-25 女性 1
2 2020-01-30 女性 1
3 2020-02-13 男性 1
4 2020-02-14 女性 1
In : plt.figure(figsize=(20, 20))
sns.lineplot(x="day", y="value", hue="sex", data=tokyo_sex)
男性(青)の方が傾向として多いように見えるけどそこまで差はない、かな?
ちなみに少ないのは「不明」と「調査中」。
東京だけで見るなら緊急事態宣言が出た4月の時より感染者数が多くなっちゃったのね。。増加傾向ではあったけど、ここ数日で急にだな。全国的にも増加傾向だけど東京が異常。東京「だけ」異常なのかは他も見てみないとわからないなー
もうしばらく外出自粛しておこう。。
その他
lineplot以外もうまく使えるように成りたい。↓を見てもうちょっと勉強。
あと違う種類のグラフを重ねる方法も調べよう。
この記事が気に入ったらサポートをしてみませんか?
