
新規ゲノム情報を取得する de novo assemblyとはどんな解析?
こんにちは、バイオインフォマティシャンの竹本です。
プラチナバイオ株式会社では、バイオインフォマティクスを利用した標的遺伝子の特定、ゲノム編集、ゲノム編集後の解析までを一気通貫で行い、顧客の目的の表現型を構築する研究開発を進めています。
近年、ゲノム解析技術の進歩により、これまで解析が困難だった生物種のゲノム情報を取得できるようになりました。この技術革新は、農業から医療まで幅広い分野に変革をもたらしています。最近、弊社でもこれまでにゲノム解析をしたことのない生物の育種、特にゲノム解析をベースにしたデータ駆動型育種に関するお問い合わせを多くいただいています。そこで今回は、新規ゲノム情報を取得する重要な手法である「De novo assembly」についてご紹介します。
de novo assembly解析とはそもそも何か?
de novo assemblyは、ラテン語で「新たに」「再び」(de novo)、「組み立てる」「集める」(assembly)という意味を持ちます。つまり、何もないところからゲノム情報を「新たに」「組み立てる」解析手法です。
ゲノム配列はA(アデニン)、T(チミン)、G(グアニン)、C(シトシン)の4種類の塩基の組み合わせでできています。この配列を読み取るのがDNAシーケンサーと呼ばれる機器です。弊社では、高性能なDNAシーケンサー2機種によるシーケンスサービスを提供しています。


DNAシーケンサーで読み取ったATGCの連続的な配列情報を、パズルのピースをつなぐように組み立てて、できるだけ長い配列情報を構築するのがde novo assembly解析です。
ここではPacBio Sequel IIeでデータを取得する場合を例に説明します。
まず、DNAシーケンサーはHiFi-reads(High-Fidelity reads; ハイファイリード)と呼ばれる長くて高精度な塩基データを出力します。
次に、これらのHiFi-readsの中で互いに似ている部分を見つけて繋いでいきます。この繋がった配列をContigs(コンティグ)と呼びます。
さらに長い配列を構築するためには、生物種のゲノムの大きさや複雑さ、解析の目的に合わせて配列をつなぐヒントとなるデータを追加します。この過程を経て、より長い配列であるScaffolds(スキャフォールド)を構築します。最終的には、Chromosome(クロモソーム)と呼ばれるゲノム全長の情報の構築を目指すこともあります。
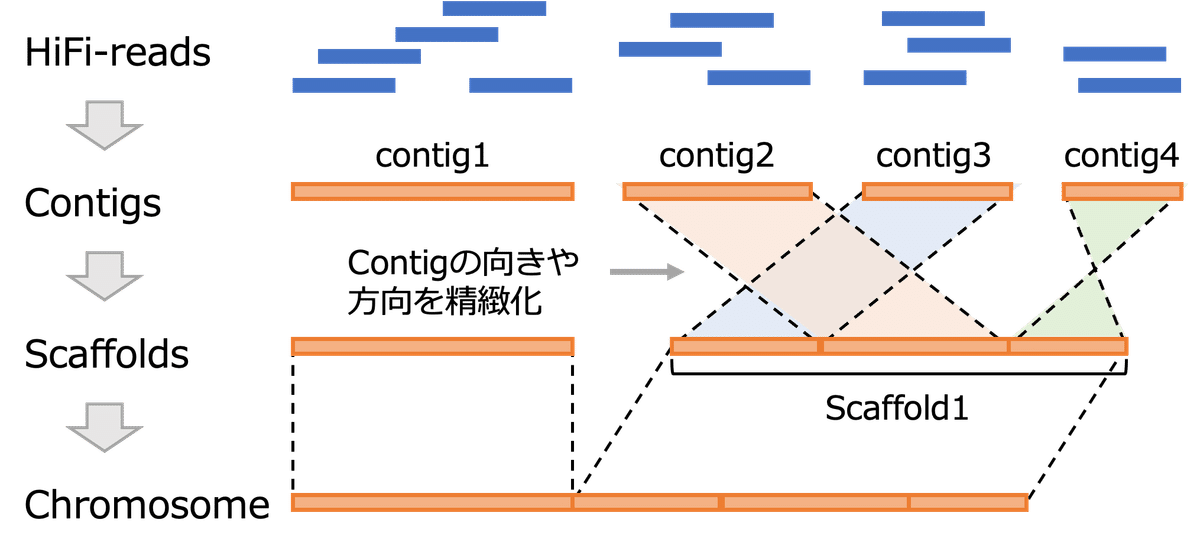
このように段階的にゲノム配列をつなぎ合わせて組み立てていくのが、de novo assembly解析の基本的なプロセスです。
近年、DNAシーケンサーの精度向上、解析ツールの技術向上に伴い、すでにゲノム情報をもつと思われてきた生物種でも再度ゲノム配列を構築して精度を向上させたり、品種が同じでも系統が異なる植物のゲノム配列を読み直して、独自のゲノム情報を構築したりする例が見られます。
de novo assembly解析結果の活用例は?
de novo assembly解析で得られたゲノム情報は、ATGCの羅列に過ぎません。しかし、この情報は多くの分野で活用可能です。以下に具体例を挙げます。
・農業分野:病気に強い作物の選抜
de novo assemblyでトマトの基準ゲノムを構築
耐病性のある品種と病気に弱い品種のゲノム情報を比較
耐病性に関連する遺伝子(DNAマーカー)を特定
DNAマーカーを使って、栽培前に耐病性品種を選抜
・医療分野:新型ウイルスの解析
ウイルスのゲノム情報をde novo assemblyで構築
既知のウイルスと比較し、特徴を把握
変異株の発生状況の追跡や、ワクチン・治療薬の開発に向けた解析
・環境分野:未知の微生物の発見
海水に含まれる複数の微生物のゲノム情報をde novo assemblyで構築
新種の微生物を同定
新種の微生物が生産する有用物質等の発見
農業分野の例については、現在、トマトやコメなどの品種改良に応用されています。
プロジェクトの最終目標は、ある遺伝子Aに見られる特徴的な配列の変化が、植物に耐病性をもたらすことを確認し、DNAマーカーによる選抜を実現することです。
DNAマーカーとは、耐病性のような形質を反映するゲノム配列中の顕著な変化のことを指します。DNAマーカーが確立されれば、DNAマーカーの有無を確認するだけで、耐病性をもつ植物ともたない植物を選抜すること(DNAマーカー選抜)が可能となります。
このプロジェクト全体を通じて、de novo assemblyによって得られた新規ゲノム配列情報が重要な基準となるゲノム情報(基準ゲノム)の役割を果たします。
解析の過程で、ゲノム情報の中からある形質を反映する遺伝子領域を同定することや、異なる形質をもつ植物同士のゲノム配列を比較するためには、基準となるゲノム情報(基準ゲノム)が必要です。
それは、基準ゲノムが遺伝子領域の同定や、配列の比較解析において参照点となるためです。この基準ゲノムが正確であればあるほど、解析結果の信頼性が高まります。
(注:遺伝子領域の特定方法や配列比較の詳細については、別の記事で解説する予定です。)
このように、de novo assembly解析によって構築されたゲノム情報(基準ゲノム)は、DNAマーカー開発の全プロセスを支える重要な基盤情報となります。
おわりに
今回は、de novo assembly解析とその活用事例についてご紹介しました。
de novo assembly解析は、ゲノム情報を活用した研究開発の基盤となる重要な解析技術です。この技術を活用することで、農業・医療・環境などさまざまな分野でブレークスルーが期待できます。
弊社では、de novo assembly解析を含むゲノム解析サービスを提供しています。解析の進め方や具体的な応用についてもご相談を承っておりますので、ご興味をお持ちの方は、お気軽に弊社問い合わせ窓口(info@pt-bio.com)までご連絡ください。
記事作成者:竹本 美沙子 / Misako TAKEMOTO, Ph.D.
2022年入社。博士(生命科学)。
大学の技術職員としてバイオインフォマティシャンのキャリアをスタート。現在はプラチナバイオ株式会社研究開発部 バイオインフォマティシャンとして、植物・動物問わず、さまざまな生物のゲノム解析を担当。
どんな人にもわかりやすくをモットーに、ゲノム解析のあれこれについて発信していきます。
