
ヘビの大規模ゲノム解析から脊椎動物の発生に関する知見が得られる
ヘビの大規模ゲノム解析から脊椎動物の発生に関する知見が得られる
https://www.cell.com/cell/fulltext/S0092-8674(23)00582-2?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS0092867423005822%3Fshowall%3Dtrue
Changjun Peng 8
呉東東 8
ジン・ロン・レン 8
秦殷
張志毅
リ・ジアタン 9
すべての著者を表示する
脚注を表示する
オープンアクセス掲載:2023年6月19日DOI:https://doi.org/10.1016/j.cell.2023.05.030
PlumXメトリクス
ハイライト
代表的なヘビゲノムの包括的な染色体レベルアセンブリ
四肢欠損、細長い体、左右非対称な肺の遺伝的基盤を解明
ゲノム変異がヘビの盲目と赤外線感受性の特殊性を駆動する
機能実験により、比較ゲノム学的発見が検証される
概要
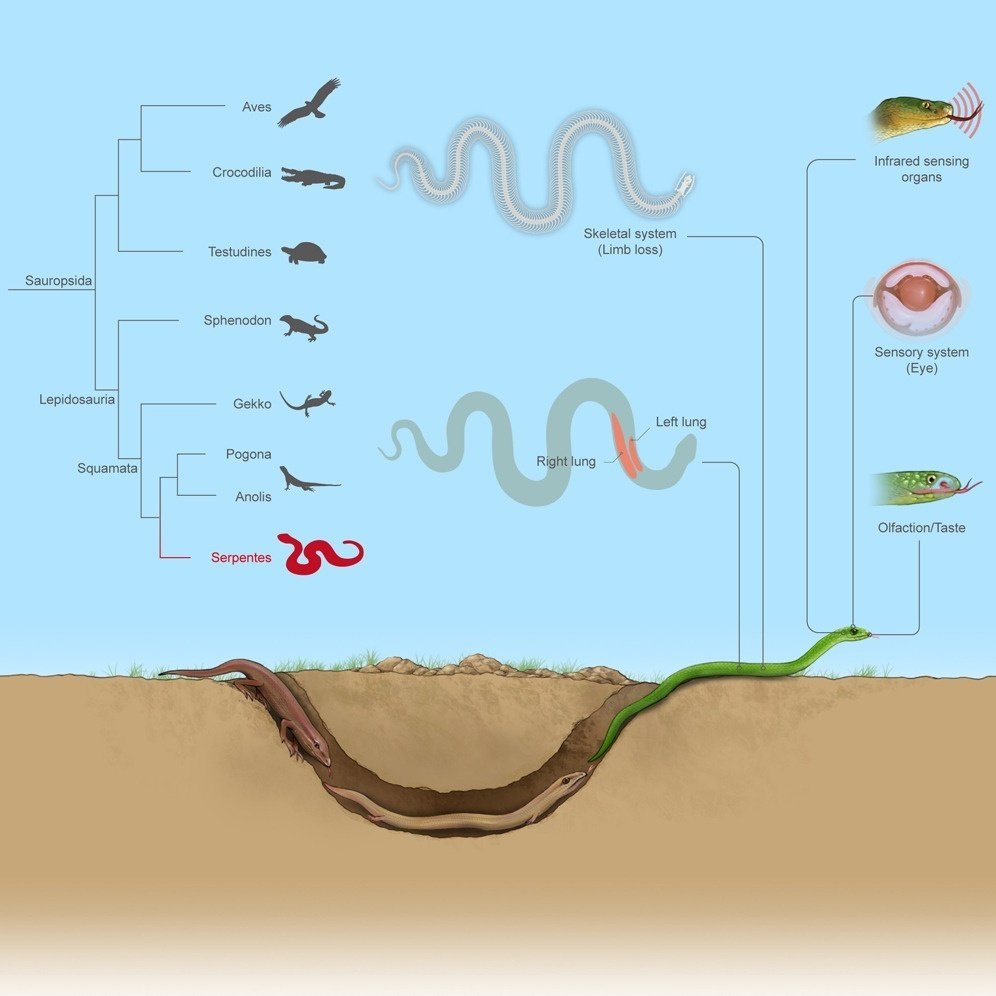
ヘビは、特に脊椎動物の骨格、器官、感覚系の進化に関連した、ユニークな形態的適応を持つ驚くべき扁形動物系統である。ヘビの表現型の遺伝的基盤を明らかにするために、我々は12科のヘビから14のde novoゲノムを組み立て、解析した。また、機能実験を用いてヘビの形態学的特徴の遺伝的基盤を調べた。その結果、ヘビにおける四肢の欠損、細長い体型、非対称な肺、感覚器系、消化器系の適応の進化に寄与している可能性のある遺伝子、制御エレメント、構造変異を同定した。また、視覚の進化、盲目のヘビにおける骨格系と食性、赤外線に敏感なヘビにおける体温受容の進化を形成した可能性のある遺伝子と制御要素をいくつか同定した。本研究は、ヘビと脊椎動物の進化と発達に関する知見を提供するものである。
グラフィカル抄録
大きな画像を見る
高解像度画像をダウンロード
キーワード
ヘビ
系統発生学
ゲノム進化
多組織トランスクリプトーム
表現型の進化
遺伝的メカニズム
はじめに
ヘビは、極地を除くすべての大陸への侵入に成功している、
1
現存する種は約30科4,000種にのぼる。
2
ヘビは効率的な捕食者であり、生態系のバランスを保つ上で重要な役割を果たしている。ヘビは穴の中に潜る祖先から分岐して以来、様々な進化的変化を経験してきた。例えば、体型の変化(細長い体型や非対称な肺など)、四肢の喪失、化学受容や体温受容の変化、摂食行動の変化などであり、他のほとんどの扁形動物(トカゲやミミズトカゲ)とは異なっている。一部のヘビは、ユニークな生活様式や形態的革新を進化させてきた。例えば、盲目のヘビは穴を掘って生活し、特殊な食性(主にキチン質を多く含むアリやシロアリなどの小型節足動物)を持つ、
3
,
4
また、赤外線に敏感なニシキヘビ、イノシシ、毒蛇は、特殊な穴器官や受容体を進化させ、体温感知を高めている。
5
,
6
このようなユニークな適応を持つヘビは、発生生物学の研究や複雑な形質の進化を研究するための優れたモデルシステムとなっている。
ヘビはそのユニークな生物学的特徴や生態学的重要性にもかかわらず、多様な表現型や自然史を含むヘビに関する我々の理解はいまだ不十分である。さらに、高品質なヘビゲノム(脊椎動物ベンチマークユニバーサルシングルコピーオーソログ[BUSCO]スコア≥90%、コンティグN50≥1 Mb)の数が限られているため、特定の形質の根底にある遺伝的メカニズムはまだ明らかにされていない。
7
,
8
,
9
および家族保険。
9
,
10
,
11
,
12
,
13
,
14
,
15
,
16
ヘビの進化についての理解を深めるため、ヘビ亜目の12科14種のヘビのゲノムをde novoでアセンブルした(図S1A)。この12科には全ヘビ種の約84%が含まれていることから、今回のゲノムセットはヘビの生命樹を非常に代表するサンプルである(表S1)。また、これらの分類群のゲノム進化を明らかにするために、既発表のゲノム(ヘビ11種、トカゲ6種、カメ1種、ワニ1種、鳥1種)と、今回新たに塩基配列を決定したトランスクリプトーム(ヘビ9種、トカゲ1種、マウスから194サンプル)を用いた(表S1)。スクアメイト系統の起源と進化、ヘビ種のユニークな形態適応が明らかになっただけでなく、今回の結果は脊椎動物の発生生物学的理解を深めるものである。
図S1図1に関連するヘビの系統樹と分岐時間樹
キャプションの全文を表示
大きな画像を見る
高解像度画像のダウンロード
成果
ゲノムシーケンス、アセンブル、アノテーション
ロングリード(PacBioおよびOxford Nanopore Technology)を用いたde novoアセンブルの結果、14のドラフトヘビゲノムが得られた(表S1)。BGIとイルミナのショートリードを用いてアセンブリーを研磨し、高品質なゲノム(コンティグN50:3.8-56.3 Mb、平均:26.2 Mb)を得た(表1)。Hi-Cリードデータを用いて12種のヘビゲノムを染色体レベルでさらにアセンブルしたところ(表1)、大部分の核型は先行研究と一致していた(表S1)。アセンブルされたゲノムは非常に完全であり(BUSCOスコア:91.50%-96.10%)(表1)、すべてのアセンブルは下流の比較解析にとって高品質であることが示された。
表1ヘビ14種のゲノムアセンブリー統計量
学名英語名略号アセンブルされたゲノムサイズ (Gb)スキャフォールドN50 (bp)コンティグN50 (bp)BUSCO(%)Euprepiophis perlacea四川ラットスネークEper1. 64215,947,93854,236,13694.1Pantherophis guttatusred cornsnakePgut1.73146,850,88226,650,24294.2Calamaria septentrionalisHong Kong dwarf snakeCsep1. 95249,103,79513,457,42391.5Cylindrophis ruffusred-tailed pipe snakeCruf1.55204,348,66647,299,78696.1Eryx tataricusTartar sand boaEtat1. 53210,175,34317,592,29795.0Hypsiscopus plumbeaplumbeous water snakeHplu1.51-38,336,52393.9Psammodynastes pulverulentuscommon mock viperPpul1. 59216,958,05332,196,31593.9Boaedon fuliginosusAfrican house snakeBful1.72159,198,18824,942,69692.4Leptotyphlops nigroterminusblack-tip worm snakeLnig1. 97-3,846,21192.5Pareas berdmoreikeeled slug snakePber1.85144,018,27113,457,42393.8Argyrophis diardiiDiard's blind snakeAdia1. 79265,014,50025,570,70493.3Gloydius shedaoensisShedao island pitviperGshe1.52191,459,3156,670,06795. 6Achalinus jinggangensis Zong's odd scales snakeAjin1.73226,357,5978,252,46192.7Xenopeltis unicolorsunbeam snakeXuni1.42199,476,79956,968,22894.2
新しいタブで表を開く
調査した種のうち、コーディング遺伝子の予測数は18,301から23,299であった(表S1)。平均的な遺伝子、コーディング配列(CDS)、エクソンの長さは、以前に発表されたヘビゲノムで報告されたものと同様であった(表S1)。本研究で紹介したゲノムおよびトランスクリプトームデータを可視化するために、ユーザーフレンドリーで一般公開されているゲノムブラウザデータベースを構築した(http://herpmuseum.cib.ac.cn/snake/)。
ヘビの系統と起源
Progressive Cactusによって作成された31タクソンのアライメントに基づく、
17
をもとに、25のヘビ、4つのトカゲ、1つのカメ、1つのワニのゲノムワイド系統樹を推定した。高品質なゲノム(コンティグN50≧180kb、または染色体レベルまでアセンブルされ、BUSCOスコア≧94%)をアウトグループ分類群として用いた。合計約51 Mbのオルソログ配列が抽出され、全ゲノム系統樹が十分に支持された(図1およびS1B)。また、31種の爬虫類で同定された1:1のオルソログタンパク質コード遺伝子1,980セットと、これらの遺伝子の4倍縮退部位、保存されたノンコーディングエレメント(CNE)を用いて、さらに系統解析を行った。最後に、ASTRAL-IIIを用いて合体種樹を推論した。
18
を用い、オルソログ配列と遺伝子を用いて合体樹を推定した。その結果、すべてのトポロジーはゲノムワイドのツリーと同じであり(図1、S1B-S1G)、ヘビの進化関係を調べた先行研究と一致した。
19
,
20
Serpentesは単系統でLacertiliaの中に入れ子になっており、現存するヘビはトカゲから発生したことを示している。さらに、ヘビの最も新しい共通祖先は、(ヨーロッパガラストカゲ+コモドドラゴン)クレードと姉妹関係にあった。ヘビでは、"Scolecophidia "クレード(盲目のヘビ)が(Typhlopidae + Leptotyphlopidae)を構成し、これは最も基層のヘビクレードであった。連結されたオーソログ配列と遺伝子樹はほぼ100%の支持を受け、種樹と同じトポロジーを持つが、HenophidiaとLamprophiidaeではそれぞれいくつかの遺伝子樹の不一致が見られる(図S1F-S1H)。異なるトポロジーの割合の差が小さく、枝の長さが比較的短いことから、この不一致は種の分岐初期に起こった不完全な系統選別(ILS)に起因する可能性が示唆される。さらに、推定された分岐時期から、ヘビは白亜紀前期(約1億1768万年前[mya])に誕生し、白亜紀-古第三紀(K-Pg、約65mya)の大量絶滅後に急速に多様化したことが示された。
21
,
22
,
23
(図1)。
図1ヘビの系統図
キャプションの全文を表示
大きな画像を見る
高解像度画像のダウンロード
ヘビゲノムの特徴と進化
染色体進化
私たちは、新たに構築した染色体レベルゲノムと、一般に公開されている4つの染色体レベルゲノム(表S1)を用いて、ヘビ類の祖先ゲノムを復元した。ヘビの祖先ゲノムの長さは約1.56Gbで、8本のマクロ染色体と15本のミクロ染色体を含む23本の染色体から構成されていた(図2AとS2A;表S2)。ヘビのZ染色体はトカゲの常染色体に由来している可能性があり、少なくとも1回の切断現象が起きている。祖先ヘビゲノムの保存は、調査したほとんどのヘビゲノムで明らかであったが、染色体融合と切断イベントが最も多く起こったのはホンコンヒメヘビであったようだ(図S2B)。
図2ヘビゲノムの進化的特徴
キャプションの全文を表示
大きな画像を見る
高解像度画像のダウンロード
図S2図2に関連するヘビゲノムの特徴
完全なキャプションを表示
大きな画像を見る
高解像度画像をダウンロード
ゲノムサイズ、GC含量、進化速度
ヘビのゲノムアセンブリーのサイズは1.42Gb(サンビームヘビ)から1.97Gb(アカオビパイプヘビ)であった(表1)。平均サイズは1.68Gbで、トカゲゲノムのサイズ(平均約1.57Gb)とほぼ同じであった、
25
,
26
のゲノムよりも小さかった。
27
やチャイニーズ・アリゲーター(約2.27Gb)よりも小さい、
28
ニワトリゲノム(約1.07Gb)より大きい。
29
(図2B)。
トランスポーザブル・エレメント(TE)はヘビゲノムの35%~55%を占めていた。相関解析の結果、TEバーストがヘビ、トカゲ、カメ、鳥、ワニのゲノムの拡大を牽引していることが確認され、LINE/CR1配列とLINE/L2配列がゲノム拡大の主なドライバーであった(図2B)。これらの結果は、Squamataのゲノムサイズとリピート含有量に強い相関がないことを示した先行研究とは対照的である。
30
これらの食い違いは、全ゲノム解析でイルミナのショートリードを使用したことが原因で、リピート含量の計算にバイアスがかかっている可能性がある。
31
CpGアイランド密度とGC含量は、すべてのヘビのゲノムで、アウトグループと再構築された爬虫類の祖先のゲノムよりも低かった(GC含量: GC含量:38.14%対40.09%;CpG島密度2.55カウント/kb対3.40カウント/kb)。さらに、CpGアイランド密度とGC含量は、進化したヘビよりも進化したヘビの方が高く、ヘビの進化の歴史の中でCpGアイランド密度とGC含量が減少してきたことを示している(図2C)。これらの結果は、Schield et al、
13
ヘビゲノムの進化パターン(ATの蓄積に言及)が鳥類と異なることを示したSchieldら、13
32
や哺乳類とは異なる。
33
その結果、r8s
34
はヘビの体長と負の相関があることがわかった(図S2C)。これは、体長がゲノム進化速度と有意に負の相関があることを示す反芻動物の研究結果と一致している35。
35
これらの結果は、体長とゲノム進化速度の関係は脊椎動物のグループ間で類似していることを示唆している。
ゲノム構造の変異
31分類群のゲノムアラインメントによると、ヘビゲノムでは、アウトグループと比較して、24,352,060 bpのユニークなセグメントと16,146,509 bpの失われたセグメントが同定された(図S2D)。このうち、長さ50bp以上の17,877の挿入と23,808の欠失がヘビ特有のゲノム構造変異(SSSV)として同定された。これらのSSSVの機能アノテーションの結果、34.7%がコーディング遺伝子領域内またはその近傍(転写開始および終了部位を挟む5kbの領域)に位置し(図S3A;表S3)、その中には四肢、眼球、眼瞼、体節、肺の発達や呼吸機能に関連する遺伝子が含まれていた(表S3)。このことは、SSSVがヘビ特異的形質の進化に貢献した可能性を示唆している。
図S3ヘビ特異的ゲノム構造変異(SSSVs)、ヘビで分岐したがアウトグループで保存されている非コードエレメント(SD-CNEs)、進化解析に用いたオルソログ遺伝子(図3およびSTAR Methods関連
キャプション
大きな画像を見る
高解像度画像のダウンロード
コーディングエレメントとノンコーディングエレメントの進化
31分類群の全ゲノムアラインメントを用いて、ヘビにおいて新たに進化した142のコーディング遺伝子と失われた156のコーディング遺伝子を同定した(表S3)。エンリッチメント解析の結果、新たに進化した遺伝子と失われた遺伝子は、感覚系と消化器系の機能に富んでいることが示された。さらに、ヘビにおいて419の正選択遺伝子(PSG)と270の急速進化遺伝子(REG)を同定した(表S3)。PSGは、骨格系、肺、眼の発達に関連する機能を含む79のマウスゲノムインフォマティクス(MGI)用語で有意に濃縮されている(表S3)。REGはMGI用語の骨格系と肺の発生に関連する機能、味覚伝達(ko04742)、骨の成熟に関与する骨化(GO:0043931)に濃縮されている(表S3)。このように、骨格系や感覚系に関与する遺伝子は、ヘビの進化において重要な役割を果たしてきた可能性がある。GERPとPhastConsのスコアから、合計116,707のCNEが31種の間で共有されていることが示された。我々は1,686個のヘビから分岐したCNE(SD-CNE、ヘビで分岐したがアウトグループで保存されているCNE)を同定し、関連遺伝子はヘビの形質に関与する48のMGI用語で濃縮されている(図S3D)。したがって、手足に加えて
36
の変化もSD-CNEの進化に起因している可能性がある。
骨格系の進化と器官適応に関与するゲノムエレメント
祖先のヘビの表現型は、おそらく穴を掘って這うような生活様式に適応しており、それに続いて他の特殊な形質が進化したのであろう。
37
,
38
ヘビの特殊な体形と骨格系(四肢の欠損、細長い体、柔軟な下顎など)は、ヘビの特徴的な形質と考えられている。ヘビは細長いボディプランを実現するために、非対称の肺を含むユニークな器官形状を進化させてきた。
39
,
40
,
41
これらの形質の遺伝的基盤を調べるため、ゲノムおよびトランスクリプトームデータを用いた比較解析を行った。
四肢喪失の基盤となる遺伝的要素の進化
現存するヘビは前肢を完全に失っており、進化したヘビにはない初歩的な後肢を残している。
42
,
43
四肢欠損の遺伝的メカニズムを調べるため、これまでの四肢発生の研究では、ヘビで分岐したノンコーディングレギュレーターの進化を明らかにすることに焦点が当てられており、これらの要素がヘビの四肢欠損に寄与している可能性が示唆されている。
44
ここでは、ヘビの四肢欠損に対するコーディング遺伝子と構造変異の進化の寄与の可能性に注目した。その結果、前肢発生開始因子RDH10
45
はヘビで欠損しており(表S3)、RDH10のもう1つのコピーがSD-CNEに囲まれていた(図S3D)ことから、ヘビの前肢欠損はこのイニシエーターの発現変化によって制御されている可能性が示唆された。
ソニックヘッジホッグ受容体PTCH1と細胞極性エフェクターCPLANE1では、それぞれ3つのトキシコフェラ特異的(すなわち、ヘビ、アンギモルフ、イグアナトカゲ)および4つのヘビ特異的アミノ酸残基欠失が同定された(図S4AおよびS4B)。PTCH1は胚の四肢形態形成に必須な役割を果たしている、
46
,
47
CPLANE1は胚の指の形態形成に関与している。
48
これらの遺伝子の欠失は、これらの遺伝子の機能を変化させ、ヘビの四肢発生に影響を与える可能性が示唆された。さらに、8つの既知の四肢発生関連遺伝子の潜在的な制御領域(転写開始および終了部位を挟む5kbの領域)がSSSVと重なっていた(表S3)。SHH遺伝子エンハンサー(ZRS)の17bpのヘビ特異的欠失は、四肢の異常発達に寄与することが知られている、
44
の欠失は、新たに配列決定されたすべてのヘビゲノムで観察された(図S4G)。また、進化したヘビではZRS配列のほぼ全体が失われていることもわかった(図S4G)。この結果は、コーディング遺伝子の変異と構造的変異の両方が、ヘビの四肢欠損にも寄与している可能性を示している。
図S4ヘビの四肢欠損、体の伸長、肺の非対称性、眼構造の単純化の遺伝的基盤(図3、4に関連
キャプションの全文を表示
大きな画像を見る
高解像度画像のダウンロード
ヘビの四肢欠損に関与している可能性のある候補遺伝子の配列を同定したが、遺伝子と表現型を結びつけるメカニズムを直接推論するのに必要な証拠が不足していた。そこで我々は、CRISPR-Cas9技術を用いて、1つのコード遺伝子(PTCH1)の欠失が四肢の発達にどのような影響を及ぼすかを調べた。Toxicoferaの中では、65%の種に四肢がない、
2
我々はトキシコフェラに特異的なアミノ酸残基の欠失を同定し、それらが四肢の発達に関連した機能を持つ可能性を示唆した。これらの欠失は細胞外トポロジカルドメインに位置していた(図S4A)。このドメインに1アミノ酸残基が挿入されると、ヒトでは手足の異常な発達につながる可能性がある。
49
したがって、これらの欠失がトキシコフェラにおける四肢欠損の分子メカニズムの根底にあるのではないかと考えた。
これらの欠失の機能的影響をin vivoで調べるため、CRISPR-Cas9技術を用いてマウスPTHC1遺伝子を編集し、946-948 bpを欠失したPTCH1マウス(以後、snake欠失PTCH1変異マウスと呼ぶ)を得た。1週齢のPTCH1変異マウスは、明らかな四肢欠損を示さなかったが、野生型(WT)マウスと比較して相対体重が有意に低く、胴体が短かった(図S4C)。1週齢のPTCH1変異マウスはまた、海綿骨体積(BV)および組織体積あたりの海綿骨BV(BV/TV)が有意に小さかった(図S4D)。さらに、成体のPTCH1変異マウスでは、第4前肢指骨と第3後肢指骨が有意に短縮していた(図3DおよびS4E)。これらの結果は、この遺伝子の欠失が四肢指の成長を含む骨格の成長を阻害することを示唆している。PTCH1変異マウスの四肢芽には、WTマウスと比較して、前肢で12個の発現差のある四肢発生関連遺伝子(DELG)(SHH、ID4、HOXD11、FGF8を含む)、後肢で22個のDELG(SP9、DLX6、PLXNA2、PITX1、EN1を含む)が含まれていた(図S4F)。これらの結果は、PTCH1の欠失がその機能を変化させ、四肢芽における遺伝子の発現を変化させることによって四肢の指の成長を阻害することを示唆している。したがって、Toxicofera内の欠失が骨格系の進化と四肢欠損に関与していることが示唆され、これはヘビや四肢のないトカゲにおける外適応を表しているのかもしれない。
図3ヘビにおける骨格系の進化と器官適応の遺伝的基盤
キャプションの全文を表示
大きな画像を見る
高解像度画像のダウンロード
嚥下と消化に関する遺伝的変異
ヘビは前肢を失っているため、獲物の捕獲と摂食を容易にするために他の形態学的特徴を進化させてきた。例えば、独立して動く上顎と大顎、頭蓋骨にある反り返った口蓋歯などである。
41
(図3A)。ここでは、85個のSD-CNEが下顎の形態形成に関与する38個のコーディング遺伝子と関連していた(図S3D)。さらに、下顎と上顎の形態形成に機能的に関連するいくつかのコーディング遺伝子は、ヘビにおいて強い選択シグナルを示した(図3A;表S3)。その中でも、LIMドメインキナーゼ2(LIMK2)とマトリックスメタロプロテアーゼ-14(MMP14)は正常な頭部の成長に必須である
50
ヘビの頭部の進化に関与している可能性がある。ホメオボックスタンパク質HOX7(MSX-1)は、象牙質を含む歯の歯牙形成の制御に関与する重要な転写抑制因子である51、
51
,
52
はヘビで急速に進化した(図3A)。これらの結果は、遺伝子や制御因子の進化が、ヘビ特有の頭蓋骨構造の進化を促進した可能性を示唆している。
ヘビの頭蓋骨の特殊な特徴により、ヘビは獲物を丸呑みすることができる。
41
獲物を丸呑みすることができる。
53
,
54
,
55
ヘビはこのように、使わないときは収縮し、食べ物を食べると素早く膨張する効率的な消化器官を進化させてきた。
56
我々は、脊椎動物の食欲刺激に関与する食欲調節ホルモンGHRLとその受容体GHSRを発見した、
57
,
58
,
59
,
60
はヘビでは失われている(図3AおよびS3F)。胆汁酸生合成抑制因子MALR1
61
もヘビで失われている(図3AおよびS3F)。対照的に、ヘビでは2つの補体因子H(CFH)が新たに進化した(図3AおよびS3E)。進化解析の結果、2つの消化器系関連遺伝子(非リソソーム型グルコシルセラミダーゼ[GBA2]と陰イオン型トリプシン-2[TRYP2])が正の選択を受けていることが明らかになった(図3A)、
62
は急速に進化した。GBA2とTRYP2は消化を担っている、
63
,
64
とCFHは食餌過剰に反応する。
65
遺伝子の交替と更新は、ヘビが長期間絶食し、その後大きな獲物を急速に消化できる理由を説明しているかもしれない。
細長いボディプランと非対称な肺の進化
四肢の欠損は運動機能に直接的な影響を及ぼし、ヘビは高度に特化した運動システムを進化させてきた。その結果、ヘビの骨格系は、細長いボディプランを含む特定の表現型を何度も進化させてきた。
66
本研究では、5つのPSG(LOX3B、FOXB1、DLC、MESP1、FOXC2)が体節形成に関連するGOタームに富んでいる(図3B)。DLCはヘビで急速に進化し(図3B)、体節形成を担う重要なタンパク質であり、ゼブラフィッシュでこのタンパク質を変異させると体節形成不全になる67。
67
,
68
FOXC2は、マウスの前方体節形成に寄与するよく知られた遺伝子である。
69
また、SD-CNEに関連する16のコーディング遺伝子が、体節サイズ変化に関連するMGI用語において有意に濃縮されていることを同定した(図S3D)。さらに、10個のHOX遺伝子が緩やかな選択を受けており(表S3)、これは以前の研究の結果と同様である。
9
我々は、体節形成と仕様に関与する2つのよく知られた中核遺伝子であるRIPPLY2とMEOX1の潜在的な制御領域にSSSV挿入を見出した
70
,
71
,
72
,
73
(表S3)。これらの遺伝子は、Ptyas dhumnades胚の骨化前脊椎(排卵後40日[dpo])でも発現しており、FOXB1は他の組織と比べて高い発現を示していた(図S4H)。これらの遺伝的要素は、ヘビにおける細長い体の進化を説明するかもしれない。
ヘビの細長い体の進化は、肺を含む内臓器官の変化を伴っていた。
41
現存するヘビでは、左肺は存在しないか、残存しているか、発達しているが右肺より小さい。
39
,
40
我々は、左右対称性の発生に重要な役割を果たすダイニンをコードする遺伝子の1つ(DNAH11)を発見した、
74
はヘビには存在しなかった。左右非対称の発生に影響するフォークヘッドボックスタンパク質J1をコードする遺伝子(FOXJ1)は
75
,
76
と肺の発達を促進する、
77
もヘビでは失われている(図3C;表S3)。肺胞上皮の発達を制御するAP3B1(図S4I)には、ヘビに特異的な6つのアミノ酸残基の欠失が検出された。
78
SD-CNE関連遺伝子は、肺の異常な発達と形態および小肺に関連するMGI用語において有意に濃縮されている(図S3D)。また、13の肺発生関連遺伝子(例えば、CRISPLD2、FGF18、TITF1、MAN2A1、GRHL2、SEC24B、NFIB、PDE4D)の潜在的な制御領域におけるSSSV挿入を同定した(表S3)。比較トランスクリプトーム解析により、40dpoのP. dhumnades胚の左右の肺で発現が異なる遺伝子が2,195個明らかになった。FGF18、NFIB、FOXF1など、これらの遺伝子のいくつかは肺の発生に必須であることが同定されている
79
,
80
(図S4J)。これらの発現差遺伝子は、肺の発生を含む機能(GO:0030324)に富んでおり、上位20の有意に発現差のある遺伝子は、肺胞の発生に関与するいくつかのGO用語に富んでいる(表S3)。また、成体の左右の肺で発現が異なる遺伝子(168遺伝子)は、胚のそれよりもはるかに少ないことに気づき、発生段階における遺伝子の発現パターンの違いの重要性を強調した。さらに、上記の潜在的非対称遺伝子の周辺にあるいくつかのCNEの同一性(祖先配列に対する相対的な)は、肺の非対称性が増すにつれて減少することも観察された(図S4K)。発現の異なる遺伝子、関連するCNE、および対称的発生関連遺伝子の欠損の同定は、ヘビの肺の非対称的発生に関する洞察を提供した。しかしながら、根本的なメカニズムを解明するためには、さらなる研究が必要である。
加重遺伝子共発現ネットワーク解析(WGCNA)を行った結果、トカゲの肺機能に関連する遺伝子はヘビとは異なることが示された。3つのハブ遺伝子(SFTPC、CHI3L1、TITF1)と4つの肺発生関連遺伝子(SAV1、SEC24B、PDE4D、WWP1)は、ヘビではグリーンアノールと比較して発現が異なっていた(図3C)。このうち、WWP1はPSGとして同定され、ヘビ肺で高発現しているSFTPCはSD-CNEと関連し、TITF1、SAV1、SEC24B、PDE4DはSSSV関連遺伝子として同定された。肺嚢形成に重要な転写因子である甲状腺転写因子1(TITF1)の制御領域、
81
には、SSSV挿入によるHOXA5結合部位が追加されていた(図S4L)。さらに、SAV1(protein salvador homolog 1)は、肺上皮細胞の分化を制限することによって肺の大きさを調節している82、
82
核内因子I-B(NFIB)は肺の繊毛細胞の分化を促進する。
79
さらに、2つの呼吸ガス交換制御因子(NR4A2とPHX2A)が、それぞれPSGとREGとして同定された。このように、左右非対称な肺の発達は、肺の機能を維持するための繊毛細胞や上皮細胞の分化だけでなく、強化された肺嚢の進化も含め、ヘビの肺の呼吸機能に変化をもたらした可能性がある。
感覚系の進化
四肢の喪失に加え、ヘビの視覚や聴覚の刺激を知覚する能力が低下しており、こうした変化は化石的な生活様式と関連している。
83
,
84
ヘビは捕食に成功した動物であり、嗅覚系を通じて獲物からの化学的な合図を感知する鋭い能力を持っている。ヘビは様々な環境に生息しており、その生活様式は化石生活から樹上生活まで幅広く、視力の低下や体温受容の強化など、多様な環境での適性を高めるために様々な適応を進化させてきた。これらの適応の遺伝的構造を理解することは、ヘビにおける感覚系の進化や、新しい形質の進化の根底にあるプロセスについての理解を深めることになる。
視覚の適応
現存するすべてのヘビは、他の表層生物である扁形類と比較して明確な視覚系を持っている。ヘビの初期進化における眼の減少は、視覚機能が低下した他の哺乳類で観察されたものと同様である。
85
,
86
トカゲと比較すると、ヘビの眼球には大きな毛様体筋、強膜骨、強膜軟骨がなく、円錐乳頭、環状パッド、前パッドも小さい。
86
これらの変化は、ヘビが網膜に焦点を合わせる能力を弱めていることを示唆している。
86
比較ゲノム解析によると、視細胞関連コーディング遺伝子(OPN4A、OPN3、OPSP、OPSB、GUC1A、CRY1、CRY2、GRK1など)は、調査したすべてのヘビ種で失われていることがわかった(図4A;表S3)。さらに、光刺激応答に重要な興奮性アミノ酸トランスポーター2(SLC1A2)遺伝子も欠損していた、
87
および3つのβクリスタリン遺伝子(CRBB3、CRBB1、CRBA1)は水晶体の発達に重要である、
88
も欠失した。15の視細胞維持および視細胞成分関連遺伝子の発現は、グリーンアノールの眼と比較して、ヘビの眼で有意に上昇した(図4A)。このうち、ESRRB遺伝子には2つのSSSV欠失があり(表S3)、6つの遺伝子(PDE6G、AGTPBP1、CLTCの2コピー、MKKS、NXNL1)はSD-CNEに囲まれていたことから、ヘビにおいてこれらの遺伝子の発現を制御する調節エレメントが進化した可能性が示された。これらの発見は、初期のヘビが明るい陸上生息地に侵入する前に、光量の少ない化石時代を過ごした可能性を示している。もしそうであれば、眼の構造と機能は明るい生息地に適応したに違いない。
図4ヘビ特有の感覚器進化に関連するゲノムの特徴。PSG、REG、新しく進化した遺伝子、SD-CNE関連遺伝子、失われた遺伝子、SSSV関連遺伝子を強調表示。
キャプションの全文を表示
大きな画像を見る
高解像度画像のダウンロード
我々は、5つの眼球発生関連遺伝子(FAT1、TWIST2、CC2D2A、CACNA1C、FBN1)のCDSに位置する5つのSSSV(1つの挿入と4つの欠失)を同定したが、そのうちCC2D2Aは32のヘビ特異的アミノ酸残基の欠失を示した(図S4M)。CC2D2Aのノックアウトは小眼症をもたらすことが報告されている。
89
ここで、30個のSSSV(14個の欠失と16個の挿入)が、22個の眼球発達関連遺伝子(例えば、RP1、AKT3、FRIZZLED-5、KERA、BFSP2、CRYBG3、RORB、SDK2、CPAMD8)の潜在的制御領域内に位置していた(図S3G;表S3)。さらに、レッドコーンスネーク胚の眼(10、30、50 dpo)のトランスクリプトームの塩基配列を決定したところ、SSSVを持つ5つの眼球発達関連遺伝子(CLIC4、MAN2A1、CRYBG3、GRHL2、CPAMD8)が、ヒト胚のそれと比較してヘビでは有意に発現が低いことがわかった。SSSVがヘビの眼球発生過程でこれらの遺伝子の発現を変化させたと考えられる(図S5A)。さらに、コード遺伝子の周辺に位置するSD-CNEは、眼の発達に関連するMGI用語において有意に濃縮されている(図S3D)。これらの結果は、SSSVとSD-CNEがヘビの目の形態の進化に関与している可能性を示唆している。
図S5ヘビの感覚系のゲノム進化(図4と5に関連
キャプションの全文を表示
大きな画像を見る
高解像度画像のダウンロード
陸上脊椎動物の目は、まぶたによって乾燥から守られている。しかしヘビでは、まぶたは角膜の上で動かない透明な鱗(メガネ)のように融合している、
41
,
86
まばたきができない。その結果、SD-CNEにアノテーションされたコーディング遺伝子は、まぶたの発達に関連するMGI用語に富んでいることが示された(図S3D)。まぶたの発達の重要な制御因子であるヒストン-リジンN-メチルトランスフェラーゼ2C(KMT2C)では、3つのヘビ特異的アミノ酸残基の欠失とその近傍の1つのSD-CNEが観察された(図S5B)、
90
このタンパク質の機能変化が、ヘビ特有のまぶたの発達に関与している可能性を示している。これらの結果は、ヘビにおける融合したまぶたの進化を支える遺伝的メカニズムの可能性に光を当てている。
低周波聴覚
ヘビは外耳、中耳、鼓膜を失っており、これらは音のエネルギーを集めて伝達するために必要な精巧な構造である。ヘビは本当に耳が聞こえないわけではないが、音の周波数帯域は50~1,000Hzに限られている、
41
これは人間(20~20,000 Hz)やトカゲ(100~7,700 Hz未満)よりもはるかに狭い。
91
,
92
しかし、ヘビでは中耳の骨と顎の骨がつながっているため、基質の振動に鋭敏に反応する(図4B)、
93
ヘビにおける単毛細胞型は、振動を感知する能力を高めるために内耳に機能的な変化が起こったことを示唆している。
94
,
95
その結果、耳の発生に重要な役割を果たす調節因子の進化速度が速くなり、多くのコード遺伝子(FGFR1、OTX1、MAFBなど)がSD-CNEに囲まれていることが示された(図4B)。また、音の知覚を制御するオトスピラリン(OTOS)も見つかった、
88
がヘビで失われていることもわかった(表S3)。このタンパク質のダウンレギュレーションは、ヒトでは有毛細胞の変性と難聴を引き起こす、
96
このタンパク質の欠如が有毛細胞の形態と聴覚に影響を与える可能性があることを示している。さらに、音の知覚に関連する3つのREG(ASIC2、POU3F4、CCDC50)と4つのPSG(ASIC2、S26A4、TMIE、GRM7)の発現レベルは、内耳ではアップレギュレーションか正常である(図4B)。すべてのSSSVのうち、合計31個が耳の発達に関連する遺伝子(PDZD7、OTOP1、MYO15A、MCOLN3など)の潜在的な制御領域に位置している(表S3)。PDZD7は聴覚受容体細胞の発生と音の知覚に重要な役割を果たしており、その欠損はマウスの難聴を引き起こす。
97
ここで我々は、SSSVを含むPDZD7の制御領域にEBF1結合部位を同定した(図S5C)。EBF1は内耳の発達において重要な転写因子である、
98
新たに進化した結合部位は、ヘビの内耳発生におけるPDZD7の発現を変化させる可能性がある。今回の観察から、遺伝子の欠損と新たな制御因子の進化が、内耳の特殊化と低周波聴覚に寄与している可能性が示唆された。
嗅覚と味覚
ヘビは視力や聴力に乏しいにもかかわらず、嗅覚系が発達しているため、多くの生息地で優れた捕食者となっている。エンリッチメント解析により、VMN2R26、VMN2R1、OR14A16、OR14J1など、ヘビで新たに進化した遺伝子の多くが嗅覚受容体活性に関与していることが確認された(図S3E)(表S3)。VMN2R26の1コピーはヘビに共有され、アウトグループはREGであった(表S3)。嗅覚シグナル増幅器UBR3
99
もヘビで急速な進化を示した。このようなゲノム上の革新が、ヘビ特有の嗅覚システムの進化の根底にあるのかもしれない。
ヘビは獲物を丸呑みし、体格に比して大きな獲物を消費する、
41
ヘビは獲物を丸呑みし、体格に比して大きな獲物を摂取する。そのため、獲物を感知するための味覚系の強化や、大きな獲物を消化するための効率的な消化器系の進化が促進されたのかもしれない。進化解析の結果、味覚伝達に関与するいくつかのREG(すなわち、TAS2R40、5HT1A、GNG13)が明らかになり、5HT1AはPSGであることがわかった。プリン作動性神経伝達を制御する遺伝子であるENTPD2は、部分的に塩味の味覚伝達に関与しているが、PSGであった(図4C)。トランスクリプトーム解析から、TAS2R40を除くすべての味覚遺伝子は、少なくとも3つの異なるヘビ科の舌と脳で発現していることが示され、これらの受容体のほとんどがまだ機能していることが示唆された(図S5D)。PKD2L1、TAS1R2s、TAS1R3も2つの異なる家系のヘビの舌で発現している(図S5D)。ヘビに味覚があるかどうかは不明である。
100
,
101
しかし、我々の統合データ解析から、ヘビの祖先は味覚受容体を持っており、おそらく味覚が強化されていたことが示唆される。
特殊化したアリ食への適応と赤外線感知
盲蛇のアリ食
盲目のヘビは非常に特殊な穴掘りヘビで、頭部は小さく、目や歯はかなり退化しており、特殊な食事(アリやアリの卵)をとる。
3
,
4
,
84
,
102
ディアード盲蛇の眼を2種の視覚指向性捕食種(アジアヴァインスネーク、キールスラッグスネーク)と比較形態学的に解析した結果、ディアード盲蛇の網膜、水晶体、角膜、メガネはほぼ融合しており、水晶体は緩く組織化されていることがわかった(図5A、S5E)。
図5盲目ヘビと赤外線に敏感なヘビの進化
キャプションの全文を表示
大きな画像を見る
高解像度画像のダウンロード
これらの形質を支える遺伝的特徴を理解するために、比較ゲノム解析とトランスクリプトーム解析を行ったところ、調査した2匹の盲目ヘビでは41個の遺伝子(例えば、RPGRIP1、RBP3、ABCA4、PRDM1)が失われていることがわかった(図5B;表S4)。これらの遺伝子は、眼の光受容体の発達と視覚知覚に関連するGO用語で有意に濃縮されている(図5B;表S4)。さらに、12個の視覚知覚関連遺伝子の発現が、ディアーズ盲ヘビでは4種の非盲ヘビと比較して有意に低下していた(図5B)。また、盲蛇のSD-CNE(BSD-CNE)は39の眼球発達関連遺伝子の潜在的制御領域に位置していることがわかった(図S5F)。遺伝子の後退と急速に進化する制御エレメントは、これらのヘビにおける「盲目」の進化を遺伝学的に説明する可能性がある。
盲目のヘビは目の機能が低下しているが、嗅覚系が発達しているため、コミュニケーション、繁殖、採食のためにフェロモンの痕跡を追うことができる。
103
進化学的解析の結果、側嗅路の発達を制御する遺伝子(SLIT2)が
104
はPSGであることがわかった(表S4)。さらに、BSD-CNE関連遺伝子は嗅球の発達に濃縮されている(図S5F)。これらの結果は、失明によって増強された嗅覚の遺伝的基盤に関する洞察を与える。
さらに、多細胞生物の成長に関与するREGとBSD-CNE関連遺伝子は、歯胚形成と骨の発達に関連するGOタームに富んでおり、この両者は盲目のヘビの小さな頭と歯の減少を説明する可能性がある(図5C)。アリキチンの消化に関しては、キチンやキトトリオースの分解酵素として知られる酸性哺乳類キチナーゼ(CHIA)コード遺伝子のGH18ドメインに2つの陽性選択部位があった、
105
,
106
が検出された(図5D;表S4)。さらに実験を行ったところ、CHIAの酵素活性はビルマニシキヘビよりもディアーズブラインドヘビで約40%高いことが示された(図5D)。このことから、食虫性哺乳類で観察されているように、この2つの置換がアリやアリの卵のキチンの消化向上に寄与している可能性が示唆された。
107
,
108
ヘビにおける赤外線感受性の収斂進化
ヘビの3つの系統-マムシ、ニシキヘビ、イノシシ-は、三叉神経を持つ特殊な温度感受性赤外線感知器官(例えば、ピット器官や唇孔)を持つことが知られている。
5
これらの捕食動物は、TRPA1遺伝子が関与する赤外線を感知する。
109
進化解析の結果、これら3つの系統には、熱反応と痛みの検出に関与するいくつかのPSGとREGが含まれていることが明らかになった(図5E)。TRPA1に加え、熱反応の必須アクチベーターであるMAP2K7、
110
は、ニシキヘビ、ボア、マムシで急速に進化した。さらに、比較トランスクリプトーム解析により、TRPV4が他の組織と比較してヒメツメクサのピット器官で高発現していることが確認された(図S5H)。さらに、ISL1は三叉神経の発達に極めて重要である、
111
ボアコンストリクターとビルマニシキヘビの両方で急速に進化した(表S5)。コーディング遺伝子に加えて、赤外線感受性のヘビにおけるノンコーディングエレメントの進化をさらに調べた。赤外線感受性のSD-CNEはPMP22とNFIBの周辺に存在し(図S5I)、これらはヒメヘビのピット器官でも高発現していた(図5E)。PMP22は熱に対する細胞応答に重要な役割を果たしている、
112
NFIBは三叉神経の主感覚核で重要な役割を果たしている。
113
これらの結果は、赤外線知覚にはコード化要素と非コード化要素の両方が重要な役割を果たしている可能性を示している。
考察
高品質なヘビゲノムの構築は、様々なヘビの表現型の進化を支える遺伝的基盤の理解を深める可能性を秘めている。我々の進化的・比較的マルチオミクス解析により、ヘビの特定の形質と相関する多くの遺伝子変異が明らかになった。さらに、発生生物学の今後の研究のための候補遺伝要素や標的遺伝要素もいくつか見つかった。リファレンス品質のゲノム配列はもちろんのこと、本研究の結果は、ヘビの進化と多様化に関する洞察を提供し、爬虫類生物学と発生生物学の知識を深めるものである。
研究の限界
ゲノムにおける遺伝的変化の特徴を明らかにすることを目的とした研究は、特にヘビのような非モデル種では、ある変化が形質の対応する変化に直接関与しているかどうかを決定する上で大きな課題に直面する。第一に、形質の変異は異なる遺伝的変化によって決定される可能性があり、それが主たる原因を特定することを難しくしている。第二に、機能実験によって候補となる変化を絞り込むことができるが、ヘビやトカゲではそのような実験を行うことが難しい。CRISPR-Cas9を用いて、モデル生物で自然発生する変異を正確にシミュレートする実験を試みることもできたが、このような実験は時間がかかり、特に遠縁の生物種間で遺伝的背景に大きな違いがある場合には信頼性に欠けることがある。このような制約があるにもかかわらず、本研究ではヘビの表現型の進化的変化の根底にある可能性のある遺伝的変化をいくつか同定することができた。本研究で得られた知見は、ヘビ、爬虫類、脊椎動物の進化と発生に関する様々な側面の解明を目指した今後の研究に役立つであろう。
STAR★方法
主要資源表
試薬またはリソースリソース識別子生物学的サンプル四川ラットスネーク本論文N/ARed cornsnake本論文N/AHong Kong dwarf snake本論文N/ARed-tailed pipe snake本論文N/ATartar sand boa本論文N/APlumbeous water snake本論文N/ACommon mock viper本論文N/AAfrican house snake本論文N/ABlack-. を持つヘビ本紙N/AKeled slug snake本紙N/ADiard's blind snake本紙N/AShedao island pitviper本紙N/AZong's odd-scale snake本紙N/ASunbeam snake本紙N/APtyas dhumnades本紙N/ACritical commercial assaysGenomic kit (Cat#13343)QIAGENN/AUltra II End Repair/dA-. テーリングキット(Cat# E7546)NEBNextN/ARNeasyミニキットQIAGENN/AUltra™ RNA Library Prep Kit for IlluminaNEBNextN/ARNA Nano 6000アッセイキットIlluminaN/ATruSeq PE Cluster Kit v3-cBot- HSIlluminaN/ATRIzol reagentInvitrogenN/ATRIzol reagentAmbionN/AT7 Ultra KitAmbionN/AMEGAclearTM KitThermoFisher ScientificN/AChitinase Assay Kit (ADS-302-F)Jiangsu Kete BiotechnologyN/AMEGAshortscript KitThermoFisher ScientificN/ADeposited dataBurmese python (Python bivittatus) genome assemblyCastoe et al.
11
NCBI: GCA_000186305.2Boa constrictor(ボアコンストリクター)ゲノムアセンブリKajitani et al.
114
http://platanus.bio.titech.ac.jp/platanus-assembler/platanus-1-2-1Xizang 温泉ヘビ(Thermophis baileyi)ゲノムアセンブリLi et al.
12
NCBI: GCA_003457575.1Xizang hot-spring snake (Thermophis baileyi) ゲノムアセンブリYan et al.
14
National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBJWY00000000Common garter snake (Thamnophis sirtalis) ゲノムアセンブリーVertebrate Genomes ProjectNCBI: GCF_009769535.1コモンガータースネーク(Thamnophis sirtalis)ゲノムアセンブリMcGlothlin et al.
115
NCBI: GCA_001077635.2アジアツルヘビ(Ahaetulla prasina)ゲノムアセンブリTang et al.
116
NCBI: ASM2864084v1ショーウミヘビ(Hydrophis curtus)ゲノムアセンブリPeng et al.
16
https://doi.org/10.6084/m9.figshare.11391606.v5Eastern ブラウンスネーク(Pseudonaja textilis)ゲノムアセンブリUniversity of New South WalesNCBI: GCA_900518735.1インドコブラ(Naja naja)ゲノムアセンブリSuryamohan et al.
15
NCBI: GCA_009733165.1キングコブラ(Ophiophagus hannah)ゲノムアセンブリVonk et al.
10
NCBI: GCA_000516915.1メインランドタイガースネーク(Notechis scutatus)ゲノムアセンブリUniversity of New South WalesNCBI: GCA_900518725.1ウェスタン・ラトルスネーク(Crotalus viridis viridis)ゲノムアセンブリSchield et al.
13
NCBI: GCA_003400415.2Brown spotted pitviper(Protobothrops mucrosquamatus)ゲノムアセンブリAird et al.
117
NCBI: GCA_001527695.3Many-banded krait(Bungarus multicinctus)ゲノムアセンブリZhang et al.
118
China National GeneBank DataBase: CNA0045869Five-pacer viper (Deinagkistrodon acutus) ゲノムアセンブリYin et al.
9
https://ftp.cngb.org/pub/gigadb/pub/10.5524/100001_101000/100196/Argentine オオテグー(Salvator merianae)ゲノムアセンブリRoscito et al.
25
https://bds.mpi-cbg.de/hillerlab/TeguGenomeData/Yangtze アリゲーター(Alligator sinensis)ゲノムアセンブリWan et al.
28
NCBI: GCA_000455745.1ヨーロッパガラストカゲ(Pseudopus apodus)ゲノムアセンブリWang et al.
119
中国国立遺伝子バンクデータベース: CNP0003553コモドドラゴン(Varanus komodoensis)ゲノムアセンブリLind et al.
26
https://figshare.com/projects/Data_for_Komodo_dragon_genome_paper/61271Common wall lizard (Podarcis muralis) ゲノムアセンブリAndrade et al.
120
NCBI: GCA_004329235.1ペインテッドリクガメ(Chrysemys picta bellii)ゲノムアセンブリShaffer et al.
27
NCBI: GCA_000241765.2 ビビトカゲ(Zootoca vivipara)ゲノムアセンブリUniversity of GlasgowNCBI: GCA_011800845.1サシトカゲ(Lacerta agilis)ゲノムアセンブリ脊椎動物ゲノムプロジェクトNCBI: GCA_009819535.1グリーンアノール(Anolis carolinensis)ゲノムアセンブリAlfoldi et al.
121
NCBI: GCA_000090745.2アナンロックアガマ(Laudakia sacra)ゲノムアセンブリYan et al.
122
National Genomic Data Center (https://bigd.big.ac.cn/gwh/): PRJCA010949ニワトリ(Gallus gallus)ゲノムアセンブリHillier et al.
29
NCBI: GCA_000002315.5ヒト(Homo sapiens)ゲノムアセンブリEnsemblEnsembl: GRCh38Szechwan rat snake (Euprepiophis perlacea)ゲノムアセンブリ本論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBOZS00000000.Red cornsnake (Pantherophis guttatus) ゲノムアセンブリー本論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBOZR00000000.Hong Kong dwarf snake (Calamaria septentrionalis)ゲノムアセンブリこの論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBWDL000000.アカオビハナヘビ(Cylindrophis ruffus)ゲノムアセンブリこの論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBWDM00000000.Tartar sand boa (Eryx tataricus)ゲノムアセンブリこの論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBWDN00000000.Many-banded krait (Bungarus multicinctus) ゲノムアセンブリー本論文CNGBdb: CNA0045869.Plumbeous water snake (Hypsiscopus plumbea) ゲノムアセンブリー本論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBWDO00000000.コモンモックバイパー(Psammodynastes pulverulentus)ゲノムアセンブリこの論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBWDP000000.アフリカヒメヘビ(Boaedon fuliginosus)ゲノムアセンブリこの論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBWDQ00000000.Black-tip worm snake (Leptotyphlops nigroterminus)ゲノムアセンブリこの論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBWDR00000000.Keeled slug snake (Pareas berdmorei) ゲノムアセンブリー本論文ナショナルゲノムデータセンター (https://bigd.big.ac.cn/gwh/): GWHBWDS00000000.Diard's blind snake (Argyrophis diardii)ゲノムアセンブリこの論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBWDT00000000.Shedao island pitviper (Gloydius shedaoensis)ゲノムアセンブリ本論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBWDU00000000.Zong's odd-scale snake (Achalinus jinggangensis)ゲノムアセンブリ本論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBWDV000000.サンビームヘビ(Xenopeltis unicolor)ゲノムアセンブリ本論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): GWHBWDW000000.ヒト胚眼のトランスクリプトームデータMellough et al.
123
NCBI: PRJNA384924アノテーションと解析に使用したトランスクリプトームデータ本論文National Genomic Data Center (https://bigd.big.ac.cn/gwh/): PRJCA012991Software and algorithmsfastp v0.19.4Chen et al.
124
https://github.com/OpenGene/fastpfastp v0.23.1Chen et al.
124
https://github.com/OpenGene/fastpsmrtlink v7.0Pacbiohttps://www.pacb.com/support/software-downloads/Guppy v3.2.2+9fe0a78Oxford Nanopore Technologyhttps://github.com/nanoporetech/taiyakiJellyfish v2.2.10Marçais and Kingsford
125
https://www.cbcb.umd.edu/software/jellyfish/GCE v1.02Liu et al.
126
https://github.com/fanagislab/GCEfindGSE v1.94Git-hubhttps://github.com/schneebergerlab/findGSE/GenomeScopeVurture et al.
127
http://qb.cshl.edu/genomescope/Platanus v1.2.4Kajitani et al.
114
http://platanus.bio.titech.ac.jp/platanus-assembler/platanus-1-2-4DBG2OLCYe et al.
128
https://github.com/yechengxi/DBG2OLCBowtie2 v2.3.2LangmeadおよびSalzberg
129
https://bowtie-bio.sourceforge.net/bowtie2/index.shtmlBowtie2 v2.3.4.3ラングミードとサルツバーグ
129
https://bowtie-bio.sourceforge.net/bowtie2/index.shtmlBWA v0.7.17-r1188Li and Durbin
130
https://github.com/lh3/bwaNextDenovo v1.0Hu et al.
131
https://github.com/Nextomics/NextDenovoNextPolish v1.01Hu et al.
132
https://github.com/Nextomics/NextPolishwtdbg2 v2.3Ruan and Li
133
https://github.com/ruanjue/wtdbg2HiC-Pro v2.8.0Servant et al.
134
https://github.com/nservant/HiC-Pro/JuiceboxDurand et al.
135
https://github.com/aidenlab/JuiceboxJuicer v1.5Durand et al.
135
https://github.com/aidenlab/juicer3D-DNA v180922Dudchenko et al.
136
https://github.com/aidenlab/3d-dnaLACHESISBurton et al.
137
https://github.com/shendurelab/LACHESISBUSCO v3.1.0Simão et al.
138
https://busco.ezlab.org/v3RepeatModeler v1.0.11Flynn et al.
139
http://www.repeatmasker.org/RepeatMasker v1.331Tarailo-Graovac and Chen
140
http://www.repeatmasker.org/AUGUSTUS v3.3.1Stanke et al.
141
http://bioinf.uni-greifswald.de/augustus/GeMoMa v1.6.1Keilwagen et al.
142
http://www.jstacs.de/index.php/GeMoMaStringTie v1.3.4Kovaka et al.
143
https://ccb.jhu.edu/software/stringtie/StringTie v2.0.4Kovaka et al.
143
https://ccb.jhu.edu/software/stringtie/STAR v2.6.1dDobin et al.
144
https://github.com/alexdobin/STAR/PASA v2.3.3Haas et al.
145
https://github.com/PASApipeline/PASApipeline/releasesGeneMark-ST 5.1 2014年3月Tang et al.
146
http://exon.gatech.edu/GeneMark/EVidenceModeler v1.1.1Haas et al.
145
http://evidencemodeler.github.io/TransposonPSI v1.0.0Urasaki et al.
147
http://transposonpsi.sourceforge.net/BLAST v2.7.1+Zhang et al.
148
https://blast.ncbi.nlm.nih.gov/Blast.cgiDESCHRAMBLERKim et al.
149
https://github.com/jkimlab/DESCHRAMBLERCircos v0.69-9Krzywinski et al.
150
http://circos.ca/Progressive Cactus v1.0.0Armstrong et al.
17
https://github.com/ComparativeGenomicsToolkit/cactus/OrthoFinder v2.2.7Emms and Kelly
151
https://github.com/davidemms/OrthoFinderPhastCons v1.3N/Ahttp://compgen.cshl.edu/phast/phastCons-HOWTO.htmlGERPN/Ahttp://mendel.stanford.edu/SidowLab/downloads/gerp/index.htmlBEDTools v2.25.0Quinlan and Hall
152
https://BEDTools.readthedocs.io/en/latest/ImageGPCheng et al.
153
https://416h86i955.zicp.fun/Cloud_Platform/front/#/ggplot2 v3.3.5ウィッカム
154
https://ggplot2.tidyverse.org/phyper v3.6.0https://www.r-project.org/ASTRAL-III v5.6.3Zhang et al.
18
https://github.com/smirarab/ASTRALtrimAl v1.4.rev22Capella-Gutiérrez et al.
155
http://trimal.cgenomics.orgIQ-TREE v1.6.8Nguyen et al.
156
http://www.iqtree.org/prank v.150803Löytynoja and Goldman
157
http://wasabiapp.org/software/prank/r8s v1.71Sanderson
34
https://sourceforge.net/projects/r8s/PAML 4.9iYang
158
http://abacus.gene.ucl.ac.uk/software/paml.htmlDiscoVistaSayyari
159
https://github.com/HeJian151004/DiscoVistaPoSSuM v1.3Beckstette et al.
160
https://possum.cbrc.pj.aist.go.jp/PoSSuM/HISAT2 v2-2.1.0Kim et al.
161
https://daehwankimlab.github.io/hisat2/preprocessCore v1.48.0Ben Bolstadhttp://www.bioconductor.org/packages/3.10/bioc/html/preprocessCore.htmlWGCNA v1.70.3Langfelder and Horvath
162
https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/REVIGOSupek et al.
163
http://revigo.irb.hr/Forward GenomicsHiller et al.
164
https://github.com/hillerlab/ForwardGenomicsRSEM v1.2.8LiおよびDewey
165
https://lilab-bcb.github.io/software/provean v1.1.5Choi et al.
166
https://www.jcvi.org/research/proveanTransDecoder v3.0.1Brian Haashttps://github.com/TransDecoder/TransDecoder/DETONATE v1.11Li et al.
167
https://deweylab.biostat.wisc.edu/detonate/DESeq2 v1.26.0Michael et al.
168
http://bioconductor.org/packages/release/bioc/html/DESeq2.htmledgeR v3.28.1Smyth
169
http://www.bioconductor.org/packages/release/bioc/html/edgeR.htmlTrinity v2.8.5Grabherr et al.
170
http://trinityrnaseq.github.ioCD-HIT v4.6Li and Godzik
171
http://cd-hit.orgHyPhy v2.5.20Kosakovsky et al.
172
http://www.hyphy.org/OtherSnake Multiomic Database本論文http://herpmuseum.cib.ac.cn/snake/Custom スクリプト本論文https://github.com/bioinformaticspcj/Coding_gene_function_annotation; https://github.com/bioinformaticspcj/Genomic_tools_for_snake_phylogenomics
新しいタブで表を開く
リソースの有無
連絡先
リソースおよび試薬に関する詳細情報およびリクエストは、リードコンタクトであるJia-Tang Li (lijt@cib.ac.cn)までお願いします。
材料の入手可能性
本研究では、新規の試薬は使用していない。
実験モデルと研究参加者の詳細
由来生物
すべての動物サンプルは合法的に収集され、中国科学院成都生物学研究所の動物研究倫理委員会の方針に従っている。すべての動物実験は、中国科学院成都生物学研究所動物研究倫理委員会の承認を得た(NO.CIBDWLL2018030)。ゲノム配列決定には、6種類のヘビ(四川ネズミヘビ、タルタルサンドボア、ウメベウスウォータースネーク、コモンモックバイパー、アフリカンハウススネーク、キールスラッグスネーク)のオス成体個体と、7種類のヘビ(レッドコーンスネーク、ホンコンドワーフスネーク、レッドテールパイプスネーク、ディアーズブラインドスネーク、シェダオアイランドピットバイパー、ゾンズオッドスケールスネーク、サンビームスネーク)のメス成体個体を用いた(表S1)。このうち、9匹のヘビ(四川ラットスネーク、タルタルサンドボア、ウンベウスウォータースネーク、アフリカンハウススネーク、キールスラッグスネーク、レッドコーンスネーク、ディアーズブラインドスネーク、シェダオアイランドピットバイパー、サンビームスネーク)もトランスクリプトーム配列決定に用いた(表S1)。ゲノム配列決定には、クロチビワムシヘビの成体1個体(組織しかないため性別不明)を用いた。RNA-seqのために、8匹のヘビ(Diard's blind snake、keeled slug snake、brown spotted pitviper、plumbeous water snake、Sichuan hot-spring keel-back、Asian vine snake、P. dhumnades、sunbeam snake)と1匹のトカゲ(green anole)から成体RNAサンプルを採取した。アカマダラヘビの胚(産卵後10、30、50日目)とP. dhumnadesの胚(産卵後40日目)もRNA-seq用に採取した(表S1)。PTCH1 CRISPR/Cas9編集は、野生型マウス(Mus musculus、系統:C57BL/6)を用いて行った(表S1)。四肢芽のトランスクリプトーム解析には、E11.5期のマウス胚を用いた。トランスクリプトーム解析に用いたサンプルの性別は明らかにしていない。
方法の詳細
種の選択
28のヘビゲノムを解析した。そのうち14は新たにアセンブルされたものであり(表S1)、ビルマニシキヘビのゲノムを含む14は過去に発表されたものである、
11
ボアコンストリクター
114
キングコブラ
10
ヒガシブラウンスネーク、インドコブラ
15
ショーウミヘビ
16
メインランドタイガースネーク、ニシガラガラヘビ、
13
西蔵温泉のヘビ、
12
,
14
ファイブ・ペーサー・バイパー
9
茶色い斑点のあるクサリヘビ
117
コモンガータースネーク
115
メニーバンド・クレイト
118
およびアジアツルヘビ。
116
これらのヘビ(基本ヘビと上級ヘビを含む)は、以前の研究によれば、ヘビの木に点在していた。
173
(図S1A)。種のサンプルとその出典を表S1に示す。
ゲノム配列決定
組織サンプルを14種のヘビから収集し(表S1)、DNAをQIAGEN® Genomic kit(Cat#13343, QIAGEN)を用いて抽出し、製造元の指示に従って通常の塩基配列決定を行った。ドデシル硫酸ナトリウム法(以前の研究
174
)を用いて、DNAの長さを維持するために精製工程を行わずに超長鎖DNAを抽出した。抽出した DNA の分解と汚染は、1%アガロースゲルで評価した。DNA純度はNanoDrop™ One UV-Vis分光光度計(Thermo Fisher Scientific, USA)を用いて検出した。OD260/280比は1.8~2.0、OD260/230比は2.0~2.2であった。DNA濃度はQubit® 4.0 Fluorometer (Invitrogen, USA)を用いて測定した。
オックスフォード・ナノポア・テクノロジー(ONT)ライブラリー調製とシーケンスには、1サンプルあたり合計3~4μgのDNAをインプット材料として使用した。サンプルを定性した後、PippinHTシステム(Sage Science社、米国)を用いて長いDNA断片のサイズ選択を行った。次に、NEBNext Ultra II End Repair/dA-tailing Kit (Cat# E7546)を用いてDNAのエンドリペアとdA-ライゲーション反応を行った。SQK-LSK109 Kit(Oxford Nanopore Technology, UK)のアダプターを用いてライゲーション反応を行い、Qubit® 4.0 Fluorometer(Invitrogen, USA)を用いてDNAライブラリーの濃度を測定した。DNAライブラリー(約700 ng)は、Nanopore PromethIONシークエンサー装置(Oxford Nanopore Technology, UK)を用いて、Gandomics Genome Center(武漢、中国)で塩基配列を決定した。
Pacbio(Continuous Long Reads、CLR)ライブラリーの調製と配列決定には、肝臓と筋肉から抽出したゲノムDNA(表S1)を、g-TUBE装置(Covaris、Woburn、MA)を用いて20kbの断片にせん断した。このDNAをAmpureXPビーズ(Agencourt, Beverly, MA)を用いてさらに精製し、濃縮した。得られたDNAを用いて、インサートサイズ20 kbのリードライブラリーを構築し、Sequel II SMRTセルで塩基配列を決定した。
ショートリードライブラリーの調製とシーケンス(イルミナリード)には、各ヘビサンプルについて、インサートサイズ350 bpのペアエンドライブラリーの全ゲノムショットガンシーケンスを実施した。ライブラリーはIllumina NovaSeq 6000プラットフォームを用い、標準的なメーカーのプロトコル(San Diego)に従ってシーケンスした。BGI-Shenzhenリードシーケンスでは、ゲノムDNAをセグメントに分割した。その後、200 bp以上400 bp以下の長さのDNAセグメントをライブラリーの構築に使用した。これらのライブラリーの塩基配列決定は、MGISEQ-2000プラットフォームを用い、標準プロトコールに従って行った。
ONTリードの品質管理を行うため、すべてのfast5ファイルをGuppy v3.2.2+9fe0a78を用いて"-c dna_r9.4.1_450bps_fast.cfg "でfastq形式に変換した。低品質塩基やエラーを含む配列を除去した後、クリーンなリードをゲノムアセンブリに用いた。Pacbioリードについては、Sequelのraw BAMファイルをPacBio smrtlink v7.0ソフトウェアを用いてデフォルトパラメータでFASTA形式のサブリードに変換した。イルミナおよびMGIデータについては、fastp v0.19.4
124
を使用し、低品質リードとアダプター付きリードをデフォルトパラメーターで除去した。
Hi-C生リードを得るために、各サンプル(Table S1)を以前の研究で記述された方法に従ってHi-Cライブラリー構築に使用した、
175
架橋、DpnII制限酵素によるクロマチン消化、DNAエンドマーキングとライゲーション、精製、剪断、ビオチンプルダウンアッセイを含む。構築されたHi-Cライブラリーは、Illumina NovaSeq 6000プラットフォームで150-bpペアエンドシーケンスされた。
ゲノムサイズの推定とアセンブリー
K-mer頻度に基づく方法を用いて各ゲノムのサイズを推定した。K-merはJellyfish v2.2.10を用いてカウントした。
125
を用い、-C -m 51 -s 10000000000 -t 50 のパラメータでカウントした。その結果をGenomeScopeの入力として使用した。
127
および findGSE v1.94 (https://github.com/schneebergerlab/findGSE/)の入力として使用し、ゲノムサイズを推定した。ゲノムをアセンブルするために、ゲノムアセンブラNextDenovo v1.0
131
read_cuoff=1k、seed_cutoff=25k "のパラメータでONTロングリードの自己補正を行った。次に、修正したリードをwtdbg2
133
をデフォルトパラメーターでアセンブルした。得られたコンティグをNextPolish v1.01
132
ソフトウェアを用い、デフォルトのパラメータで行った。このステップでは、ロングリードによるアライメントを3ラウンド、ショートリードによるアライメントを4ラウンド行った。次に、Hi-Cショートリード(利用可能な場合)をfastp v0.19.4を用いてフィルターし、低品質配列(品質スコア≤15)、アダプター配列、および30 bpより短い配列を除去した。残りのリードをHiC-Pro v2.8.0に入力した。
134
およびBowtie2 v2.3.2
129
に入力し、一意にマップされた有効リードを得た。最後に、LACHESIS(CLUSTER MIN RE SITES = 100; CLUSTER MAX LINK DENSITY = 2.5; CLUSTER NONINFORMATIVE RATIO = 1.4; ORDER MIN N RES IN TRUNK = 60; ORDER MIN N RES IN SHREDS = 60)を用いて、ポリッシュしたコンティグのクラスタリング、順序付け、アンカーリングを行った。
137
を有効なHi-Cデータと組み合わせて染色体レベルのアセンブリを得る。
Diardのブラインドヘビゲノムのアセンブリには、まずGenomic Character Estimator (GCE) v1.02
126
を用いてゲノムサイズを推定した。次に、以前の研究で説明したハイブリッド法を用いてゲノムのアセンブリを行った。
118
簡単に言うと、Platanus v1.2.4
114
を用いてコンティグを構築した。DBG2OLC
128
を使用し、Pacbioロングリードでコンティグを拡張した。最後にコンセンサスコンティグを取得し、HiC-Pro v2.8.0を用いてHi-Cデータと染色体スケールゲノムをアセンブルした、
134
Juicer v1.5を用いた、
135
および3D-DNA v180922を用いた。
136
Hi-Cスキャフォールディング中に検出されたアセンブリーエラーは、Juiceboxを使用してさらに視覚的に修正された。
135
ゲノム品質の評価
ゲノムアセンブリーの質を評価するために、ゲノムモードのBenchmarking Universal Single-copy Orthologs (BUSCO) v3.1と脊椎動物の系統データ(vertebrata_odb9)を使用しました。
138
(表S1)。
RNA抽出と配列決定
ゲノムアノテーションのためのトランスクリプトームエビデンスを得るために、新たに配列決定された種の36サンプル(表S1)をRNA配列決定のために収集した。RNAはTRlzol(Invitrogen)を用いて抽出し、その後RNeasy Mini Kit(QIAGEN)を用いて精製した。精製したRNAは、NEBNext® Ultra™ RNA Library Prep Kit for Illumina® (NEB, USA)を用いて、メーカーの推奨に従ってPolyA RNAシーケンスライブラリーを構築した。最後に、イルミナHiSeq 2000プラットフォームを用いてRNAシーケンスを行った。
異なる種における遺伝子の発現レベルを調べるため、グリーンアノール(系統学的にヘビに近縁)の異なる組織から194サンプルを収集した。
23
)、キールナメクジヘビ、P. dhumnades、レッドコーンスネーク、アジアンバインスネーク、サンビームスネーク、Diard's blind snake、Sichuan hot-spring keelback、brown-spotted pitviper、およびマウスの異なる組織から194サンプルを収集した。詳細情報は表S1に示す。
全サンプルの全RNAは、Trizol(Ambin社、米国)を用いて、製造元の説明書に従って抽出した。RNAの分解と汚染は1%アガロースゲルで評価した。RNA 純度の測定には NanoPhotometer® spectrophotometer (IMPLEN, CA, USA) を用い、RNA の完全性は Agilent Bioanalyzer 2100 システムと RNA Nano 6000 Assay Kit (Agilent Technologies, CA, USA) を用いて測定した。得られた全RNAは、NEBNext® Ultra™ RNA Library Prep Kit for Illumina® (NEBNext、米国)を用いて、メーカーの推奨に従ってライブラリーを構築した(Novogene Company、北京、中国)。インデックスコードされたサンプルは、メーカーの指示に従って、TruSeq PE Cluster Kit v3-cBot-HS(イルミナ)を用いてcBot Cluster Generation System上でクラスタリングした。すべてのcDNAライブラリーはNovaSeq 6000プラットフォームで150-bpペアエンドシーケンスを行った。
ゲノムアノテーション
コード遺伝子の構造アノテーションでは、相同性ベースとde novo予測の両方を用いてすべてのリピートをアノテーションした。RepeatModeler v1.0.11
139
de novo transposable element ライブラリーの構築に使用した。構築されたすべてのライブラリーは、Repbase(20170127)のリピートとともに1つのライブラリーに統合され、RepeatMasker v1.331の入力として使用された。
140
の入力として使用した。同じプロトコルを用いて、アウトグループ種のゲノム中のリピートもアノテーションした(図2)。
次に、de novo ベース、ホモロジーベース、トランスクリプトームベース(利用可能な場合、表S1)の方法で、リピートマスクアセンブリー中のタンパク質コード遺伝子をアノテーションした。AUGUSTUS v3.3.1
141
を用い、"--gff3=on --hintsfile=hints.gff --extrinsicCfgFile=extrinsic.cfg --allow_hinted_splicesites=gcag,atac --min_intron_len=30 --softmasking=1" というパラメータで de novo 予測を行った。キングコブラ、ビルマニシキヘビ、コモンガータースネーク、ブラウンスポッテッドピットバイパー、ヒガシブラウンスネーク、西蔵春蛇の全プロテオームをGeMoMa v1.6.1に入力した。
142
に入力し、相同遺伝子構造予測を行った。転写産物に基づく遺伝子予測のために、RNA-seqリードをSTAR v2.6.1dを用いてアセンブリーにマッピングした。
144
StringTie v1.3.4
143
次に、STAR v2.6.1dを用いてRNA-seqリードをアセンブリにマッピングした。次に、PASA v2.3.3を用いて、抽出した転写産物配列をゲノムにマッピングした。
145
遺伝子予測を行った。GeneMark-ST
146
もPASAで抽出した転写産物を用いて遺伝子予測を行った。3つの方法からの遺伝子予測は、EVidenceModeler v1.1.1を用いて統合された。
145
(それぞれの重み:Augustus: 1; GeMoMa: 5; PASA: 10; GeneMark-ST: 10)をパラメータとして統合した: 「(重み: Augustus: 1; GeMoMa: 5; PASA: 10; GeneMark-ST: 10)。最終的な遺伝子予測は、TransposonPSI v1.0.0によって同定されたトランスポゾン挿入遺伝子を除去して得られた。
147
(表S1)。
新たにアセンブルしたゲノムのコード遺伝子の機能をアノテーションするために、blastp v2.7.1+ を用いた
148
のNRデータベース(version:20190401)、SwissProtデータベース(version:20200709)、Kyoto Encyclopedia of Genes and Genomes (KEGG)データベース(version:20200712)に対して検索を行った(E-value ≤ 1e-5)。SwissProtに対するblastp v2.7.1+検索の結果は、idmapping.tb.gz (ftp://ftp.pir.georgetown.edu/databases/idmapping/, version:20200709)から関連するGOタームを検索するために、社内のPerlスクリプトを用いて処理された(表S1)。
染色体進化解析
ヘビにおける染色体の進化を調べるために、4匹のトカゲ(アウトグループとして使用)と16匹のヘビのゲノムを選択し(図S2A)、Shield et al.
13
まず、19のゲノムを潜在的な100-bpマーカーに分割し、blastn v2.7.1+を用いてDiard's blind snakeゲノムに対して各マーカーをアライメントした。アライメント長50bp以上のベストヒットを保持した。これらのマーカーを各染色体上で処理するには、同じディアーズブラインドのヘビ染色体との相同性を支持する少なくとも5つの連続したマーカーを必要とした。連続する5つ以上のマーカー群はそれぞれ1つの確定マーカーに統合された。次に、DESCHRAMBLER
149
を用いて、蛇類の共通祖先の核型を再構築した(図2、S2A;表S2)。最後に、Circos v0.69-9を用いて、選択した種の染色体と再構築したヘビ祖先の染色体間のシンテニーを可視化した。
150
プログレッシブカクタスアライメント
Progressive Cactus v1.0.0は、リファレンスフリーの全ゲノムアライメントソフトウェアである、
17
は、鳥類
17
および哺乳類
176
ゲノムのアライメントに使用されている。ヘビ25種とアウトグループ6種のゲノムをProgressive Cactus v1.0.0を用いてアラインメントし、全ゲノムアラインメント(WGA)を得た。ガイドツリーは、選択した種の4倍縮退第3コドン(4d)位置から作成した(「系統樹の構築」のセクションを参照)。Progressive Cactus v1.0.0ソフトウェアは、Rongjianバイオインフォマティクスビッグデータコンピューティングクラウドサービスプラットフォーム上でパラメータを指定して実行した: 最終的なhalファイルを得るために、"-binariesMode local --disableCaching --disableHotDeployment" というパラメータを付けて実行した。Progressive Cactusパッケージに実装されているhal2mafコマンド(--onlyOrthologs)は、生の31タクソンゲノムアライメントをマルチプルアライメントフォーマット(MAF)ファイル形式で生成するために使用された。最後に、Progressive CactusパッケージのmafDuplicateFilterとmafFilterの2つのコマンドをMAFファイルに適用し、デフォルトパラメータで最終的な31タクソンアラインメントを得た。
保存されたノンコーディングエレメント(CNE)の解析
PhastCons v1.3 (http://compgen.cshl.edu/phast/phastCons-HOWTO.html) (パラメーター: "expected length = 45, target coverage = 0.3, rho = 0.3") とGERP (パラメーター: "-d 0.01") (http://mendel.stanford.edu/SidowLab/downloads/gerp/index.html)の両方を使用して、31タクソンのアラインメントから進化的に保存された要素を同定した。4d-siteツリー(詳細は「系統樹の構築」のセクションを参照)をphyloFit(PhastCons v1.3パッケージ;パラメータ:-EM -precision HIGH -subst-mod REV)の入力として使用し、PhastConsが必要とする中立枝の長さを推定した。PhastConsとGERPの結果はBEDTools v2.25.0によって結合された。
152
(パラメータ:"-d 10")で結合した。信頼性の高いCNEを得るために、カスタムPerlスクリプトとBEDTools v2.25.0を用いて、アノテーションされたコード領域と重なる保存領域を持つエレメントを厳密にフィルターした。31種のCNEを同定するために、ペインテッドカメゲノムのアノテーションされたコード領域を使用した。25種のヘビのCNEを同定するために、Diard's blind snakeのコード領域のアノテーションを使用した。最後に、長さ30 bp以上のCNEを以降の解析に使用した。
SD-CNEを検出するために、我々は以前の研究で説明した方法に従った
16
に記載された方法に従って、アライメントされた祖先配列と各 種のCNE配列間の配列同一性を推定した。次に、Roscito et al.
36
SD-CNEはグローバルZスコア<0と定義した(図S3C)。
SD-CNEを、GREATの制御ドメインの概念を用いて、機能アノテーションのための潜在的な標的遺伝子に関連付けた。
177
転写開始部位の上流 5 kb、下流 1 kb の基底(プロモーター関連)ドメインを定義した。また、基底ドメインを次の遺伝子の基底ドメインまで拡張した遠位ドメイン、あるいは、どちらか一方向に最大300kbのドメインを定義した。社内のRスクリプトを用いて、超幾何学的検定(phyper v3.6.0 (https://www.r-project.org/)関数を用い、lower.tail = F)により、CNEの潜在的標的遺伝子に対して有意に濃縮されたGene Ontology (GO)またはMouse Genome Informatics (MGI) phenotype ontology termsを求めた。p値は、R v3.6.0のp.adjust関数に実装されている "Benjamini and Hochberg "法を用いて補正され、有意な濃縮の閾値は調整p値<0.05であった(図S3D)。
系統樹の構築
全ゲノム進化系統樹を推定するために、各生物種の31タクソンアラインメントからオーソログなゲノムセグメントを、社内のPerlスクリプトを用いて1つの配列に連結し、trimAl v1.4.rev22(http://trimal.cgenomics.org)を用いてトリミングした。IQ-TREE v1.6.8を用いて、合計51,302,441サイトを最尤(ML)系統樹の構築に使用した。
156
,
178
,
179
ソフトウェアを使用した: 「alrt 3000 -bb 3000 -bnni"(図S1B)。CNEを用いて構築したツリーについては、全ゲノムツリーを構築するのと同じ手順で、各生物種のCNE配列を連結し、トリミングした。その後、IQ-TREE v1.6.8ソフトウェアを用いて、"-alrt 5000 -bb 5000 -bnni "というパラメータで木を推定した(図S1E)。
オルソログ遺伝子ツリーを構築するために、まずOrthoFinder v2.2.7を使用した。
151
(デフォルトパラメータ)を用いて、25匹のヘビ、4匹のトカゲ(コモンウォールトカゲ
120
ヨーロッパガラストカゲ、コモドドラゴン、
26
およびアルゼンチンジャイアントテグー
25
ペインテッド・ガメ
27
長江ワニ
28
合計1,980の1:1シングルコピー遺伝子ファミリーが選択された。各ファミリーのコード配列(CDS)をタンパク質に翻訳し、prank v.150803を用いてアライメントした。
157
各ファミリーのCDSをタンパク質に翻訳し、prank v.150803を用いてアラインメントした。各遺伝子ファミリーのCDSを連結し、IQ-TREE v1.6.8をパラメータとしてツリー構築用のスーパー遺伝子配列を作成した: パラメータは"-alrt 10000 -bb 10000 -bnni "である(図S1C)。4d部位に基づいて木を構築するために、社内のPerlスクリプトを用いて1,980個の単一コピー遺伝子の4d部位を抽出した。合計135,405部位を連結し、IQ-TREE v1.6.8ソフトウェアを用いて、"-alrt 10000 -bb 10000 -bnni "のパラメータでML系統樹を構築した(図S1D)。
連結木(図S1B-S1E)のほとんどのノードはよく支持されたが、全ゲノム木、4d-site木、CNE木の2つのノードは100%支持されなかった。ゲノム領域間の系統的不一致を調べるため、アラインメントされたゲノムから51,302個の1kb独立ウィンドウを抽出し、全ゲノムに基づいて合体系統樹を推定した。また、各シングルコピー遺伝子を用いて系統樹を構築した。これらの系統樹はすべてASTRAL-III v5.6.3を用いて推定・構築した。
18
を用いて構築した。
ゲノムのオーソログセグメント樹と遺伝子樹のトポロジーは、それぞれ約94.40%と93.49%が連結樹と一致した。DiscoVista
159
は、ASTRAL-III合体遺伝子木を用いて不一致を要約するために使用された。
発散時間の推定
まず、31種の全ゲノム木(WGAデータセットから構築)を用いて、r8s v1.71を用いて分岐時間を推定した。
34
r8s v1.71のLangley-Fitch法を用いて、Timetree (http://www.timetree.org/)から得られた6つの時点を用いて木を較正した(図S1I)。次に、PAML v4.9iのMCMCTREEを用いて分岐時間を推定した。
158
HKY85モデルを使用し、100,000回の反復と10,000回のバーンインを行った。定常分布の収束を確認するためにプログラムを2回実行し、結果を確認した(図S1J)。
31分類群の全ゲノムアラインメントを用いて、r8s v1.71を用いて、各ヘビ系統、ヘビの共通祖先、6つのアウトグループ分類群のDNA進化率を推定した。各ヘビの体長(これを従属変数とした)を取った後、それぞれの値を対数変換したところ、ヘビの体長はDNA進化率と負の相関があることがわかった(R2 = -0.57; P < 0.03)(図S2C)。
構造変異体の同定
ヘビの最近共通祖先(MRCA)におけるヘビ特異的構造変異(SSSV)を同定するために、Progressive CactusパッケージのfindRegionsExclusivelyInGroupプログラムを用いて、31分類群のアライメントからヘビとアウトグループのMRCAの系統特異的配列を抽出した。このプログラムでは、系統特異的配列を、1)対象系統にのみ存在し、2)非対象系統にはアライメントされず、3)対象系統のMRCAの再構築ゲノムに存在する配列と定義している。
17
ヘビ系統特異的ゲノム配列(≧50 bp)をヘビ特異的ゲノム挿入、アウトグループ系統特異的ゲノム配列(≧50 bp)をヘビ特異的ゲノム欠失と定義した(図S2D)。これらの挿入と欠失はすべてSSSVである(図S3A)。SSSVと重複するレギュレーター領域(転写開始および終了部位を挟む5kb)またはCDSを持つ遺伝子をSSSV関連遺伝子と定義した(表S3)。SSSVと100bp以上重なるタンパク質コード遺伝子は、失われた遺伝子または新しく進化した遺伝子とみなし、以降の解析に用いた。CC2D2Aの立体構造はAlphaFold2パッケージを用いて予測した。
180
TITF1とPDZD7のSSSV挿入によってもたらされる結合部位は、PoSSuM Search v1.3によって予測された。
160
グリーンアノールの結合位置特異的スコアリングマトリックス(PSSM)を用いて予測した。また、Cistrome Data Browserにより、RP(regulatory potential)スコアを算出した。
181
オルソログ遺伝子の同定
低品質なゲノムアノテーションがオーソログ遺伝子解析に及ぼす悪影響を回避し、計算時間を短縮するため、ワオトカゲ、ヨーロッパガラストカゲ、コモドドラゴン、アルゼンチンジャイアントテグ、ビビトカゲ、サンドトカゲのゲノム、および21種のヘビゲノム(BUSCOスコア≥87%)のみを用いてオーソログ遺伝子の解析を行った。より信頼性の高いオルソグループを得るために、相互ベストヒット(RBH)法を用いてオルソログ遺伝子セットを確立した。これらのヘビ、ワラトカゲ、コモドドラゴン、アルゼンチンジャイアントテグ、ビビトカゲ、サンドトカゲのタンパク質は、E値≦1e-5でヨーロッパガラストカゲのタンパク質とアライメントされた。各遺伝子を代表するコード遺伝子の最長転写産物(LT)を選択した全種から抽出した。LT配列はタンパク質に翻訳された。各遺伝子対のベストヒット(E-value ≤ 1e-5)を得て、少なくとも3匹のトカゲと10匹のヘビと共有するヨーロッパガラストカゲの遺伝子ファミリーを保持した。合計17,015のオルソログ遺伝子ファミリーが得られ、1種あたり約14,926.81ファミリーが得られた(図S3H)。
新たに進化した遺伝子の同定
ヘビの祖先分岐において新たに進化した遺伝子を検出するために、まず西蔵温泉ヘビゲノムのアノテーションに基づいて、100 bp以上のヘビ系統特異的配列と重複するCDSを持つ遺伝子を選択した。その後、アウトグループ系統で見つかったオルソログ遺伝子をすべて取り除いた。無作為に選んだ4匹のヘビ(Zong's odd-scaled snake、Keeled slug snake、Red corn snake、Sunbeam snake)と無作為に選んだ6匹のトカゲ(Komodo dragon、European glass lizard、Green anole、Anan's rock agama、European wall lizard、viviparous lizard)の生のイルミナショートリード(30×カバレッジ)を、Burrows-Wheeler Aligner (BWA) v0.を用いて西蔵温泉ヘビゲノムにマッピングした。 7.17-r1188
130
を用い、デフォルトパラメータでマップした。ヘビが新たに進化した遺伝子は、以下の2つの条件を満たす遺伝子と定義した: 1)すべてのトカゲにおいて、遺伝子のCDSの長さの30%未満が少なくとも3×リードでカバーされていた、2)少なくとも3匹のヘビにおいて、遺伝子のCDSの長さの50%以上が少なくとも3×リードでカバーされていた。合計142遺伝子がこれらの基準を満たした(表S3)。
失われた遺伝子の同定
ヘビ祖先において特に失われた遺伝子を同定するために、ヨーロッパトカゲゲノムのアノテーションに従って、CDSがアウトグループ系統特異的配列の少なくとも100bpとオーバーラップする遺伝子を選択した。ヘビにオルソログがある遺伝子は、「オルソログ遺伝子の同定」で得たオルソログ遺伝子セットに従って取り除いた。次に、tblastn v2.7.1+を用いて、ヨーロッパトカゲ(アウトグループ種)の失われた遺伝子のアミノ酸配列を、選択された25のヘビゲノム全てにアラインメントした。アラインメントの長さが配列の60%以上で、同一性が70%以上の遺伝子は取り除かれた。次に、5匹のトカゲ(グリーンアノール、アナンロックアガマ、ヨーロッパオビトカゲ、コモドドラゴン、ビビトカゲ)と新たに配列決定された14匹のヘビの生のイルミナショートリードを用いて、候補遺伝子を再チェックした(表S1)。リードはBWA v0.7.17-r1188を用いてヨーロッパオナガトカゲにマッピングした。
130
BEDToolsv2.25.0を用いてカバレッジ深度を計算した(図S3B)。ヘビ特異的に失われた遺伝子は、以下の2つの基準のいずれかを満たす遺伝子であった。1)少なくとも2匹のトカゲにおいて、遺伝子のCDSの長さの30%以上が少なくとも2×リードでカバーされている遺伝子、2)すべてのヘビにおいて、遺伝子のCDSの長さの100%が0×リードでカバーされている遺伝子。ヘビ特有の失われた遺伝子の2番目のグループは、1)少なくとも2匹のトカゲにおいて、遺伝子のCDSの長さの30%以上が少なくとも2×リードでカバーされている遺伝子、2)少なくとも13匹のヘビにおいて、遺伝子のCDSの長さの50%以上が0×リードでカバーされている遺伝子であった。全体で156遺伝子がヘビのMRCAで失われたと考えられた(表S3)。
ヘビに特異的な新しく進化した遺伝子と失われた遺伝子のエンリッチメント解析は、「保存されたノンコーディングエレメント(CNE)の解析」(図S3EおよびS3F)に記載されているように、社内のRスクリプトを介して超幾何学的検定を用いてそれぞれ行われた。
ポジティブ選択遺伝子(PSG)の同定
PAMLパッケージv4.9iのcodemlプログラムを17,015の遺伝子ファミリーのそれぞれについてブランチサイトモデルで実行し、ヘビ系統の祖先枝におけるPSGの可能性を同定した。まず、OrthoFinder v2.2.7を用いてシングルコピー遺伝子を作成し、IQ-TREE v1.6.8を用いてPAML v4.9i用の27種の系統樹を構築した。次に、尤度比検定(LRT)を行い、フォアグラウンドブランチにおいて部位が正に選択されることを認める代替モデルと、部位が中立または純化選択にさらされるヌルモデルとを比較した。p値は自由度1のカイ二乗統計に基づいて計算した。p値≦0.05で、少なくとも1つの陽性部位を持つPSGが選択された(表S3)。濃縮されたMGI、GO、およびKEGG用語は、上述のように超幾何学的検定を用いて得られた(「保存された非コード要素(CNE)の解析」のセクションを参照)(表S3)。
急速に進化する遺伝子(REG)の同定
PSGの同定に用いたのと同じオルソログ遺伝子セットとツリートポロジーを用いてREGを検出した。すべての枝が同じ速度で進化するヌルモデル(model=0, NSsites=0)と、手前の枝の進化速度が異なる代替モデル(model=2, NSsites=0)を仮定するPAML v4.9iの枝モデルを用いた。この2つのモデルをLRT(df=1)を用いて識別し、p値を求めた。dS(同義置換率)>3またはω0>5の遺伝子
182
を持つ遺伝子は取り除かれ、p値<0.05で残ったものが前景枝のREGとみなされた。PSGと同じ枝でREGを検出した(上記参照)。同定されたREGと、超幾何学的検定(「保存されたノンコーディングエレメント(CNE)の解析」のセクションを参照)によって同定されたエンリッチMGI、GO、KEGGタームを表S3に示す。
緩和選択解析
HOX遺伝子はPSG同定に用いたオルソログ遺伝子セットから選択した。我々はHyPhy v2.5.20を用いて緩和選択下で遺伝子を同定した
172
,
183
を用い、デフォルトのパラメーターとPSGの同定に用いた木のトポロジーで、緩和選択下で遺伝子を同定した。各遺伝子のp値は "Benjamini and Hochberg "法で補正し、有意に緩和選択下にある遺伝子は補正p値<0.05の遺伝子とした。
ヘビ形質に関連する遺伝子のトランスクリプトーム解析
参照ゲノムとのトランスクリプトーム比較のために、生のRNA-seqリードをfastp v0.23.1を用いてパラメータでフィルターした: 「g -q 5 -u 50 -n 15 -l 150 --min_trim_length 10 --overlap_diff_limit 1 --overlap_diff_percent_limit 10"。高品質リードは、HISAT2 v2-2.1.0を用いて対応する参照ゲノムにマッピングした。
161
(デフォルトパラメーター)。StringTie v2.0.4(デフォルトパラメータ)を用いて、マップされたリード数と100万マップリードあたりのキロ塩基あたりのフラグメント数(FPKM)を計算した。遺伝子発現の違いを生物種間で比較するため、RパッケージpreprocessCore v1.48.0を用いてFPKMを分位正規化した。差次的発現遺伝子(DEGs)は、fold-change > 2およびStudent's t-test p値 < 0.05のものと定義した。また、WGCNAを実施し、選択されたサンプルの中で最も有意に関連する遺伝子を同定した。
118
). 分位正規化したFPKMをWGCNA Rパッケージv1.70.3に入力した。
162
候補遺伝子は以下の基準で同定した: モジュール固有遺伝子(ME)と遺伝子発現値間のピアソン相関(モジュールメンバーシップ、MM)≧0.8、および遺伝子発現値とサンプル間の相関(遺伝子有意性、GS)≧0.8。
P.dhumnadesの肺と椎弓の発達に関するトランスクリプトーム解析のために、生のRNA-seqリードを、上記のようにfastp v0.23.1を用いてフィルターした(「RNA抽出と配列決定」のセクションを参照)。クリーンリードはTrinity v2.8.5を用いてデノボアセンブルした。
170
パラメータ"-group_pairs_distance 230 --min_contig_length 350 --min_glue 4 "でデノボアセンブルした。未加工のTrinity転写産物をDETONATE v1.11を用いてフィルターした。
167
CD-HIT v4.6 (http://cd-hit.org)を用いて、-c 0.90パラメータで残りの転写産物の冗長性を除去した。ユニゲンの発現プロファイルを定量化するために、Trinity v2.8.5のalign_and_estimate_abundance.plスクリプトとbowtie2 v2.3.4.3を使用した。
129
およびRSEM v1.2.8
165
を使用して、リードカウントとFPKM値を取得しました。DEGはedgeR v3.28.1を用いて同定した。
169
レッドコーンスネーク(受精後10、30、50日目)とヒト胚(受精後4.7~8週目)の胚眼の比較トランスクリプトーム解析を上記と同様に行った(表S1)。以前の研究で推奨された8つのハウスキーピング遺伝子(PSMB4、VCP、CHMP2A、RAB7A、EMC7、GPI、PSMB2、VPS29)を用いた。
184
FPKM値を正規化し、遠縁種間(レッドコーンスネーク対ヒト)の遺伝子発現レベルを比較する。
ブラインドヘビの適応進化解析
パラフィン切片を用いて、ディアードの盲蛇と非盲蛇(アジアツルヘビとキールスナメクジヘビ)の違いを理解するために、目の比較形態学的解析を行った(図S5E)。
これらの適応的表現型の根底にある遺伝的基盤を探るため、2つのゲノムのコーディングエレメントとノンコーディングエレメントの進化を調べた。オルソログ遺伝子の同定」で得られたオルソログ遺伝子ファミリーから、ディアーズブラインドスネークとスレンダーブラインドスネークに共通するオルソログを含む遺伝子ファミリーを抽出した。"正選択遺伝子(PSG)の同定 "と "急速進化遺伝子(REG)の同定 "で述べたのと同じ方法で、2匹のヘビの最も共通な祖先のPSGとREGを同定した。合計171遺伝子が有意な正の選択を受けたと判定され、盲蛇祖先の432遺伝子がREGであった(表S4)。REGの有意に濃縮されたGO用語(p値<0.01)は、「保存されたノンコーディングエレメント(CNE)の解析」で上述したように、我々のカスタムRスクリプトで超幾何学的検定を用いて得られ、次にREVIGOを用いてクラスタ化された。
163
(を用いてクラスタ化した(図S5G)。新しく進化した遺伝子と失われた遺伝子の同定」に記載した方法に従って、失われた可能性のあるコーディング遺伝子を同定するために、盲蛇の最も共通な祖先ゲノムをスキャンした(表S4)。西蔵温泉ヘビゲノムのアノテーションに従って、29の非盲目爬虫類(23のヘビ、4つのトカゲ、揚子江ワニ、31分類群のアラインメントからペインテッド・カメを含む)の系統特異的配列(Progressive Cactusによって同定)と少なくとも100bpのCDSオーバーラップを示し、「オルソログ遺伝子の同定」に従って両方の盲目ヘビで失われた遺伝子は、盲目ヘビで失われた候補特異的遺伝子と考えられた。次に、tblastn v2.7.1+を用いて、西蔵温泉ヘビの失われた遺伝子のアミノ酸配列を2つのブラインドヘビゲノムにアライメントした。アラインメントの長さが配列の60%以上で、同一性が70%以上の遺伝子は取り除かれた。さらに、BWA v0.7.17-r1188を用いて、2匹の盲蛇の生のイルミナショートリードを西蔵温泉蛇のゲノムにマッピングし、残った遺伝子が失われていることを確認した。
130
をデフォルトパラメータで使用した。以下の 2 つの基準のいずれかを満たす遺伝子をブラインドヘビ特有の失われた遺伝子と定義した: 1)両方のブラインドヘビにおいて0×リードでカバーされているCDSの長さが50%以上、2)両方のブラインドヘビにおいて少なくとも2×リードでカバーされているCDSの長さが20%未満(表S4)。盲蛇の系統分岐CNEは、"Forward Genomics "分岐法を用いて検出された
164
p値<1e-5で検出された。これらは、「保存されたノンコーディングエレメント(CNE)の解析」で説明したように、潜在的な標的遺伝子と関連していた(図S5F)。
5種のヘビ(Diard's blind snake、Asian vine snake、Keeled slug snake、Xizang hot-spring snake、Western Rattlesnake)の眼RNAサンプルを用い、それぞれの種について少なくとも3つの生物学的複製を用い(表S1)、盲目のヘビにおける眼の機能低下を探索した。RNA単離、ライブラリー構築、配列決定はNovogene社(中国)が行った。リファレンスベースの転写産物アセンブリーは、Bowtie2 v2.3.4.3を用いて行った。
129
遺伝子発現プロファイルはRSEM v1.2.8を用いて決定した。
165
DEGを同定するために、まずRBHを用いて9,579個の単一コピー遺伝子のセットを構築し、全遺伝子のFPKM値を分位正規化した。Diard's blind snakeにおけるこれらの遺伝子を、Studentのt検定を用いて他のヘビにおける遺伝子と比較した(p値 < 0.05)。4つの比較のすべてで発現が低下した遺伝子を保持した。
赤外線センシングの進化解析
マムシ、ニシキヘビ、および赤外線に敏感なボアの一部は、ピット器官または赤外線受容体を持っており、赤外線信号を感知することができる。
185
これらの特異な表現型を支える遺伝的メカニズムを調べるために、まず「オルソログ遺伝子の同定」で得られたオルソログ遺伝子セットから、GOデータベースに従って220の温度感知関連遺伝子と14の三叉神経発生関連遺伝子を同定した。マムシ、ビルマニシキヘビ、ボアコンストリクターの枝の最も共通な祖先は、枝サイトモデルと枝モデル(それぞれ「正選択遺伝子(PSG)の同定」と「急速に進化する遺伝子(REG)の同定」のセクションで説明)を用いて遺伝子間のPSGとREG(表S5)を同定するためにテストされた。全生物種の祖先タンパク質配列はprank v.150803を用いて構築した。
157
マムシ(Shedao pitviper, five-pacer viper)、ビルマニシキヘビ、ボアコンストリクターのアミノ酸部位が収束している遺伝子を社内のPerlスクリプトを用いて選択した。収束部位は以下の基準に基づいて定義した: 1)対象種では同じ部位であるが、他の種および全種の最近共通祖先では異なる部位、2)PROVEANスコア<-1.7の部位。このような収束アミノ酸部位を少なくとも1つ含むTRPA1遺伝子を同定した。Forward Genomics "分岐法を用いた、
164
CNEの進化解析を行い、分岐したCNE(p値<5e-3)を同定した。その後、「保存されたノンコーディングエレメント(CNE)の解析」で述べたように濃縮解析を行った。ピット器官の機能をさらに調べるため、7匹のカワアイサの組織について比較トランスクリプトミクス解析を行った(表S1)。全RNA抽出、塩基配列決定、定量は、「RNA抽出と塩基配列決定」および「ヘビ形質関連遺伝子のトランスクリプトーム解析」の項に記載した方法に従い、ヒメハナヘビゲノムを参照として行った。DEGはDESeq2 v1.26.0で以下の基準で同定した。
168
対数変換したfold change > 2、調整p値 < 0.05。
PTCH1遺伝子の機能実験
マウスPTCH1のR946del-L947del-R948del変異を持つ点変異マウスは、Shanghai Model Organisms Center, Inc.(中国、上海)によってデザインされ、作製された。簡単に説明すると、Cas9 mRNAは、mMESSAGE mMACHINE T7 Ultra Kit(Ambion、TX、USA)を用いて、製造業者の指示に従ってin vitro転写された;mRNAは、MEGAclear™ Kit(ThermoFisher、USA)を用いて精製された。5′-GGCTGAGAAGTAAGTAGCAC-3′配列をCas9標的ガイドRNA(sgRNA)として選択し、MEGAshortscript Kit(ThermoFisher、米国)を使用してin vitro転写し、その後MEGAclear™ Kit(ThermoFisher、米国)を使用して精製した。転写されたCas9 mRNAとsgRNA、および120bpの一本鎖オリゴデオキシヌクレオチド(ssODN)をC57BL/6Jマウスの接合子に共注入した。得られたF0マウス(C57)をPCRで検証し、以下のプライマーペアを用いて塩基配列を決定した: F1、5′-CGCCCTGAATGACCTGTT-3′;R1、5′-GGAAGAGAGCACGATG-3′。予想される点突然変異を有するF0マウスを野生型(WT)C57BL/6Jマウスと交配し、F1マウスを作製した。F1マウスの遺伝子型はPCRで同定し、塩基配列決定で確認した。
1週齢のPTCH1 WTマウス(n=13)およびスネーク欠失を有するPTCH1変異マウス(n=13)を無作為に選択した。各マウスの相対体重と体長(首から尾根まで)を測定し、平均値で割って正規化した。WTマウスとPTCH1欠損マウスの値を比較するために独立スチューデントのt検定を行った。8週齢の2群(n = 6)の相対体重も同じ方法で測定した。
マウスを犠牲にした後、WTマウスとPTCH1変異マウス(n = 4)の脛骨を採取し、10%中性緩衝ホルマリンで24時間固定し、余分な軟部組織を除去した。Skyscan1276マイクロCT装置(BrukermicroCT、Kontich、ベルギー)を用い、ソース電圧55kV、ソース電流200μA、AI、0.25mmフィルター、ピクセルサイズ6μm、回転ステップ0.3度で脛骨をスキャンした。その後、NReconソフトウェア(Bruker microCT, Kontich, Belgium)を用いて、以下の設定で画像を再構成した:リングアーチファクト補正:5;スムージング:3;ビーム硬化補正:30%。海綿骨体積(BV)、組織体積(TV)、組織体積あたりの骨体積(BV/TV)、海綿骨数(Tb.N)、および海綿骨厚さ(Tb.Th)は、CTAnプログラム(BrukermicroCT, Kontich, Belgium)を用いて解析した。
成体(約9週齢)のWT PTCH1マウス(n = 8)およびPTCH1変異マウス(n = 8)を10%抱水クロラール(0.04 ml/10 g)を用いて麻酔した。高分解能in vivo X線マイクロトモグラフィー(SkyScan 1276, Bruker, Germany)を用いて、両群のマウス(n = 8)の全身のX線画像を取得した。ImageJソフトウェア(バージョンv1.53k;米国国立衛生研究所)を用いて、体長(鼻から尾根まで)、尾の長さ、右手足の全指の指骨の長さ、右手足の長さを測定した。各サンプルについて3回測定し、平均値を求めた。尾長、指骨長、右肢長の平均値を対応する体長で割って相対値を求めた。
変異マウスの遺伝子の発現が四肢の発生過程で変化しているかどうかを調べるため、WTマウスと変異マウス(各3匹)のE11.5における胚の前肢芽と後肢芽の塩基配列を決定し、トランスクリプトームデータを得た。RNA抽出、塩基配列決定、定量は、mm10マウスゲノムを参照として、「RNA抽出と塩基配列決定」および「ヘビ形質に関連する遺伝子のトランスクリプトーム解析」の項に記載した方法で行った。DESeq2 v1.26.0パッケージ
168
を使用した。
キチナーゼ酵素活性測定
ディアーズブラインドスネークとビルマニシキヘビのCHIA遺伝子のCDSを大腸菌を用いてコドン最適化した後、化学合成し、NdeI-XhoI制限部位を用いてpET-28a (+)プラスミドにサブクローニングした。その後、N末端のHISターゲットを発現を示すために配列に付加した。
組換え発現プラスミドを大腸菌BL21(DE3)に形質転換し、細胞を30μg/mlのアンピシリンを含むルリアブロス寒天培地プレートに広げ、37℃で一晩逆さまにして培養した。次に、6,000rpm、4℃で20分間遠心分離して細胞を回収し、さらに精製するために細胞ペレットを集めた。ペレットを溶解バッファーに懸濁し、超音波ホモジナイザーで破砕した。溶解液を12,000rpm、4℃で30分間遠心した。上清を回収し、80℃で30分間インキュベートした後、12,000rpm、30分間、4℃で遠心分離し、変性タンパク質を除去した。上清をニッケルチャージ(Ni-NTA)ビーズにアプライし、アフィニティー精製を行った。カラムを2容量の洗浄バッファー(8M尿素、50mM Tris、300mM NaCl、20/50mMイミダゾール、pH8.0)で2回洗浄し、続いて1容量の溶出バッファーで標的タンパク質を回収した。その後、タンパク質をゲルろ過で精製し、保存用バッファー(50 mM Tris, 300 mM NaCl, 0.1% sarkosyl, 2 mM DTT, pH 8.0)に透析し、凍結乾燥した。
ヘビ類のキチナーゼ酵素活性は、キチナーゼアッセイキット(ADS-302-F, Jiangsu Kote Biotechnology Co. 各ヘビのキチナーゼ酵素活性の測定は5回行った。グループ間のキチナーゼ活性の差はStudentのt検定を用いて評価した(図5D)。
定量化と統計解析
本研究で用いた統計手法は、図、図の説明、および方法に示した。統計解析は、Rソフトウェアv3.6.0、DEseq2 v1.26.0およびedgeR v3.28.1パッケージを用いて行った。
データおよびコードの利用可能性
この研究で作成されたすべてのアセンブリーと生のシーケンスデータは、バイオプロジェクト番号PRJCA012991のもと、National Genomics Data Centerで入手可能である。データは発表日現在、一般公開されている。アセンブルと対応するアノテーションはSnake Multiomic Databaseでも公開されている。カスタムコードはGitHubで公開されている(https://github.com/bioinformaticspcj/Coding_gene_function_annotation, https://github.com/bioinformaticspcj/Genomic_tools_for_snake_phylogenomics)。本論文で報告されたデータの再解析に必要な追加情報は、リクエストに応じてリードコンタクトから入手可能である。
謝辞
Peng ShiとYong Shao(Kunming Institute of Zoology, Chinese Academy of Sciences)のデータ解析と図の修正に関する示唆に感謝する。本研究は、中国科学院(CAS)の戦略的重点研究プログラム(XDB31000000)、中国国家自然科学基金(32220103004; 32000296)、第2回チベット高原科学探検・研究プログラム(STEP)(2019QZKK0501)、中国科学院国際パートナーシッププログラム(151751KYSB20190024)、四川省科学技術プログラム(2021JDJQ0002)の支援を受けた。
著者貢献
J.-T.L.が研究を開始し、デザインした。C.P.、D.-D.W.、J.-L.R.、Z.-L.P.、Z.M.、W.W.、Y.L.、Z.W.、C.D.、K.J.、Y.Q.が本研究を実施した。原稿はC.P.とJ.-L.R.が執筆。J.-T.L.、D.-D.W.、C.L.P.、Z.-Y.Z.が原稿を編集した。著者全員が最終原稿を読み、承認した。
利害関係
著者らは、競合する利益はないと宣言している。
補足情報
xlsxファイルをダウンロードする(.04 MB)
xlsxファイルのヘルプ
表S1. 図1およびSTAR Methodsに関連する、収集したサンプル、シーケンス、アセンブリー、アノテーション、系統解析、トランスクリプトーム解析の基本情報
.xlsxのダウンロード (.01 MB)
xlsxファイルのヘルプ
表S2. 図2およびS2に関連する、蛇類の原始染色体とゲノム変異率
ダウンロード .xlsx (.12 MB)
xlsxファイルのヘルプ
表S3. 図3および図4に関連する、ヘビの最近の共通祖先におけるコーディング遺伝子の進化解析
ダウンロード .xlsx (.05 MB)
xlsxファイルのヘルプ
表S4. 図5およびS5に関連する、盲蛇における失われた遺伝子、正選択遺伝子(PSG)、および急速に進化する遺伝子(REG
ダウンロード.xlsx (.02 MB)
xlsxファイルのヘルプ
表S5. 図5に関連する赤外線感受性のヘビの進化解析
参考文献
ツーク G.R.
ヴィット L.
コールドウェル J.P.
Herpetology: an Introductory Biology of Amphibians and Reptiles.
Elsevier/Academic Press, 2001
記事で見る
Google Scholar
Uetz P.
爬虫類データベース。
(ハンブルク動物博物館)
http://reptile-database.org/
日付:2022年
記事で見る
グーグル・スカラー
アビラ R.W.
フェレイラ V.L.
ソウザV.B.
ブラジル中央部の半落葉樹林におけるブラインドスネークTyphlops brongersmianus(Typhlopidae)の生物学。
Herpetol. J. 2006; 16: 403-405
記事で見る
Google Scholar
水野哲也.
小島康之.
シロアリの獲物の首を切るブラインドスネーク.
J. Zool. 2015; 297: 220-224
論文で見る
グーグル・スカラー
ゴリス R.C.
ヘビの赤外線器官:視覚の不可欠な部分。
J. Herpetol. 2011; 45: 2-14
論文で見る
スコパス (61)
クロスリファレンス
グーグル学者
ダルバニヤン F.
モザファリK.
リュー L.
シャルマ P.
ヘビの赤外線視覚を支えるソフトマター力学とそのメカニズム。
Matter. 2021; 4: 241-252
記事で見る
要旨
全文
全文PDF
グーグル・スカラー
マーグレス M.J.
ラウツソーR.M.
ストリックランド J.L.
メイソン A.J.
シュレーマー T.D.
ホフマン E.P.
スティアーズ E.
エルズワース S.A.
ナイストローム G.S.
ホーガン M.P.
ほか
タイガー・ガラガラヘビのゲノムから、単純な毒の表現型の根底にある複雑な遺伝子型が明らかになった。
Proc. Natl. Sci. USA. 2021; 118e2014634118
論文で見る
グーグル奨学生
マイヤーズ E.A.
ストリックランド J.L.
ロートソー R.M.
メイソン A.J.
シュレーマー T.D.
ナイストロム・G.S.
ホーガン M.P.
ユセフ S.
ロキータ D.R.
パーキンソン C.L.
ゲノムアセンブリーにより、フィア毒蛇(Azemiops feae)の毒の進化における系統特異的遺伝子重複の役割が明らかになった。
Genome Biol. ゲノム生物学(Genome Biol.
論文で見る
グーグル奨学生
Yin W.
Wang Z.-J.
Li Q.-Y.
Lian J.-M.
Zhou Y.
Lu B.-Z.
Jin L.-J.
Qiu P.-X.
Zhu W.-B.
Zhu W.-B.
他。
ヘビの遺伝子とゲノムの進化的軌跡を五味子毒蛇の比較解析から明らかにした。
Nat. Commun. 2016; 7: 13107
論文で見る
グーグル スカラー
フォンク F.J.
ケースウェル N.R.
ヘンケル C.V.
ハイムバーグ A.M.
ヤンセン H.J.
マクレアリーR.J.
ケルクカンプ H.M.
ヴォス R.A.
ゲレイロ I.
カルヴェテJ.J.
他
キングコブラゲノムが明らかにしたヘビ毒システムにおけるダイナミックな遺伝子進化と適応。
Proc. Natl. Sci. USA. 2013; 110: 20651-20656
論文で見る
スコープス (327)
パブコメ
クロスフィルム
グーグル奨学生
カストー T.A.
デ・コーニングA.P.J.
ホールK.T.
カード D.C.
シールド D.R.
フジタ・M.K.
ルッジェーロ R.P.
デグナー J.F.
ダザ J.M.
Gu W.
他。
ビルマニシキヘビのゲノムから、ヘビにおける極端な適応の分子的基盤が明らかになった。
Proc. Natl. Acad. Sci. USA. 2013; 110: 20645-20650
論文で見る
スコープス (202)
PubMed
クロスフィルム
グーグル奨学生
Li J.-T.
ガオ Y.-D.
Xie L.
Deng C.
Shi P.
Guan M.-L.
Huang S.
Ren J.L.
Wu D.-D.
D.-D. Ding L.
他。
外温性ヘビにおける高地適応の比較ゲノム学的研究。
Proc. Natl. Acad. Sci. USA. 2018; 115: 8406-8411
論文で見る
スコープス (66)
PubMed
クロス
グーグル奨学生
シールドD.R.
カードD.C.
ヘールズN.R.
ペリー B.W.
パスケシG.M.
ブラックモン H.
アダムス R.H.
コービン A.B.
スミス C.F.
ラメシュ B.
他
染色体の起源と進化、用量補償、ヘビにおける毒調節の基礎となるメカニズム。
ゲノム研究 2019; 29: 590-601
論文で見る
スコープス (69)
PubMed
クロス
グーグル奨学生
ヤン C.
Wu W.
Dong W.
Zhu B.
Chang J.
Lv Y.
Yang S.
Li J.T.
温泉ヘビの温度馴化と寒冷応答の収束。
Innovation (Camb). 2022; 3: 100295
論文で見る
Google Scholar
スルヤモハン K.
クリシュナンクッティ S.P.
ギロリーJ.
ジェビット M.
シュレーダー M.S.
ウー M.
クリアコース・B
マテュー O.K.
ペルーマル R.C.
コルーダロフI.
他。
インドコブラの参照ゲノムとトランスクリプトームにより、毒毒素の包括的同定が可能になった。
Nat. Genet. 2020; 52: 106-117
論文で見る
スコープス (80)
PubMed
クロス
グーグル奨学生
Peng C.
任 J.-L.
Deng C.
Jiang D.
Wang J.
Qu J.
Chang J.
Yan C.
Jiang K.
Murphy R.W.
et al.
ショーウミヘビ(Hydrophis curtus)のゲノムから海洋環境への二次適応が明らかになった。
Mol. Biol. Evol. 2020; 37: 1744-1760
論文で見る
Google Scholar
アームストロング J.
ヒッキー G.
ディーカンス M.
フィデス I.T.
ノバック A.M.
デラン A.
ファン・Q.
Xie D.
Feng S.
スティラー J.
他
プログレッシブカクタスは千ゲノム時代のマルチゲノムアライナーである。
Nature. 2020; 587: 246-251
論文で見る
スコープス (90)
PubMed
クロス
グーグル奨学生
張 C.
ラビーM.
サヤリ E.
ミララブ S.
ASTRAL-III:部分的に分解された遺伝子樹からの多項式時間種樹再構築。
BMC Bioinformatics. 2018; 19: 153
論文で見る
グーグル・スカラー
フィゲロア A.
マッケルビー A.D.
グリズマー L.L.
ベル C.D.
レイルヴォー S.P.
現存するヘビの種レベルでの系統樹と新しいコルボリッド亜科・属の記述。
PLoS One. 2016; 11e0161070
論文で見る
グーグル・スカラー
ザヘル H.
マーフィー R.W.
アレドンドJ.C.
グラボスキーR.
マチャド-フィーリョ P.R.
マーロー K.
モンティネリ G.G.
クアドロス A.B.
オルロフ N.L.
ウィルキンソンM.
他
ヘビ類(Squamata: Serpentes)の大規模分子系統学、形態学、分岐時間推定、および化石記録。
PLoS One. 2019; 14e0216148
論文で見る
グーグル・スカラー
Hsiang A.Y.
フィールド D.J.
ウェブスター T.H.
ベールケ A.D.
デイビス M.B.
ラシコ R.A.
ゴーティエ J.A.
The origin of snakes: revealing the ecology, behavior, and evolutionary history of early snakes using genomics, phenomics, and the fossil record.
BMC Evol. Biol.
論文で見る
スコープス (146)
PubMed
クロス
グーグル奨学生
クライン C.G.
ピサニD.
フィールドD.J.
レイキンR.
ウィルス M.A.
ロングリッチ N.R.
白亜紀-古第三紀におけるヘビの進化と分散。
Nat. Commun. 2021; 12: 5335
論文で見る
グーグル奨学生
Zheng Y.
Wiens J.J.
系統発生学的アプローチとスーパーマトリクスアプローチを組み合わせ、52遺伝子、4162種に基づく扁形爬虫類の時間較正系統樹(トカゲとヘビ)。
Mol. Phylogenet. Evol. 2016; 94: 537-547
論文で見る
スコープス (408)
PubMed
クロス
グーグル奨学生
スコセC.R.
地球史アトラス(テキサス大学アーリントン校地質学部)。
2001
記事で見る
Google Scholar
ロスキートJ.G.
サミース K.
ピペル M.
フランソワズK.-J.
ウィンクラー S.
ダール A.
パプウトソグルーG.
マイヤーズG.
Hiller M.
テグトカゲSalvator merianaeのゲノム:イルミナ、PacBio、オプティカルマッピングデータの組み合わせによる高連続アセンブリの作成。
GigaScience. 2018; 7: giy141
論文で見る
スコープス (8)
クロスリファレンス
グーグル奨学生
リンド A.L.
ライ Y.Y.Y.
モストヴォイ Y.
ホロウェイ A.K.
イアヌッチ A.
マック A.C.Y.
フォンディ M.
オルランディーニ V.
エッカルバー W.L.
ミラノM.
他
コモドドラゴンのゲノムから、オオトカゲの心臓血管系と化学感覚系の適応が明らかになった。
Nat. Ecol. Evol. 2019; 3: 1241-1252
論文で見る
スコープス (10)
PubMed
クロス
グーグル奨学生
シェイファーH.B.
ミンクスP.
ウォーレンD.E.
シェドロック A.M.
トムソン R.C.
バレンズエラ N.
アブラミアン J.
雨宮 C.T.
バーデンホースト D.
ビガー K.K.
他
ニシガメゲノム、ゆっくりと進化する系統における極端な生理的適応の進化のモデル。
ゲノム生物学 2013; 14: R28
論文で見る
日本学術振興会特別研究員
パブコメ
クロスリファレンス
グーグル奨学生
ワン Q.-H.
パン S.-K.
Hu L.
Zhu Y.
Xu P.-W.
Xia J.-Q.
Chen H.
He G.-Y.
He G.-Y. J.
Ni X.-W.
他。
絶滅危惧種チャイニーズ・アリゲーターの潜水および感覚特性に関するゲノム解析とシグネチャーの発見。
細胞研究 2013; 23: 1091-1105
論文で見る
(87件)
PubMed
クロス
グーグル奨学生
ヒリアー L.W.
ミラーW.
バーニーE.
ウォーレンW.
ハーディソン R.C.
ポンティング C.P.
ボーク P.
バート D.W.
グローネン M.A.M.
デラニーM.E.
他
ニワトリゲノムの塩基配列と比較解析から、脊椎動物の進化についてユニークな視点が得られた。
Nature. 2004; 432: 695-716
論文で見る
日本学術振興会特別研究員
PubMed
クロス
グーグル奨学生
パスケシG.I.M.
アダムス R.H.
カードD.C.
シールド D.R.
コービン A.B.
ペリー B.W.
レイエス=ベラスコJ.
ルッジェーロ R.P.
ヴァンデウェッジM.W.
ショートJ.A.
他
鳥類と哺乳類が設定したゲノムリピートエレメント進化のパラダイムに、哺乳類が挑戦している。
Nat. Commun. 2018; 9: 2774
論文で見る
スコープス (63)
PubMed
クロス
グーグル奨学生
リーA.
マッカーシー S.A.
フェドリゴ O.
ダマスJ.
フォルメンティ G.
コレン S.
ウリアーノ=シルバ M.
チャウ W.
フンタンマサン A.
キム・J.
他。
全脊椎動物種の完全でエラーのないゲノムアセンブリーに向けて。
Nature. 2021; 592: 737-746
論文で見る
筑波大学
PubMed
クロスリファレンス
グーグル奨学生
ウェーバー C.C.
ブソーB.
ロミギエJ.
ジャービス E.D.
エレグレンH.
鳥類の塩基組成における系統間差のドライバーとしてのGCバイアス遺伝子転換の証拠。
ゲノム生物学 2014; 15: 549
論文で見る
スコープス (55)
PubMed
クロス
グーグル奨学生
デュレL.
ガルティエ N.
偏った遺伝子転換と哺乳類ゲノム景観の進化。
Annu. Rev. Genomics Hum. Genet. 2009; 10: 285-311
論文で見る
スコープス (454)
PubMed
クロス
グーグル奨学生
サンダーソン M.J.
R8s:分子時計がない場合の分子進化の絶対速度と分岐時間を推測する。
Bioinformatics. 2003; 19: 301-302
論文で見る
筑波大学
PubMed
クロスリファレンス
グーグル奨学生
陳 麗華
Qiu Q.
Jiang Y.
Wang K.
Lin Z.
Li Z.
Bibi F.
Yang Y.
Wang J.
Nie W.
et al.
反芻動物の大規模ゲノム解読により、その進化と特徴的な形質に関する知見が得られた。
Science. 2019; 364: EAAV6202
論文で見る
スコープス (173)
PubMed
クロス
グーグル奨学生
ロスキートJ.G.
サミースK.
パラ G.
ランガーB.E.
ペッツォルトA.
メビウス C.
ビックル M.
ロドリゲス M.T.
ヒラーM.
表現型の消失は、進化における遺伝子制御ランドスケープの広範な分岐と関連している。
Nat. Commun. 2018; 9: 4737
論文で見る
スコープス (38)
PubMed
クロス
グーグル奨学生
パイロンR.A.
バーブリンクF.T.
ウィーンズJ.J.
トカゲとヘビの4161種を含むSquamataの系統樹と改訂された分類。
BMC Evol. Biol.
論文で見る
日本学術振興会特別研究員
PubMed
クロス
グーグル奨学生
森永G.
バーグマン P.J.
トカゲの四足動物からヘビ類への移行における化石運動の進化。
Proc. Biol. Biol. Sci. 2020; 287: 20200192
論文で見る
Google Scholar
ピーズ M.
キーファー I.
ティーレバイン J.
Oechtering G.
Krautwald-Junghanns M.E.
Python regius、Boa constrictor、Python reticulatus、Morelia viridis、Epicrates cenchria、およびMorelia spilotaの種の健康なヘビの肺のコンピュータ断層撮影。
獣医。Radiol. Ultrasound. 2009; 50: 487-491
論文で見る
スコープス (0)
PubMed
クロス
グーグル奨学生
ファン・ソルトB.J.
メッチャーB.D.
ポエルマンR.E.
ヴェルヴストB.
フォンク F.J.
ミュラーG.B.
リチャードソン M.K.
ヘビの心肺系の発達におけるヘテロクロニーと早期の左右非対称性。
PLoS One. 2015; 10e116416
論文で見る
グーグル・スカラー
リリーホワイト H.B.
How Snakes Work: 世界のヘビの構造・機能・行動.
オックスフォード大学出版局、2014年
記事で見る
グーグル・スカラー
コーン M.J.
ティックルC.
ヘビにおける四肢の欠如と軸性パターン形成の発生的基盤。
Nature. 1999; 399: 474-479
論文で見る
ヘビ
PubMed
クロス
グーグル奨学生
アペステギアS.
ザヘル H.
頑丈な後肢と仙骨を持つ白亜紀の陸生ヘビ。
Nature. 2006; 440: 1037-1040
論文で見る
哺乳類哺乳類哺乳類哺乳類哺乳類哺乳類哺乳類哺乳類哺乳類哺乳類
PubMed
クロス
グーグル奨学生
クボンE.Z.
カムネバ O.K.
メロ U.S.
バロッツィ I.
オスターワルダー M.
マンニオン B.J.
ティシエール V.
ピックル C.S.
プラジザー・フリック I.
リーE.A.
他
ヘビの進化における四肢エンハンサーの進行性機能喪失。
Cell. 2016; 167: 633-642.e11
論文で見る
スコープス (173)
PubMed
概要
全文
全文PDF
グーグル奨学生
サンデルL.L.
サンダーソンB.W.
モイセイエフG.
ジョンソンT.
ムシェギアンA.
ヤング K.
レイ J.P.
マ J.X.
ステーリング・ハンプトン K.
トレーナー P.A.
RDH10は胚性レチノイン酸の合成に必須であり、四肢、頭蓋顔面、臓器の発生に必要である。
Genes Dev. 2007; 21: 1113-1124
論文で見る
遺伝子発現
PubMed
クロス
グーグル奨学生
牧野 進
桝谷浩之
石島 潤
矢田雄一郎
白石貴志
マウスの間葉系異形成(mes)は、patched(ptc)の最もC-末端の細胞質ドメインの欠失によって生じる。
Dev. Biol: 95-106
論文で見る
スコパス (41)
PubMed
クロス
グーグル奨学生
グッドリッチL.V.
ミレンコビッチL.
ヒギンズ K.M.
スコット M.P.
マウスのパッチド突然変異体における神経細胞運命の変化と髄芽腫。
Science. 1997; 277: 1109-1113
論文で見る
筑波大学
PubMed
クロス
グーグル奨学生
Damerla R.R.
Cui C.
ガブリエルG.C.
リュー X.
クレイジ B.
ギブス B.C.
フランシス R.
リー Y.
チャタジー B.
サンアグスティンJ.T.
他
繊毛移行帯欠損と小脳および他の繊毛症関連異常を有するジュベール症候群の新規Jbts17変異マウスモデル。
Hum. Mol. Genet. 2015; 24: 3994-4005
論文で見る
グーグル・スカラー
チダンバラム A.
ゴールドスタイン A.M.
ガイラニ M.R.
ジェラード B.
ベール S.J.
ディジョヴァンナJ.J.
ベイル A.E.
ディーン M.
白人およびアフリカ系アメリカ人の母斑基底細胞腫症候群患者におけるショウジョウバエpatched遺伝子のヒト相同遺伝子の変異。
がん研究 1996; 56: 4599-4601
論文で見る
PubMed
グーグル奨学生
パーマー K.
フェアフィールドH.
ボルゲイアS.
カーテン M.
ハッサン M.G.
ディオンヌ L.
ヨン・カルスト S.Y.
クームズ H.
ブロンソン R.T.
ラインホルトL.G.
他
頭蓋顔面異形の自然発生モデルマウスの発見と特徴づけ。
Dev. Biol.
論文で見る
(20件)
PubMed
クロス
グーグル奨学生
ファンデンブガード M.-J.H.
ドーランドM.
ビーマーF.A.
ヴァン・アムステルH.K.P.
MSX1変異はヒトの顎顔面裂および歯牙形成不全と関連している。
Nat. Genet. 2000; 24: 342-343
論文で見る
スコパス (452)
PubMed
クロス
グーグル奨学生
ヴァスターディスH.
カリンブックスN.
グトゥア S.W.
シードマンJ.G.
シードマン C.E.
ヒトMSX1ホメオドメインのミスセンス変異が選択的歯牙形成不全を引き起こす。
Nat. Genet. 1996; 13: 417-421
論文で見る
日本学術振興会特別研究員
PubMed
クロス
グーグル奨学生
セコール S.M.
ダイヤモンドJ.
ビルマニシキヘビの摂食に対する適応反応:ポンピングの前に支払う。
J. エキスポ・バイオロジー 1995; 198: 1313-1325
論文で見る
PubMed
Google Scholar
セコール S.M.
ダイヤモンドJ.
極端な生理学的調節の脊椎動物モデル。
Nature. 1998; 395: 659-662
論文で見る
スコープス (225)
PubMed
クロス
グーグル奨学生
ワン T.
リンドム E.
ヘビの消化に対する生理学的反応: 統合生理学者のためのごちそう。
Comp. Biochem. 生理学。A Mol. 統合生理学。生理学 2021; 254: 110891
論文で見る
グーグル奨学生
ペリー B.W.
アンドリュー A.L.
モスタファ・カマルA.H.
カードD.C.
シールド D.R.
パスケシG.I.M.
ペレグリーノ M.W.
マッケシー S.P.
チョードリー S.M.
セコールS.M.
他
ヘビの複数種の比較から、摂食後の腸再生の基礎となる協調的シグナル伝達ネットワークが明らかになった。
Proc. Biol. Sci. 2019; 286: 20190910
論文で見る
PubMed
グーグル奨学生
北澤 毅
貝谷博之
脊椎動物におけるモチリンとグレリンによる消化管運動制御機構。
Front. Endocrinol. 2019; 10: 278
論文で見る
スコープス (0)
PubMed
クロス
グーグル奨学生
Ryu S.
許 I.-S.
Cho E.-Y.
Cho Y.
Park T.
Yoon S.C.
Joo Y.H.
Hong K.S.
統合失調症患者における抗精神病薬誘発体重増加と60の候補遺伝子の関連研究。
Pharmacopsychiatry. 2016; 49: 51-56
論文で見る
Google Scholar
シュレーター J.C.
フェン C.M.
スモール B.C.
チャネルキャットフィッシュの摂餌量、血糖値、視床下部のNPYおよびPOMC発現における腸神経ペプチドの役割の解明。
Comp. Biochem. 生理学。A Mol. Integr. Physiol. 2015; 188: 168-174
論文で見る
グーグル・スカラー
コスタンティーニ V.J.A.
ヴィチェンティーニ E.
サッバティーニ F.M.
ヴァレリオ E.
レポーレ S.
テッサリ M.
サルトーリ M.
ミシェラン F.
メロット S.
ビフォーネA.
他
新規グレリン受容体アンタゴニストであるGSK1614343は、げっ歯類およびイヌにおいて予期せぬ摂食量および体重の増加をもたらす。
Neuroendocrinology. 2011; 94: 158-168
論文で見る
スコープス (49)
PubMed
クロス
グーグル奨学生
ベルニュ L.
リーJ.M.
チン R.G.
Auwerx J.
Reue K.
Diet1はFGF15/19腸肝シグナル伝達軸で機能し、胆汁酸と脂質レベルを調節する。
Cell Metab. 2013; 17: 916-928
論文で見る
スコパス (57)
PubMed
要旨
全文
全文PDF
グーグル奨学生
Zhang H.
エイブルズ E.T.
ポープ C.F.
ワシントン M.K.
ヒプケンス S.
ミーンズ A.L.
パス G.
セウファート J.
コスタ R.H.
ライターA.B.
ら
膵内分泌および管分化におけるHNF6の複数の時間特異的役割。
Mech. Dev. 2009; 126: 958-973
論文で見る
膵臓
PubMed
クロス
グーグル奨学生
マクドナルド R.J.
スタリーS.J.
スウィフトG.H.
似ているが非allelicな2つのラット膵臓トリプシノゲン。クローニングされたcDNAの塩基配列。
J. Biol. Chem. 1982; 257: 9724-9732
論文で見る
パブコメ
要旨
全文PDF
グーグル奨学生
ブーツ R.G.
フェルフック M.
ドンカー・クープマンW.
ストライランド A.
ヴァン・マール J.
オーバークレフト H.S.
Wennekes T.
アーツJ.M.F.G.
非リソソーム型グルコシルセラミダーゼのβ-グルコシダーゼ2としての同定。
J. Biol. Chem. 2007; 282: 1305-1312
論文で見る
スコープス(143)
パブコメ
要旨
全文
全文PDF
グーグル奨学生
トゥーミーC.B.
ケリーU.
セイバン D.R.
ボウズ・リックマンC.B.
補体因子Hによる加齢黄斑変性症様病態の制御。
Proc. Natl. Acad. Sci. USA. 2015; 112: e3040-e3049
論文で見る
スコープス (109)
PubMed
クロス
グーグル奨学生
レアル F.
コーン M.J.
ヘビにおける四肢の進化的喪失に関する発生学的、遺伝学的、ゲノム学的洞察。
Genesis. 2018; 56e23077
論文で見る
グーグル・スカラー
チョーラポイカイル S.
ウィレムスB.
ストレーレP.
ガジェフスキー M.
her1とher7変異体の解析から、体節時計モジュールの時空間的分離が明らかになった。
PLoS One. 2012; 7e39073
論文で見る
Google Scholar
ヴァン・イーデン F.J.
グラナート M.
シャッハ U.
ブランド M.
古谷経衡
ハフター P.
ハンマーシュミット M.
ハイゼンベルク C.-P.
ジアン Y.-J.
ケイン D.A.
ら
ゼブラフィッシュ(Danio rerio)における体節形成とパターン形成に影響を及ぼす変異。
Development. 1996; 123: 153-164
論文で見る
PubMed
クロス
グーグル奨学生
久米利明
姜 浩
トプチェフスカJ.M.
ホーガン B.L.
マウス翼状らせん転写因子Foxc1とFoxc2は、ともに心臓血管の発生と体節形成に必要である。
Genes Dev. 2001; 15: 2470-2482
論文で見る
筑波大学
PubMed
クロス
グーグル奨学生
スクンツ S.
マンクーB.
グエン M.-T.T.
ハスター E.
ナカヤマ A.
トゥルニエ・ラッサーブ E.
ライト C.V.E.
パクニス V.
バルティ K.
Arnheiter H.
中胚葉ホメオドメインタンパク質MEOX1の欠損は、強膜の極性を破壊し、軸索骨格の頭蓋頸部関節のリモデリングを引き起こす。
Dev. 生物学 2009; 332: 383-395
論文で見る
哺乳類
PubMed
クロス
グーグル奨学生
マンクー B.S.
スクンツ S.
ハリガン I.
グリゴリエヴァ E.
キャンディアA.
ライト C.V.E.
アーンハイター H.
パクニス V.
Meoxホメオボックス遺伝子の協調作用は、体節の形成、パターニング、分化に不可欠な遺伝経路の上流で必要である。
Development. 2003; 130: 4655-4664
論文で見る
筑波大学
PubMed
クロス
グーグル奨学生
河村明宏
越田慎一郎
土方裕之
大林章
近藤秀樹
高田真一
グルーチョ関連転写抑制因子ripply1は前中胚葉から体節への適切な移行に必要である。
Dev. Cell. 2005; 9: 735-744
論文で見る
(72件)
PubMed
要旨
全文
全文PDF
グーグル奨学生
チャン T.
コンドウ A.
細谷明彦
日立K.
雪田明彦
岡林啓一郎
中村秀樹
小沢秀行
清成弘之
道上敏明
他
Ripply2はマウス初期発生における正確な体節形成に必須である。
FEBS Lett.
論文で見る
筑波大学
PubMed
クロス
グーグル奨学生
サプリD.M.
ブリュックナー M.
キューンM.R.
ウィッテ D.P.
ロウ L.A.
マクグラス J.
コラレス J.
ポッター S.S.
左右ダイニンのATP結合ドメインの標的欠失により、左右非対称性の発生を規定する役割が確認された。
Development. 1999; 126: 5495-5504
論文で見る
PubMed
クロスフィルム
グーグル奨学生
Tian T.
Zhao L.
Zhang M.
Zhao X.
Meng A.
foxj1aとfoxj1bの両方がゼブラフィッシュ胚の左右非対称発生に関与している。
Biochem. Biochem. Res. Commun. 2009; 380: 537-542
論文で見る
スコープス (22)
PubMed
クロス
グーグル奨学生
ウォルマイヤーJ.
フランクD.
シューマークA.
ネーテ-メンヘンT.
シンドリック S.
オルブリッヒ H.
ロゲス N.T.
アプレア I.
ドハティ G.W.
ペネカンプP.
他
FOXJ1のde novo変異は、水頭症および左右非対称性のランダム化を伴う運動性繊毛症をもたらす。
Am. J. Hum. Genet. 2019; 105: 1030-1039
論文で見る
スコープス (89)
PubMed
要旨
全文
全文PDF
グーグル奨学生
ペレティエG.J.
ブロディ S.L.
リアピス・エイチ
ホワイト R.A.
ハケット B.P.
発達中の肺および腎上皮に発現するヒトのフォークヘッド/翼状らせん転写因子。
Am. J. Physiol: L351-L359
論文で見る
Google Scholar
Lyerla T.A.
ルシニアックM.E.
ボルヒャーズM.
ジャーライスG.
タン J.
大竹 P.
ノバック E.K.
Swank R.T.
Hermansky-Pudlak症候群のマウスモデルにおける肺の構造、組成、機能の異常。
Am. Am. Physiol. 肺細胞。Mol. Physiol.
論文で見る
PubMed
クロス
グーグル奨学生
Hsu Y.-C.
オシンスキーJ.
キャンベル C.E.
リトワック E.D.
ワン D.
リュー S.
バチュルスキー C.J.
Gronostajski R.M.
間葉系核因子I Bは肺成熟期の細胞増殖と上皮分化を制御する。
Dev. 生物学 2011; 354: 242-252
論文で見る
スコープス (50)
PubMed
クロス
グーグル奨学生
マフラプーM.
エネルベックS.
カールソンP.
ソニックヘッジホッグシグナル伝達の標的であるフォークヘッド遺伝子Foxf1のハプロ不全は、肺と前腸の奇形を引き起こす。
Development. 2001; 128: 2397-2406
論文で見る
PubMed
クロスレビュー
グーグル奨学生
Zhang Y.
ラスN.
ハンネンハリ S.
ワン Z.
カッポラ T.
Kimura S.
アトキナ・ヴァッサーマン E.
ルー M.M.
ビアーズ M.F.
モリシー E.E.
GATA因子とNkx因子は、in vivoにおいて組織特異的な遺伝子発現と発生を相乗的に制御する。
Development. 2007; 134: 189-198
論文で見る
(0件)
PubMed
クロス
グーグル奨学生
リー J.H.
キム T.S.
ヤン T.H.
Koo B.K.
オー S.P.
リー K.P.
オ H.J.
リー S.H.
コン Y.Y.
Kim J.M.
ら
マウス上皮組織の発生におけるWW45の重要な役割。
EMBO J. 2008; 27: 1231-1242
論文で見る
スコープス (131)
PubMed
クロスフィルム
グーグル奨学生
Yi H.
ノレル M.A.
現代のヘビの埋没起源。
Sci. Adv. 2015; 1e1500743
論文で見る
グーグル・スカラー
シモンエス B.F.
サンパイオ F.L.
ジャレッドC.
アントニアッツィ M.M.
ロエウ E.R.
ボウメーカーJ.K.
ロドリゲス A.
ハート N.S.
ハント D.M.
パートリッジJ.C.
他
視覚系の進化と祖先ヘビの性質。
J. Evol. Biol. 2015; 28: 1309-1320
論文で見る
スコープス (53)
PubMed
クロス
グーグル奨学生
ゼン D.G.
ノースカット R.G.
いくつかの扁形動物の前脳と中脳とヘビの起源との関係。
J. Morphol. 1973; 140: 135-151
論文で見る
グーグル・スカラー
カプレット C.L.
リー M.S.Y.
シャイン R.
モカニー A.
ダウンハワーJ.F.
目の解剖学から見たヘビ(Serpentes)の起源。
Biol. J. Linn. 2004; 81: 469-482
論文で見る
Google Scholar
原田知行
原田知行
渡辺正明
井上雄彦
坂川貴史
中山 N.
佐々木 聡
奥山 聡
渡瀬和男
和田和彦
他
網膜における2つのグルタミン酸トランスポーターGLASTとGLT-1の機能。
Proc. Natl. Acad. Sci. USA. 1998; 95: 4663-4666
論文で見る
スコープス (219)
PubMed
クロス
グーグル奨学生
ゴーデ P.
リブストーンM.S.
ルイス S.E.
トーマス P.D.
Gene Ontologyコンソーシアムにおける機能アノテーションの系統的伝播。
Brief. Bioinform. 2011; 12: 449-462
論文で見る
遺伝子発現
パブコメ
クロスフィルム
グーグル奨学生
ヴェレリ S.
マンジュナス S.H.
ファリスR.N.
メイ-シメラH.
ブルックス M.
フォスケット T.A.
ガオ C.
ロンゴ T.A.
リュー P.
ナガシマ K.
他
繊毛病関連遺伝子Cc2d2aは、繊毛の生合成において、母遠心点上の分層付属体の集合を促進する。
Nat. Commun. 2014; 5: 4207
論文で見る
スコープス (52)
PubMed
クロス
グーグル奨学生
リー S.
リー D.-K.
Dou Y.
リー J.
Lee B.
Kwak E.
Kong Y.-Y.
リー S.-K.
ローダー R.G.
リー J.W.
ヒストンH3リジン4メチル化酵素の標的遺伝子特異性決定因子としてのコアクチベーター。
Proc. Natl. Acad. Sci. USA. 2006; 103: 15392-15397
論文で見る
スコープス (127)
PubMed
クロス
グーグル奨学生
フェティプラスR.
内耳有毛細胞の電気的同調。
Trends Neurosci. 1987; 10: 421-425
記事で見る
要旨
全文PDF
グーグル奨学生
マンリー G.A.
トカゲの聴覚器官の構造と機能の進化。
J. Neurobiol. 2002; 53: 202-211
論文で見る
スコープス (0)
パブコメ
クロス
グーグル奨学生
クリステンセン C.B.
クリステンセン-ダルスガードJ.
ブラントC.
マドセン P.T.
ニシキヘビ(Python regius)の鼓膜外耳による聴覚:振動はよく聞こえ、音圧はあまり聞こえない。
J. Exp. Biol: 331-342
論文で見る
スコパス (0)
パブコメ
クロス
グーグル奨学生
ミラー M.R.
走査型電子顕微鏡によるカメとヘビの脳底乳頭の研究。
Am. J. Anat. 1978; 151: 409-435
論文で見る
Google Scholar
ミラー M.R.
ベック J.
トカゲとヘビにおける聴覚有毛細胞神経支配のさらなる連続透過型電子顕微鏡による研究。
Am. J. Anat. 1990; 188: 175-184
論文で見る
Google Scholar
デルプラット B.
ブーランジェ A.
ワン J.
ボードワン V.
ギトン M.J.
ヴァンテオ S.
デチェスヌ C.J.
プジョル R.
ラヴィーニュ-ルビヤールM.
ピュエルJ.-L.
他
新規内耳タンパク質であるオトスピラリンのダウンレギュレーションが有毛細胞の変性と難聴を引き起こす。
J. Neurosci. 2002; 22: 1718-1725
論文で見る
PubMed
クロスフィルム
グーグル奨学生
Zou J.
Zheng T.
レン C.
アスキュー C.
リュー X.-P.
パン B.
ホルト J.R.
Wang Y.
Yang J.
PDZD7の欠損は蝸牛有毛細胞におけるアッシャー症候群2型タンパク質複合体を破壊し、マウスの難聴を引き起こす。
Hum. Mol. Genet. 2014; 23: 2374-2390
論文で見る
スコープス (45)
PubMed
クロス
グーグル奨学生
サジャン S.A.
ルーベンスタインJ.L.R.
Warchol M.E.
Lovett M.
初期内耳発生におけるDlx5の直接下流標的の同定。
Hum. Mol. Genet. 2011; 20: 1262-1273
論文で見る
グーグル奨学生
Tasaki T.
Sohr R.
Xia Z.
ヘルウェグ R.
Hörtnagl H.
ヴァルシャフスキー A.
クォン Y.T.
嗅覚およびその他の感覚系に機能するユビキチンリガーゼUBR3の生化学的および遺伝学的研究。
J. Biol. Chem. 2007; 282: 18510-18520
論文で見る
スコープス (0)
パブコメ
要旨
全文
全文PDF
グーグル奨学生
クロール J.C.
盲蛇Leptotyphlops dulcis(爬虫綱Leptotyphlopidae)の口腔上皮における味蕾。
南西部。Nat. 1973; 17: 365-370
論文で見る
グーグル・スカラー
エメリング C.A.
爪ケラチン、味覚受容体、光関連遺伝子のゲノム回帰からヘビの生態と進化の起源を探る。
Mol. Phylogenet. Evol. 2017; 115: 40-49
論文で見る
グーグル・スカラー
Kley N.J.
Texas blindsnake, Leptotyphlops dulcis (Scolecophidia: Leptotyphlopidae)の下顎と懸棘の形態学。
J. Morphol. 2006; 267: 494-515
論文で見る
Google Scholar
ゲールバック F.R.
ワトキンスJ.F.
クロール J.C.
チフルスネーク、レプトチフルスネーク、およびコロブリスネークのフェロモン痕跡追跡研究。
Behaviour. 1971; 40: 282-294
論文で見る
グーグル・スカラー
グエン・バ・シャルベット K.T.
プランプ A.S.
テシエ・ラヴィーニュ M.
Chédotal A.
Slit1およびSlit2タンパク質は、外側嗅覚路の発達を制御している。
J. Neurosci. 2002; 22: 5473-5480
論文で見る
PubMed
グーグル奨学生
ブーツ R.G.
ブロマートE.F.
スワートE.
ガウハラリ・ヴァン・デル・フルクトK.
Bijl N.
モー C.
プレイス A.
アーツ J.M.
キトトリオシダーゼとは異なる新規酸性哺乳類キチナーゼの同定。
J. Biol. Chem. 2001; 276: 6770-6778
論文で見る
スコープス (423)
PubMed
要旨
全文
全文PDF
グーグル奨学生
ハートルD.
He C.H.
コラーB.
ダ・シルバ C.A.
ホーマー R.
リーC.G.
Elias J.A.
酸性哺乳類キチナーゼはADAM17/上皮成長因子受容体依存性経路を介して分泌され、肺上皮細胞によるケモカイン産生を刺激する。
J. Biol. Chem. 2008; 283: 33472-33482
論文で見る
スコープス (0)
パブコメ
要旨
全文
全文PDF
グーグル奨学生
ジャニアック M.C.
チェイニー M.E.
トシ A.J.
霊長類における酸性哺乳類キチナーゼ遺伝子(CHIA)の進化と体重および食虫性との関係。
Mol. Biol. Evol. 2018; 35: 607-622
論文で見る
スコープス (33)
PubMed
クロス
グーグル奨学生
田畑英夫
糸魚川明彦
鯉沼達也
田山秀樹
樫村明博
坂口雅彦
マトスカ V.
バウアー P.O.
大山文雄
非昆虫食は肉食動物における酸性キチナーゼの構造的・機能的変化をもたらす。
Mol. Biol. 2022; 39: msab331
論文で見る
Google Scholar
グラチェワ E.O.
インゴリア N.T.
ケリー Y.M.
コルデロ・モラレス J.F.
ホロペーター G.
チェスラー A.T.
サンチェス E.E.
ペレス J.C.
ワイズマン J.S.
ジュリアスD.
ヘビによる赤外線検出の分子基盤。
Nature. 2010; 464: 1006-1011
論文で見る
スコープス(310)
PubMed
クロス
グーグル奨学生
フォルツI.N.
ゲルR.E.
ヴィーラーJ.S.
ルッカク M.
サーモン R.A.
シュレーダー J.W.
ヒトのマイトジェン活性化プロテインキナーゼ7(MKK7)は、環境ストレスや生理的刺激によって活性化されるc-Jun N末端キナーゼ/ストレス活性化プロテインキナーゼ(JNK/SAPK)として高度に保存されている。
J. Biol. Chem. 1998; 273: 9344-9351
論文で見る
スコープス (0)
パブコメ
要旨
全文
全文PDF
グーグル奨学生
Sun Y.
ダイクス I.M.
リャン X.
エング S.R.
エバンス S.M.
ターナー E.E.
感覚神経発生におけるIslet1の中心的役割は、感覚と脊髄の遺伝子制御プログラムを結びつける。
Nat. Neurosci. 2008; 11: 1283-1293
論文で見る
スコープス (148)
パブコメ
クロス
グーグル奨学生
岡本吉郎
ペリバンD.
Wiszniewski W.
ベック C.R.
スナイプス G.J.
ルプスキーJ.R.
カジャヴィ M.
クルクミンはトレムブラーJマウスにおいて一過性の細胞ストレス応答を促進する。
Hum. Mol. Genet. 2013; 22: 4698-4705
論文で見る
スコープス (35)
PubMed
クロス
グーグル奨学生
クンバサールA.
プラチェスC.
Gronostajski R.M.
リチャーズ L.J.
リトワックE.D.
転写因子Nfibの欠失は、脳底橋および他の苔状線維核の形成を遅らせる。
J. Comp. Neurol. 2009; 513: 98-112
論文で見る
グーグル奨学生
梶谷 亮
栃本和彦
野口英昭
豊田章男
小倉康夫
奥野雅之
矢花美紀
原田雅史
永易 E.
丸山裕之
他。
全ゲノムショットガンショートリードからの高ヘテロ接合体ゲノムの効率的de novoアセンブリー。
ゲノム研究 2014; 24: 1384-1395
論文で見る
(701件)
PubMed
クロスリファレンス
グーグル奨学生
マクグロスリンJ.W.
チャッカロブチャックJ.P.
ジェーンズ D.E.
エドワーズ S.V.
フェルドマン C.R.
ブロディ E.D.
フレンダー M.E.
ブロディー E.D.
ガーターヘビThamnophis sirtalisの3つの電位依存性ナトリウムチャネル遺伝子におけるテトロドトキシン耐性の並行進化。
Mol. Biol. Evol. 2014; 31: 2836-2846
論文で見る
論文リスト (46)
PubMed
クロス
グーグル奨学生
タン C.-Y.
チャン X.
Xu X.
Sun S.
Peng C.
Song M.-H.
Yan C.
Sun H.
Liu M.
Xie L.
他。
遺伝的マッピングと色彩変異の分子機構.
ゲノム生物学 2023; 24: 46
論文で見る
グーグル奨学生
Aird S.D.
アローラ J.
バルア A.
Qiu L.
Terada K.
ミケーエフ A.S.
マムシの集団ゲノム解析から、毒の化学的性質の根底にある微小進化の力が明らかになった。
Genome Biol. Evol. 2017; 9: 2640-2649
論文で見る
スコープス (45)
PubMed
クロス
グーグル奨学生
チャン Z.-Y.
Lv Y.
Wu W.
Yan C.
Tang C.-Y.
Peng C.
Li J.-T.
致死性エラピッドにおける無視された3本指トキシンサブファミリーの構造と機能の分岐。
Cell Rep.
論文で見る
Google Scholar
Wang Z.
Peng C.
Wu W.
ヤン C.
Lv Y.
Li J.-T.
保存されたノンコーディングエレメントの進化による発達制御が、スクアメイトの四肢欠損に関する洞察を与えている。
Sci. China Life Sci. 2023;https://doi.org/10.1007/s11427-023-2362-5
論文で見る
クロスリファレンス
グーグル奨学生
アンドラーデ P.
ピニョ C.
ペレス・イ・デ・ラヌーザG.
アフォンソ S.
ブレジャ J.
ルビン C.-J.
ワラマンO.
ペレイラ P.
サバティーノ S.J.
ベラティ A.
他。
プテリンおよびカロテノイド遺伝子の制御変化が、ワカゲにおけるバランスのとれた色彩多型の背景にある。
Proc. Natl. Sci. USA. 2019; 116: 5633-5642
論文で見る
スコープス (107)
PubMed
クロス
グーグル奨学生
アルフォルディJ.
ディ・パルマF.D.
グラバー M.
ウィリアムズC.
コング L.
マウセリ E.
ラッセル P.
ロウ C.B.
グロール R.E.
ジャッフェJ.D.
他。
グリーンアノールトカゲのゲノムと鳥類・哺乳類との比較解析。
Nature. 2011; 477: 587-591
論文で見る
筑波大学
PubMed
クロス
グーグル奨学生
ヤン C.
Zhang Z.-Y.
Lv Y.
Wang Z.
Jiang K.
Li J.-T.
哺乳類の高地適応に関する新たな知見。
Int. J. Mol. 科学、2022; 23: 10081
論文で見る
Google Scholar
メロウ C.B.
バウアー R.
コリン J.
ドルガウ B.
ゼルティ D.
ドラン D.W.P.
ジョーンズ C.M.
イズオグ O.G.
ユー M.
ハラムD.
et al.
発達中のヒト網膜の統合転写解析。
Development. 2019; 146: dev169474
論文で見る
スコープス (10)
PubMed
クロス
グーグル奨学生
陳 S.
Zhou Y.
Chen Y.
Gu J.
fastp: 超高速オールインワンFASTQプリプロセッサ。
Bioinformatics. 2018; 34: i884-i890
論文で見る
スコープ(5763)
PubMed
クロスリファレンス
グーグル奨学生
マルセG.
キングスフォード C.
k-merの出現を効率的に並列カウントするための高速でロックフリーのアプローチ。
Bioinformatics. 2011; 27: 764-770
論文で見る
(1922年)
PubMed
クロスリファレンス
グーグル奨学生
Liu B.
Shi Y.
Yuan J.
Hu X.
Zhang H.
Li N.
Li Z.
チェン Y.
Mu D.
Fan W.
ゲノムの特徴量をk-mer頻度から推定するde novoゲノムプロジェクト。
2013https://doi.org/10.48550/arXiv.1308.2012
論文で見る
クロスフィルム
Google Scholar
Vurture G.W.
セドラゼックF.J.
ナッテスタッド M.
アンダーウッド C.J.
ファング H.
グルトウスキー J.
Schatz M.C.
GenomeScope:ショートリードからリファレンスフリーの高速ゲノムプロファイリング。
Bioinformatics. 2017; 33: 2202-2204
論文で見る
スコープ (677)
PubMed
クロスリファレンス
グーグル奨学生
イェ C.
ヒル C.M.
ウー S.
Ruan J.
Ma Z.S.
DBG2OLC:第3世代シーケンシング技術のロングエラーリードを用いた大規模ゲノムの効率的アセンブリー。
Sci. Rep. 2016; 6: 31900https://doi.org/10.1038/srep31900
論文で見る
スコープス (164)
PubMed
クロス
グーグル奨学生
ラングミードB.
サルツバーグ S.L.
Bowtie 2による高速ギャップドリードアライメント。
Nat. Meth. 2012; 9: 357-359
論文で見る
筑波大学
パブコメ
クロス
グーグル奨学生
Li H.
ダービン R.
Burrows-Wheeler変換を用いた高速で正確なロングリードのアライメント。
Bioinformatics. 2010; 26: 589-595
論文で見る
論文リスト
PubMed
クロスリファレンス
グーグル奨学生
胡 J.
王Z.
孫 Z.
Hu B.
Ayoola A.O.
Liang F.
Li J.
サンドバル J.R.
クーパー D.N.
イェ K.
他。
ノイズの多いロングリードの効率的なエラー訂正と正確なアセンブルツール。
2023https://doi.org/10.1101/2023.03.09.531669
論文で見る
クロスフィルム
グーグル奨学生
Hu J.
ファン J.
Sun Z.
Liu S.
NextPolish:ロングリードアセンブリのための高速で効率的なゲノムポリッシングツール。
Bioinformatics. 2020; 36: 2253-2255
論文で見る
ゲノムアセンブリー
パブコメ
クロスフィルム
グーグル奨学生
Ruan J.
Li H.
wtdbg2による高速かつ正確なロングリードアセンブリ。
Nat. Meth. 2020; 17: 155-158
論文で見る
論文 (0)
パブコメ
クロス
グーグル奨学生
セルヴァン N.
ヴァロコーN.
ラジョアB.R.
ヴィアラ E.
チェン C.-J.
ヴェール J.-P.
ハード E.
デッカー J.
バリロ E.
HiC-Pro:Hi-Cデータ処理のための最適化された柔軟なパイプライン。
ゲノムバイオロジー 2015; 16: 259
論文で見る
スコープス (989)
PubMed
Crossref
グーグル奨学生
デュラン N.C.
シャミム M.S.
マチョルI.
ラオ S.S.P.
ハントリー M.H.
ランダー E.S.
エイデン E.L.
Juicerはループ解像度のHi-C実験を解析するためのワンクリックシステムを提供します。
Cell. Syst. 2016; 3: 95-98
論文で見る
PubMed
要旨
全文
全文PDF
グーグル奨学生
ダドチェンコ O.
バトラ S.S.
オメル A.D.
ナイキスト S.K.
ホーガー M.
デュラン N.C.
シャミム M.S.
マチョル I.
ランダー E.S.
エイデンA.P.
ほか
Hi-Cを用いたイエネズミゲノムのde novoアセンブリーにより染色体長のスキャフォールドが得られた。
Science. 2017; 356: 92-95
論文で見る
スコープス (796)
PubMed
クロス
グーグル奨学生
バートン J.N.
アデイ A.
パットワルダンR.P.
Qiu R.
キッツマンJ.O.
Shendure J.
クロマチン相互作用に基づくde novoゲノムアセンブリーの染色体スケールのスキャフォールディング。
Nat. Biotechnol. 2013; 31: 1119-1125
論文で見る
筑波大学
パブコメ
クロス
グーグル奨学生
シマン F.A.
ウォーターハウス R.M.
イオアニディスP.
クリベンツェワ E.V.
ズドブノフ E.M.
BUSCO: シングルコピーオルソログによるゲノムアセンブリとアノテーションの完全性の評価。
Bioinformatics. 2015; 31: 3210-3212
論文で見る
スコープス (5799)
PubMed
クロス
グーグル奨学生
フリンJ.M.
ハブリー・R.
グベールC.
ローゼン J.
クラーク A.G.
フェショットC.
Smit A.F.
トランスポーザブル・エレメント・ファミリーの自動ゲノム発見のためのRepeatModeler2。
Proc. Natl. Acad. Sci. USA. 2020; 117: 9451-9457
論文で見る
スコープス (558)
パブコメ
クロス
グーグル奨学生
タライロ・グラオバック M.
チェン N.
第4章. RepeatMaskerを用いたゲノム配列中の繰り返し要素の同定。
Curr. Protoc。バイオインフォマティクス。2009; 第4章: 4.10.1-4.10.14
論文で見る
パブコメ
グーグル奨学生
シュタンケ M.
ディーカンスM.
バエルチR.
Haussler D.
ネイティブおよびシンテナリーマッピングされたcDNAアラインメントを用いたde novo遺伝子探索の改善。
Bioinformatics. 2008; 24: 637-644
論文で見る
遺伝子発現
PubMed
クロスリファレンス
グーグル奨学生
ケイルワーゲンJ.
ウェンク M.
エリクソンJ.L.
シャタット M.H.
グラウ J.
Hartung F.
相同性に基づく遺伝子予測のためのイントロン位置保存の利用。
Nucleic Acids Res.
論文で見る
スコープス (269)
PubMed
Crossref
グーグル奨学生
コバカ S.
ジミンA.V.
ペルテアG.M.
ラザギR.
サルツバーグ S.L.
Pertea M.
StringTie2を用いたロングリードRNA-seqアラインメントからのトランスクリプトームアセンブリ。
Genome Biol.
論文で見る
スコープス (447)
PubMed
クロスフィルム
グーグル奨学生
ドビンA.
デイビスC.A.
シュレシンジャーF.
ドレンコウ J.
ザレスキー C.
ジャ S.
バトゥット P.
シャイソン M.
Gingeras T.R.
STAR:超高速ユニバーサルRNA-seqアライナー。
Bioinformatics. 2013; 29: 15-21
論文で見る
筑波大学
PubMed
クロスリファレンス
グーグル奨学生
ハースB.J.
サルツバーグ S.L.
Zhu W.
ペルテア M.
アレンJ.E.
オービス J.
ホワイト O.
ビューエル C.R.
Wortman J.R.
EVidenceModelerとProgram to Assemble Spliced Alignmentsを用いた真核生物遺伝子の自動構造アノテーション。
ゲノム生物学 2008; 9: R7
論文で見る
遺伝子アノテーション
PubMed
クロスリファレンス
グーグル奨学生
タン S.
ロムサゼA.
ボロドフスキー M.
RNA転写産物におけるタンパク質コード領域の同定。
Nucleic Acids Res. 2015; 43: e78
論文で見る
スコープス (181)
PubMed
Crossref
グーグル奨学生
浦崎直樹
高木秀明
夏目 聡
植村 彰。
谷合直樹
宮城直樹
福島雅之
鈴木 S.
太郎良 K.
玉木美智子
他
熱帯・亜熱帯地域の野菜・薬用植物であるゴーヤ(Momordica charantia)のゲノム配列(ドラフト)。
DNA Res. 2017; 24: 51-58
論文で見る
PubMed
グーグル奨学生
Zhang Z.
シュワルツ S.
ワグナーL.
ミラーW.
DNA配列の整列のための貪欲なアルゴリズム。
J. Comput. 生物学 2000; 7: 203-214
論文で見る
筑波大学
PubMed
クロス
グーグル奨学生
キム J.
ファーレ M.
オーヴィル L.
カピタヌ B.
ラーキン D.M.
マー J.
ルーイン H.A.
真獣類の染色体の復元と進化の歴史。
Proc. Natl. Sci. USA. 2017; 114: e5379-e5388
論文で見る
スコープス (0)
PubMed
クロス
グーグル奨学生
Krzywinski M.
シャインJ.
ビロルI.
コナーズ J.
ガスコイン R.
ホースマン D.
ジョーンズ S.J.
マーラ M.A.
Circos:比較ゲノム学のための情報美学。
ゲノム研究 2009; 19: 1639-1645
論文で見る
筑波大学
PubMed
クロス
グーグル奨学生
エムズD.M.
ケリー S.
OrthoFinder:全ゲノム比較における基本的なバイアスを解決することで、オルソグループ推論の精度が劇的に向上。
ゲノム生物学 2015; 16: 157
論文で見る
スコープス (1748)
PubMed
Crossref
グーグル奨学生
クインラン A.R.
ホール I.M.
BEDTools:ゲノムの特徴を比較するための柔軟なユーティリティ群。
Bioinformatics. 2010; 26: 841-842
論文で見る
筑波大学
PubMed
クロスリファレンス
グーグル奨学生
Chen T.
リウ Y.-X.
Huang L.
ImageGP:科学研究者のための使いやすいデータ可視化ウェブサーバー。
iMeta. 2022; 1: e5
記事で見る
グーグル・スカラー
ウィッカム H.
ggplot2: データ分析のためのエレガントなグラフィックス。
シュプリンガー・フェアラーク, 2016
論文で見る
クロスリファレンス
グーグル・スカラー
カペラ=グティエレス S.
シジャ=マルティネスJ.M.
Gabaldón T.
大規模系統解析における自動アライメントトリミングツール。
Bioinformatics. 2009; 25: 1972-1973
論文で見る
筑波大学
PubMed
クロスリファレンス
グーグル奨学生
グエン L.-T.
シュミットH.A.
von Haeseler A.
ミン B.Q.
IQ-TREE:最尤系統推定のための高速かつ効果的な確率的アルゴリズム。
Mol. Biol. Evol. 2015; 32: 268-274
論文で見る
スコープス (10950)
PubMed
クロス
グーグル奨学生
ロイティノヤA.
ゴールドマン N.
系統を意識したギャップ配置が配列アライメントと進化解析におけるエラーを防ぐ。
Science. 2008; 320: 1632-1635
論文で見る
論文
PubMed
クロス
グーグル奨学生
Yang Z.
PAML 4: 最尤法による系統解析。
Mol. Biol. Evol. 2007; 24: 1586-1591
論文で見る
筑波大学
PubMed
クロス
グーグル奨学生
サヤリ E.
ホイットフィールドJ.B.
ミララブ S.
DiscoVista:遺伝子ツリーの不一致を解釈可能に可視化したもの。
Mol. Phylogenet. Evol. 2018; 122: 110-115
論文で見る
スコープス (56)
PubMed
クロス
グーグル奨学生
ベックステットM.
ホーマンR.
ジーゲリッヒR.
Kurtz S.
位置特異的スコアリング行列のマッチングのための高速インデックスベースアルゴリズムとソフトウェア。
BMC Bioinformatics. 2006; 7: 389
論文で見る
哺乳類
パブコメ
クロスリファレンス
グーグル奨学生
キム D.
ラングミード B.
Salzberg S.L.
HISAT: 低メモリ容量の高速スプライスアライナー。
Nat. Meth. 2015; 12: 357-360
論文で見る
スコープス (10337)
PubMed
クロス
グーグル奨学生
ラングフェルダー P.
ホルバス S.
WGCNA:重み付け相関ネットワーク解析のためのRパッケージ。
BMC Bioinformatics. 2008; 9: 559
論文で見る
筑波大学
PubMed
クロスリファレンス
グーグル奨学生
スーペックF.
ボシュニャクM.
シュクンカ N.
Šmuc T.
REVIGOは遺伝子オントロジー用語の長いリストを要約し、可視化する。
PLoS One. 2011; 6e21800
論文で見る
論文リスト
PubMed
クロス
グーグル奨学生
ヒラーM.
シャールB.T.
インディアンV.B.
キングスレー D.M.
ヘイギー・L.R.
Bejerano G.
近縁種間の独立した表現型ロスを利用して、遺伝子型と表現型を結びつける「フォワードゲノミクス」アプローチ。
Cell. 2012; 2: 817-823
論文で見る
筑波大学
パブコメ
要旨
全文
全文PDF
グーグル奨学生
Li B.
デューイ C.N.
RSEM:参照ゲノムの有無にかかわらず、RNA-Seqデータからの正確な転写産物の定量化。
BMC Bioinformatics. 2011; 12: 323
論文で見る
論文リスト
PubMed
クロスリファレンス
グーグル奨学生
Choi Y.
シムズ G.E.
マーフィー S.
ミラー J.R.
チャン A.P.
アミノ酸置換とインデルの機能的効果を予測する。
PLoS One. 2012; 7e46688
論文で見る
スコープス(2019)
クロスリファレンス
グーグル奨学生
リー・B.
フィルモア N.
バイ Y.
コリンズ M.
トムソン J.A.
スチュワート R.
Dewey C.N.
RNA-Seqデータからのde novoトランスクリプトームアセンブリーの評価。
ゲノム生物学 2014; 15: 553
論文で見る
スコープス (182)
PubMed
クロスフィルム
グーグル奨学生
ラブM.I.
フーバーW.
Anders S.
DESeq2によるRNA-seqデータのフォルドチェンジと分散のモデレート推定。
ゲノム生物学 2014; 15: 550
論文で見る
論文
PubMed
クロスレビュー
グーグル奨学生
ロビンソン M.D.
マッカーシー D.J.
Smyth G.K.
edgeR:デジタル遺伝子発現データの差分発現解析のためのBioconductorパッケージ。
Bioinformatics. 2010; 26: 139-140
論文で見る
(2010年)
PubMed
クロスリファレンス
グーグル奨学生
グラバー M.G.
ハースB.J.
ヤスールM.
レヴィン J.Z.
トンプソン D.A.
アミット I.
アディコニス X.
ファン L.
レイチョードリー R.
Zeng Q.
他。
参照ゲノムなしのRNA-Seqデータからの全長トランスクリプトームアセンブリ。
Nat. Biotechnol. 2011; 29: 644-652
論文で見る
筑波大学
PubMed
クロス
グーグル奨学生
Li W.
Godzik A.
Cd-hit: タンパク質または塩基配列の大規模セットをクラスタリングおよび比較するための高速プログラム。
Bioinformatics. 2006; 22: 1658-1659
論文で見る
筑波大学
PubMed
クロスリファレンス
グーグル奨学生
コサコフスキー ポンド S.L.
プーンA.F.Y.
ベラスケス R.V.
ウィーバー S.
ヘプラー N.L.
マレル B.
シャンク S.D.
マガリス B.R.
ブービエ D.
ネクルテンコA.
他
HyPhy 2.5-系統樹を用いた進化仮説検定のためのカスタマイズ可能なプラットフォーム。
Mol. Biol. Evol. 2020; 37: 295-299
論文で見る
スコープス (98)
PubMed
クロス
グーグル奨学生
バーブリンクF.T.
グラジオティンF.G.
パイロンP.A.
カンドールD.
ドネラン S.
フランシス I.
キョウ J.S.
クラウス F.
マーフィー R.W.
ヌーナンN.
他
Squamata(トカゲ、ヘビ、両生類)のゲノムスケールデータを調査した結果、従来の主要な形態学的関係は支持されなかった。
Syst. Biol. 2019; 69: 502-520
論文で見る
グーグル・スカラー
チェン・エイチ.
ランガサミー M.
タン S.Y.
ワン・エイチ
ジークフリート B.D.
Western corn rootworm beetlesからの全DNA抽出のための5つの方法の評価。
PLoS One. 2010; 5e11963
論文で見る
Google Scholar
Liu Z.
Zhang L.
Yan Z.
Ren Z.
Han F.
Tan X.
Xiang Z.
Dong F.
Yang Z.
Liu G.
et al.
石灰岩ラングールのカルスト生息地における生理的・形態的適応のゲノムメカニズム。
Mol. Biol. Evol. 2020; 37: 952-968
論文で見る
Google Scholar
ズーノミア・コンソーシアム
科学的発見と保全のための比較ゲノムマルチツール。
Nature. 2020; 587: 240-245
記事で見る
スコープス (95)
PubMed
クロス
グーグル奨学生
マクリーンC.Y.
ブリストルD.
ヒラーM.
クラーク S.L.
シャー B.T.
ロウ C.B.
ベンゲル A.M.
Bejerano G.
GREATはシス制御領域の機能的解釈を向上させる。
Nat. Biotechnol. 2010; 28: 495-501
論文で見る
遺伝子発現
PubMed
クロス
グーグル奨学生
ホアンD.T.
チェルノモールO.
フォン・ヘーセラーA.
ミン B.Q.
Vinh L.S.
UFBoot2:超高速ブートストラップ近似の改良。
Mol. Biol. Evol. 2018; 35: 518-522
論文で見る
スコープス (3523)
PubMed
クロス
グーグル奨学生
カルヤナモワース S.
ミン B.Q.
ウォン T.K.F.
フォン・ヘーセラーA.
Jermiin L.S.
ModelFinder:正確な系統推定のための高速モデル選択。
Nat. Meth. 2017; 14: 587-589
論文で見る
スコープス (5914)
PubMed
クロス
グーグル奨学生
ジャンパーJ.
エヴァンス R.
プリッツェルA.
グリーン・T.
フィガーノフ M.
ロンネバーガー O.
トゥニャスヴナクールK.
ベイツ R.
ジーデク A.
ポタペンコA.
他
AlphaFoldによる高精度タンパク質構造予測。
Nature. 2021; 596: 583-589
論文で見る
筑波大学
PubMed
クロス
グーグル奨学生
Zheng R.
ワン C.
Mei S.
Qin Q.
Wu Q.
Sun H.
Chen C.-H.
Brown M.
Zhang X.
Meyer C.A.
et al.
Cistrome Data Browser:遺伝子制御解析のための拡張データセットと新しいツール。
Nucleic Acids Res. 2019; 47: D729-D735
論文で見る
スコープス (322)
PubMed
クロスリファレンス
グーグル奨学生
カスティーリョ-デイビスC.I.
コンドラショフF.A.
ハートルD.L.
Kulathinal R.J.
2つの動物門におけるタンパク質分岐の機能ゲノム分布:共進化、ゲノム競合、制約。
ゲノム研究 2004; 14: 802-811
論文で見る
ゲノム配列
PubMed
クロス
グーグル奨学生
コサコフスキー池S.L.
フロストS.D.W.
ミューズ S.V.
HyPhy: 系統樹を用いた仮説検定.
. 2005; 21: 676-679
論文で見る
Google Scholar
アイゼンバーグ E.
レヴァノン E.Y.
ヒトのハウスキーピング遺伝子、再訪。
Trends Genet. 2013; 29: 569-574
論文で見る
日本学術振興会特別研究員
PubMed
要旨
全文
全文PDF
グーグル奨学生
Ebert J.
ヘビの赤外線感覚:行動学的および解剖学的検討(Crotalus atrox, Python regius, Corallus hortulanus)。
(博士論文)Rheinische Friedrich Wilhelms University.
論文で見る
グーグル
論文情報
出版履歴
出版 2023年6月19日
受理 受理:2023年5月19日
改訂版受理 2023年4月6日
受理:2023年4月6日 2022年8月23日
出版段階
インプレス、ジャーナル予稿集
識別
DOI: https://doi.org/10.1016/j.cell.2023.05.030
著作権
© 2023 The Author(s). 発行:エルゼビア社
ユーザーライセンス
クリエイティブ・コモンズ 表示 - 非商用 - 改変禁止 (CC BY-NC-ND 4.0) | ja.wikipedia.orgで無料でダウンロードできます。
再利用方法
サイエンスダイレクト
ScienceDirectでこの記事にアクセスする
図表
図
図S1図1に関連するヘビの系統樹と分岐時間樹
図1ヘビの系統樹
図2ヘビゲノムの進化的特徴
図S2図2に関連するヘビゲノムの特徴
図S3図3およびSTAR Methodsに関連する、ヘビ特異的ゲノム構造変異(SSSV)、ヘビで分岐したがアウトグループで保存されている非コードエレメント(SD-SNE)、および進化解析に用いたオルソログ遺伝子
図S4ヘビの四肢欠損、胴体伸長、肺の非対称性、眼構造の単純化の遺伝的基盤(図3、4に関連
図3ヘビにおける骨格系の進化と器官適応の遺伝的基盤
図4ヘビ特有の感覚器進化に関連するゲノムの特徴(PSG、REG、新しく進化した遺伝子、SD-CNE関連遺伝子、失われた遺伝子、SSSV関連遺伝子を強調表示
図S5図4と図5に関連するヘビ感覚器系のゲノム進化
図5盲目および赤外線感受性のヘビの進化
表
表1ヘビ14種のゲノムアセンブリ統計量
関連論文
このサイトのコンテンツは、あらゆる科学分野の医療従事者および研究者を対象としています。
研究ジャーナル
細胞
癌細胞
細胞化学生物学
細胞ゲノム
細胞宿主と微生物
細胞代謝
細胞レポート
セルレポーツ医学
セルレポートメソッド
セルレポート 物理科学
セルレポート サステイナビリティ
細胞幹細胞
細胞システム
化学
化学触媒
カレントバイオロジー
発生細胞
デバイス
ヘリオン
免疫
アイサイエンス
ジュール
マター
医学
分子細胞
ニューロン
一つの地球
パターン
STARプロトコル
構造
トレンドレビュージャーナル
生化学
バイオテクノロジー
癌
細胞生物学
化学
認知科学
生態学・進化学
内分泌学・代謝学
遺伝学
免疫学
微生物学
分子医学
神経科学
寄生虫学
薬理学
植物科学
パートナージャーナル
AJHG
生物物理ジャーナル
生物物理学レポート
HGGアドバンス
分子植物
分子治療ファミリー
ネクサス
植物通信
幹細胞レポート
イノベーション
コレクション
ベスト・オブ・セルプレス
セルプレスレビュー
セルプレスセレクション
コンソーシアムハブ
Nucleusコレクション
スナップショット・アーカイブ
ジャーナルを超えて
セルキャリアネットワーク
セルメンター
細胞シンポジウム
ラボリンク
ウェビナー
記事を進化させる
コミュニティ・レビュー
図360
スニークピーク
STARメソッド
社会における科学
セル・ピクチャー・ショー
セルプレスポッドキャスト
セルプレスビデオ
ぬり絵とコミック
リサーチ・アーク
コネクト
セルプレスについて
採用情報
お問い合わせ
ヘルプ&サポート
ニュースルーム
出版アラート
アクセス
購読申し込み
今すぐ読む
司書に薦める
インフォメーション
広告主の皆様へ
リクルーターの方へ
図書館員の方へ
利用規約
プライバシーポリシー
アクセシビリティ
当サイトでは、サービスの提供・向上およびコンテンツのカスタマイズのためにクッキーを使用しています。クッキーの設定を更新するには、このサイトのクッキー設定をご覧ください。
著作権 © 2023 Elsevier Inc.第三者が提供する一部のコンテンツを除く。
この記事が気に入ったらサポートをしてみませんか?
