
【W8】タンパク質データの取得_11_Step3_最終工程
【W8の目的とStep3概要】
(i) PDBデータベースからEGFRの全てのPDB IDを取得し、
(ii) X線結晶構造解析による構造で、最も質の良い4つのタンパク質ーリガンド複合体構造を取得して保存します。
Python版はより発展的です。
上記の(ii)を実行中。デモデータを使ってW8のStep2を学びました。
出力は下記のようなデータテーブルです。

今回から最終工程のStep3。
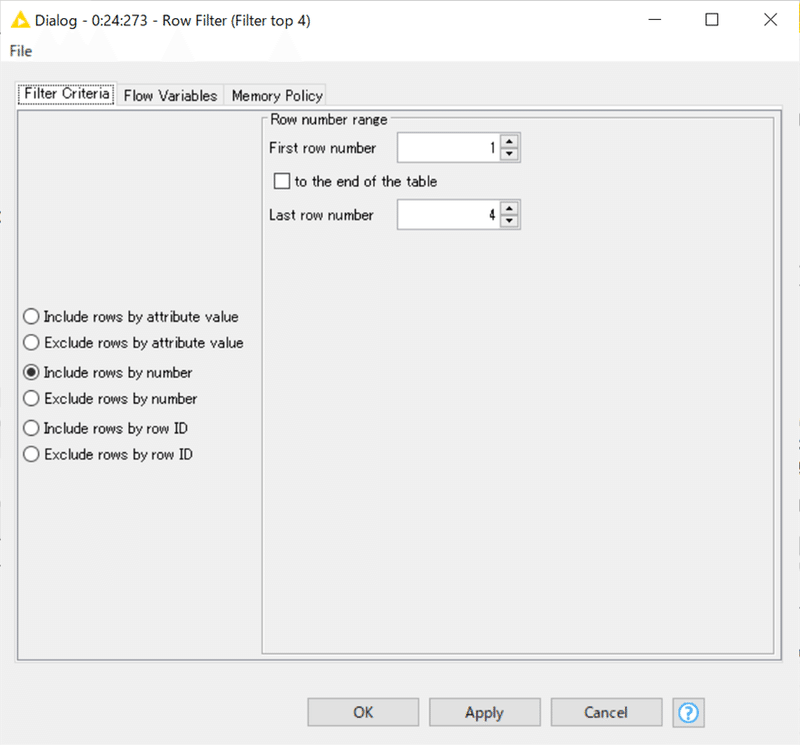
「最も質の良い4つのタンパク質ーリガンド複合体構造」すなわち解像度順でTop 4に絞って共結晶のデータを取得します。

上位4つに絞るのですが、同スコアで4位が3つあるうち、3POZだけが残りますね。
PDBからデータが入手できた順(Row ID)に並ぶので、PDB_IDの小さいデータすなわち大まかにいえばより古いPDBデータが上に来るのではと推測します。
【PDBデータ取得】
【Row Filter】
設定:
結果:

先述の通り4レコードに絞りました。
【PDB Downloader】

日本語化されたディスクリプションを一部引用して少し加筆し説明とします。
このマニアックなノードまで作成し解説もつけてくれるVernalis社と日本語訳して下さったInfocom社の尽力に再び感謝しつつ。
このノードおよび対応するソースノードでは,RCSBのPDBから構造および配列ファイルをダウンロードすることができます.サポートしているフォーマットは以下の通りです(全ての構造で全てのフォーマットが利用できるわけではありません!)。
・ PDB
・ mmCIF
・ Structure Factors
・ PDBML
・ FASTA Format Sequence
・ NMR Restraints
・ NMR Restraints v2
・ NMR Chemical Shifts
PDBファイルはPDBセルとして、PDBMLはXMLセルとしてキャストされます。
構造IDのチェックは行っていません。
構造IDが無効な場合、欠損値を含むセルが返されます。
警告 - PDBMLオプションは非常に大きなテーブルになる可能性があります。
今回はPDB形式でデータを入手しますので下記の設定となります。
設定:

結果:

PDBの1セルに1つのPDBデータが入っているのでかなりの情報量です。
その中身の説明をしだしたらそれだけで1章分になりますので割愛しますが、下記の記事などとても参考になるでしょう。Magattacaさんは本当に素晴らしい!
【String Manipulation】

pdb_filenameというカラムを新たに作り、今回の各PDBファイルの保存名を定めています。
設定:
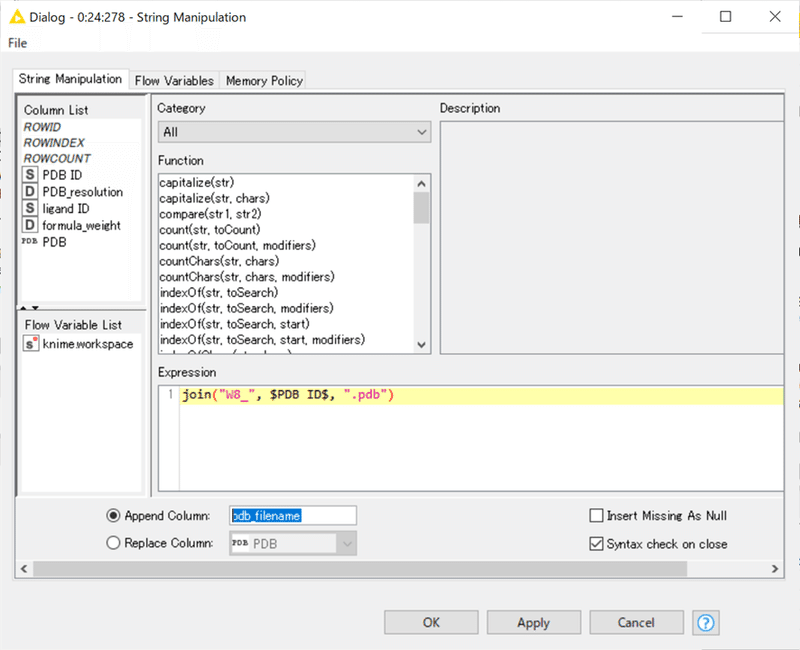
結果:

このデータテーブルはW8のメタノードの出力にもなっています。

すなわち上記の出力ポートも全く同じ内容です。
【PDB Saver】

日本語化されたディスクリプションを一部引用して少し加筆し説明とします。
このノードでは、テーブルの列に格納されているPDBファイルのコピーを、2番目の列に格納されているファイルパスに保存します。
ファイルパスは完全なパスでなければならず、例えばC:\Temp\myfile.pdbのようになります。
完全なフォルダパスが存在しない場合は作成を試みます。
返されるカラムは、入力カラムのいずれかが欠損している場合は欠損値を含み、ファイルが正常に書き込まれた場合は「true」、書き込まれなかった場合は「false」となります(上書きがオフに設定されていてファイルが存在していた場合や、ディレクトリが作成できなかったり有効なパスではなかった場合など)。
設定:

“Save all files in parent folder”にチェックが入っていて、デモデータでは相対パスで保存先が指定されています。
結果:

Trueすなわち書き込み成功です。さて、どこに保存されているかというと、皆さんのKNIME APのknime-workspaceフォルダを探してください。

相対パスに関してはまっきーさんのブログを引用しておきますね。
最後にもう一つ
Note for "PDB Saver": Please update the output path by clicking on Browse or specify one of your choice (double click "PDB Saver" node to access its configuration menu).
デモデータでは上記の場所にファイルを書き込みますが、皆さんが利用される際は各自で適切なフォルダを指定した方がいいでしょう。そんなことまで丁寧にworkflow内にコメントしてくれているTOCの設計者の方々。
最後の最後まで至れり尽くせりの配慮にあらためて最大限の謝意を示して、私も筆をおこうと思います。
TeachOpenCADDは本当に素晴らしいケモインフォマティクス教材です!
本当に本当にありがとう!!
記事を読んでいただきありがとうございます。 先人の智慧をお借りしつつ、みなさんに役立つ情報が届けられたらと願っています。 もしサポートいただけるなら、そのお金はKNIMEの無料勉強会の開催資金に充てようと思います。
