McNemar検定
こんにちは、ヤマグチです。今回は検定のお話です。
いま大学院の研究で二群をマッチングして比較する研究をやっています。結果の検定にカイ二乗検定を使用したところ、ボスからMcNemar検定を使うように言われたので、調べてみました。こちらの奥村先生の解説がとてもわかりやすかったです。
こちらではMcNemer検定の説明とRでデータフレームからMcNemar検定を行う流れを解説します。
McNemar検定とは
結果が二値であるデータの解析に使われる検定です。対応のあるt検定のカテゴリ変数版のような感じです。奥村先生の例では、20人にプリテストとポストテストを受けさせて、合格・不合格を調べています。
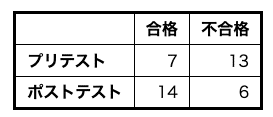
2x2表なので、普通はカイ二乗検定やフィッシャーの正確確率検定を行いますが、前提として各マスの数字が独立している必要があります。今回のような前後関係にあるデータにはこれらの検定は行えません。そこで登場するのがMcNemar検定です。マッチングしたデータにもこれがあてはまり、McNemar検定を使用します。
上記の奥村先生の例ではフィッシャーの検定をおこなうとp = 0.05616で有意差を認めませんが、McNemark検定では p = 0.0455となり有意になります。検出感度も上がるようですね。
McNemer検定は何をやっている?
McNemar検定はペアごとの結果を分類して検討しています。上記の例では、ある人のプレテストとポストテストの結果は
合格 ー 合格
合格 ー 不合格
不合格 ー 合格
不合格 ー 不合格
の4パターンに分けられます。これを2x2表にすると以下のようになります。

ここでどちらも合格、どちらも不合格は前後で差がありません。そこで前後に差のある、合格ー不合格、不合格ー合格の2マスに注目します。
合格から不合格になった人は1名(5%)しかいないのに、不合格から合格になった人は8人もいます。これは違いがあるように感じますね。
McNemar検定ではこの違いが有意かどうかをχ二乗検定を使って調べます。
データフレームからMcNemar検定をおこなう
普段はデータフレームからTidyverseを使ってデータの変形をおこなっているので、2x2表もデータフレームから作成して、そのまま検定をおこなう流れをやってみました。コードはこちらにおいてあります。
デモデータ作成
まず、上記の奥村先生の例と一致するデモデータを作ります。
暴露にAとBがあり、1:1でマッチングしたとします。
# デモデータ作成 -------------------------------------------------------------------
A_success = data_frame(id = 1:7, exposure = "A", outcome = 1, group = 1:7)
A_failure = data_frame(id = 8:20, exposure = 'A', outcome = 0, group = 8:20)
B_success = data_frame(id = 21:26,exposure = 'B', outcome = 1, group = 1:6)
B_failure = data_frame(id = 27, exposure = 'B', outcome = 0, group = 7)
B_success_2 = data_frame(id = 28:35, exposure = 'B', outcome = 1, group = 8:15)
B_failure_2 = data_frame(id = 36:40, exposure = 'B', outcome =0, group = 16:20)
data <- A_success |>
bind_rows(A_failure) |>
bind_rows(B_success) |>
bind_rows(B_failure) |>
bind_rows(B_success_2) |>
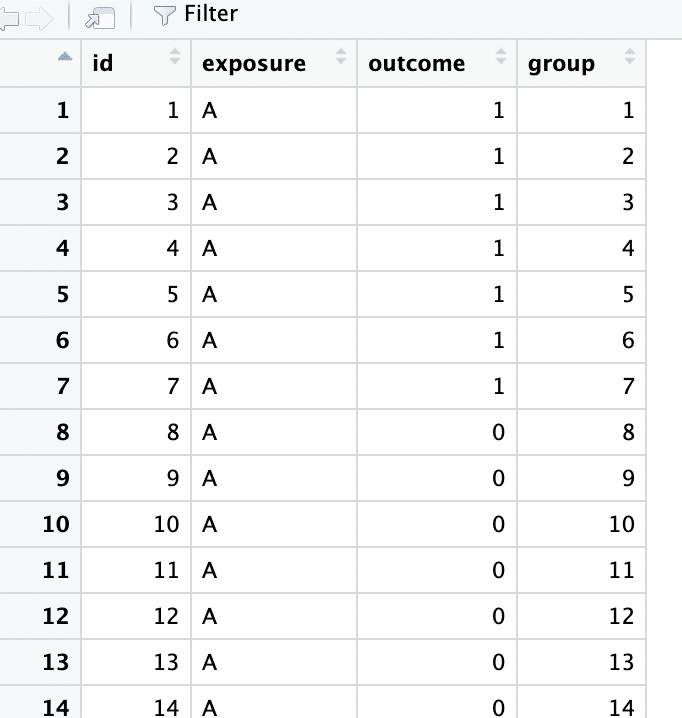
bind_rows(B_failure_2) |> view()これを実行すると下記のようなデータフレームが出来上がります。
outcomeに1と0があります。groupにはマッチングしたグループ毎の連番を振っています。1から20までの20グループですね。
McNemar列を作成
それでは、まず2x2表を作るための列を作成します。ここではMcNemar列と名付けました。以下のコードを実行します。
# データ準備 ------------------------------------------------------------
data_for_McNemar <- data |>
#グループ化
group_by(group) |>
# データフレームに他の列がある場合は、対象の列に絞り込む
select(id,exposure,outcome,group) |>
# exposureで並べ替えてAとBの順番を揃える
arrange(exposure) |>
# outcomeの列を結合してMcNemar用の列を作る
mutate(McNemar = paste0(outcome,collapse = "")) |>
ungroup() |>
# A群だけ取り出す(ペアで考えるため)
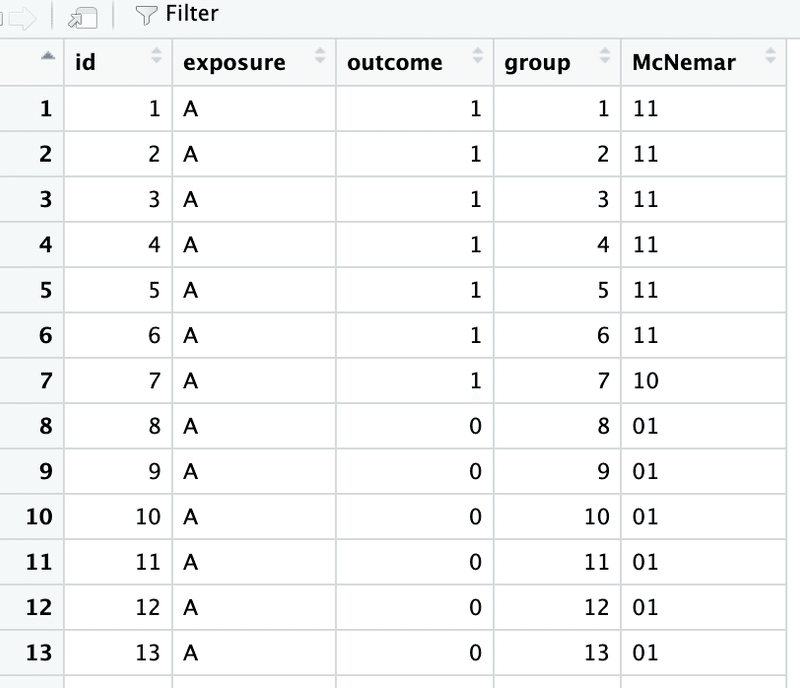
filter(exposure == "A") |> view()データフレームをgroupでグループ化して、そのグループ毎にoutcomeを文字列として結合し、McNemarという列に入れています。
例えばあるグループが合格(1)、合格(1)であれば、11、不合格(0)、合格(1)であれば01という具合です。結果的にMcNemar列には11, 10, 01, 00の4つの文字列のいずれかが入ります。
2x2表を作成
次にMcNemar列のデータ個数をcount()関数で調べます。
# McNemar用の文字列を数える
data_for_McNemar <- data_for_McNemar |>
count(McNemar)
# McNemar n
# 1 00 5
# 2 01 8
# 3 10 1
# 4 11 6このn列をpull()関数で取り出し、matrixに変形します。
# McNemar用の文字列を数える
data_for_McNemar <- data_for_McNemar |>
count(McNemar) |>
# McNemar n
# 1 00 5
# 2 01 8
# 3 10 1
# 4 11 6
# n列をベクトルで取り出す
pull(n) |>
# ベクトルをmatrixに変形
matrix(nrow =2)
# [,1] [,2]
# [1,] 5 1
# [2,] 8 6
2x2表ができあがりました。ここで数字の位置が想定通りでないと検定結果が変わってしまうので要チェックです。
McNemar検定
このmatrixをmcnemar.test()に入れてあげると検定の出来上がりです。
# 検定
mcnemar.test(data_for_McNemar)
# McNemar's chi-squared = 4, df = 1, p-value = 0.0455二項分布を用いる方法
奥村先生によると、上記のmcnemar.testはχ二乗検定を用いていますが、二項分布を使ったほうがより厳密に計算できるそうです。その場合は以下のやりかたになります。
最初にexact2x2 packageの読み込みが必要です。
library(exact2x2)
# 依存するtestthatがmatches,is_nullなどをマスクするので注意
mcnemar.exact(data_for_McNemar)
# Exact McNemar test (with central confidence intervals)
#
# data: data_for_McNemar
# b = 1, c = 8, p-value = 0.03906
# alternative hypothesis: true odds ratio is not equal to 1
# 95 percent confidence interval:
# 0.00281705 0.93235414
# sample estimates:
# odds ratio
# 0.125
こちらのほうが、さらにp値は小さくなっています。
まとめ
マッチングしたデータに対してはχ二乗検定ではなくて、McNemar検定をおこないましょう。
この記事が気に入ったらサポートをしてみませんか?
