
【中編】 新型コロナ人工ウイルス論: 新型コロナ変異株の進化をRNAウイルス進化の視点から俯瞰する
前編に引き続き私自身の論文を参考資料として引用しながら、新型コロナ人工ウイルス論についてお話ししていこうと思います。
以下が論文のリンクです ⬇︎
https://www.mdpi.com/2674-0583/2/3/17
SARS-CoV-2変異株はN変異に偏っている

SARS-CoV-1はSARSの原因ウイルスであり、RaTG13は新型コロナウイルスの近接起源と推定されるコウモリ由来のウイルスです。SARS-CoV-1とRaTG13、プロト-武漢の間のN変異、S変異の頻度を見てみましょう。
図1はそれぞれの遺伝子におけるN変異数 (赤) とS変異数 (青) のグラフです。ここでのN変異はアミノ酸配列を変える変異、S変異はアミノ酸配列を変えない変異です。SARS-CoV-1とRaTG13、SARS-CoV-1とプロト-武漢の間の変異数は約6000、RaTG13とプロト-武漢の間の変異数は約1000です。SARS-CoV-1、RaTG13、プロト-武漢の変異を比較すると、ほとんどの遺伝子でS変異がN変異よりも頻度が高い事が分かります (図1)。
一般論として、遺伝子のサイズに比例して変異の頻度は上がります。コロナウイルスのスパイク遺伝子の大きさ (長さ) は3.8 kbであり、これは比較的大きな遺伝子です。そしてORF1ab (オープンリーディングフレーム1ab) は21.3 kbの巨大タンパクであり、30 kbあるSARS-CoV-2ゲノムの71.2%を占めています。ORF1abはプロテアーゼによる切断後にたくさんの小さなタンパク群となり、そのうちのいくつかのタンパクはゲノム複製と転写を行うレプリカーゼ-転写酵素複合体として知られる大きなタンパク複合体に集合します。
それに比べ他の遺伝子、ORF3a、E、M、ORF6、ORF7a、NS7b、ORF8、Nのサイズは小さいです。ORF1abは巨大遺伝子、スパイク遺伝子は中程度のサイズの遺伝子、他の遺伝子は小さな遺伝子と考えて下さい。各遺伝子の大きさの違いを考えると、ORF1abが他の遺伝子よりも頻繁に変異するのは理にかなっています。

プロト-武漢と他のプロト-変異体の間での遺伝子変異の数はせいぜい数十程度です。このように、武漢型からプロト-変異体への変異は限定的です。また興味深い事に、プロト-変異体の変異はN変異に顕著に偏っています。
また、この中でもプロト-オミクロンは最も多くの変異を保有しており、1つの挿入と5つの欠失以外にも52の変異を持ち、そのうち30はスパイク遺伝子に位置しています (図2)。ここで注目すべきは、スパイクタンパクをコードする遺伝子の30変異のうち29変異がN変異である事です (図2)。オミクロンのS変異はORF1abに集中しています。
同様に他の変異体でも変異のほとんどはスパイク遺伝子とORF1abに集中しており、他の遺伝子にはほとんど変異がありません。さらに興味深い事には、プロト-オミクロンとプロト-ラムダ以外のプロト-変異体のスパイク遺伝子にはS変異が無いのです。

変異がS変異またはN変異になる可能性はコドンに依存します。そのため配列やサイズの異なる遺伝子の変異パターンを比較する際には、起こり得るN変異とS変異の数で標準化する必要があります。これらの数値はN部位、S部位と呼ばれ、その変異頻度は、N部位あたりのN変異の数 (dN) とS部位あたりのS変異の数 (dS) の比として表す事ができます。dN、dSの値とはそれぞれ「N変異、S変異の起こりやすさ」と解釈して下さい。標準化によってORF1ab、スパイク遺伝子以外の小さな遺伝子のN変異、S変異の起こりやすさも明確になります。
図3のように、SARS-CoV-1、RaTG13、プロト-武漢の間の進化をdN、dSで標準化するとdNに比べてdSがはるかに大きい事が分かります。これは進化の過程でN変異が競争に負けて淘汰された結果であり、つまり中立進化が働いたと考えられます。
また、SARS-CoV-1、RaTG13、プロト-武漢の進化の過程ではスパイク遺伝子でもdSはdNよりも高い事に注目して下さい。

すべてのプロト-変異株においてスパイク遺伝子のdNはdSよりも顕著に高いのです。こうした傾向はSARS-CoV-1、RaTG13、プロト-武漢の進化の過程では見られませんでしたので、スパイク遺伝子の中立進化からの逸脱はプロト-変異株の特徴です。一方スパイク遺伝子とは対照的に、プロト-デルタを除くすべての変異株ではORF1abのdNはdSより低いのです。
さらにほとんどのプロト-変異株にはスパイク遺伝子以外にもdNのみの遺伝子が存在しているのも奇妙です。なぜなら、それは武漢型からプロト-変異株に進化する間にアミノ酸配列を変えないS変異の蓄積も起こらなかった事を意味するからです。
RNAウイルスのdN/dS比は低い
SARS-CoV-1やRaTG13からの武漢型への進化と、武漢型から他のプロト-変異株への進化は大きくパターンが異なります。従って、これらのどちらのパターンが一般的なのかを知るためには、コロナウイルス同士だけではなく他の生物やウイルスの進化と比較する必要があります。

ヒトを含めた一般的な哺乳類やウイルスの進化と比較する事でSARS系統のコロナウイルスや変異株の進化を俯瞰する事ができます。このために私はNikolaevらが解析した17種の哺乳類系統のdN/dS比とLinらが解析した29種の異なるウイルスのdN/dS比を参考にしました (図5)。Linらの解析にはコロナウイルスが含まれていなかったため、Kistlerらによるヒト季節性コロナウイルスのRdRpとスパイクタンパク質をコードする遺伝子のデータも含めました。
本来、哺乳類とウイルスのゲノム間のdN/dS比は1よりもはるかに低くなります。実際レトロウイルスのdN/dS比は0.255、DNAウイルスは0.052〜0.254、RNAウイルスのdN/dS比は0.046〜0.075 (平均0.051) でした。このように、とりわけRNAウイルスはdN/dS比が低く、一般的なRNAウイルスではN変異よりもS変異の方が顕著に多いのです。これはRNAウイルスのゲノムがRNA修復の欠如に起因して突然変異率が高いためと考えられます。ほとんどのN変異は競争に負けて淘汰されてしまうからです。

SARS-CoV-1、RaTG13、プロト-武漢の平均dN/dS比は0.091でした (図2)。これは一般的なRNAウイルスでの傾向と類似しています。しかしプロト-変異株のdN/dS比は非常に高く、0.61 (プロト-ラムダ) から2.63 (プロト-デルタ) (平均1.19) でした (図6)。プロト-変異株のdN/dS比と一般的なRNAウイルスのdN/dS比の相違を分散分析するとp値は1.15 x 10⁻⁹であり、その違いは有意です。

図7はさらにそれぞれのウイルスの個々の遺伝子のdN/dS比を解析したものです。ほとんどのRNAウイルスの遺伝子のdN/dS比は0.1以下と非常に低く、RNAウイルスでは遺伝子レベルにおいても中立進化が優勢です。
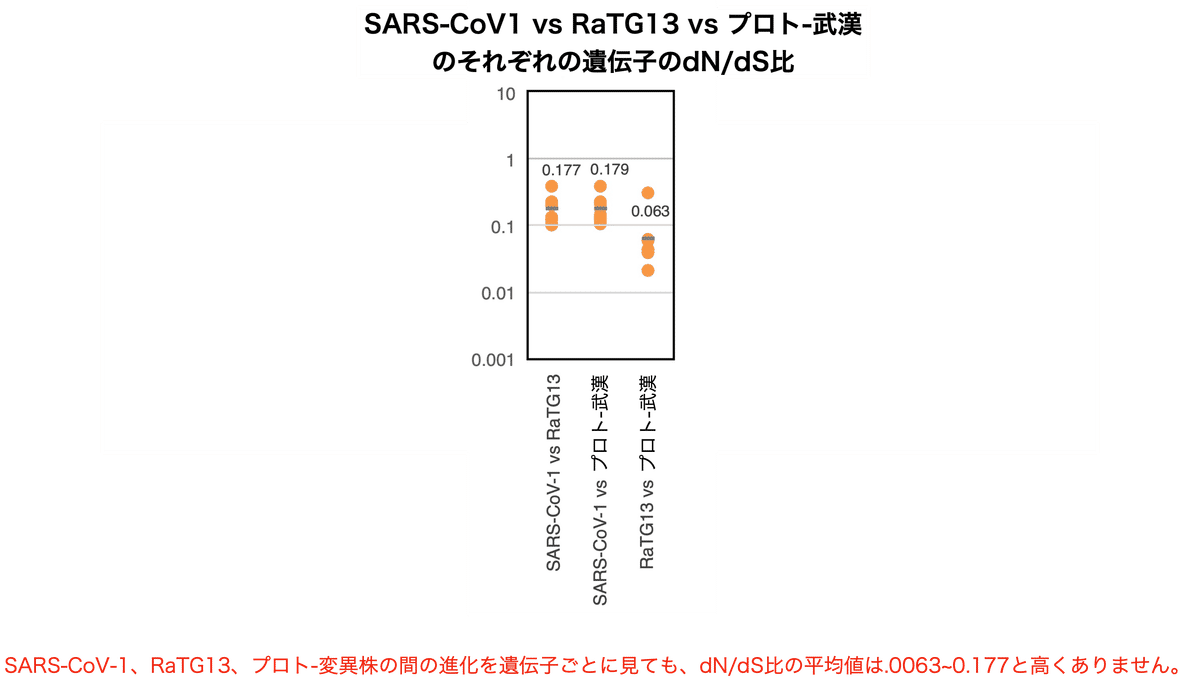
また、SARS-CoV-1、RaTG13、プロト-武漢の個々の遺伝子についてもdN/dS比は0.063から0.179と低く、中立進化の結果が見て取れます (図8)。

一方、プロト-変異株の遺伝子の多くはS変異を欠いていました (表1)。これも非常に奇妙な話なのです。なぜなら、本来S変異は遺伝子の機能には影響しないため、ウイルスが増殖する際の複製エラーによって時間とともに蓄積されるはずだからです。武漢株からプロト-オミクロンまで進化する間にも1つも変異を受けなかった遺伝子すら存在します。ところがプロト変異株の遺伝子の中にはS変異が無いにも関わらずN変異を含む遺伝子も多数あり、こうした遺伝子ではdN/dS比が無限大になります。
#コロナワクチン
#ワクチン
#コロナ
*記事は個人の見解であり、所属組織を代表するものではありません。
この記事が気に入ったらサポートをしてみませんか?
