ANCOM-BC2結果からボルケーノプロットを作成
ボルケーノプロットはlog fold changeと発現差の統計的有意性の関係性の可視化のため、遺伝子発現解析で広く用いられます。
今回は以前の記事(下記リンク参照)で実施したANCOM-BC2解析の結果からボルケーノプロットを作成します。
実行環境とソフト
実行環境:Ubuntu 22.04.2 LTS (GNU/Linux 5.15.146.1-microsoft-standard-WSL2 x86_64)
実行ソフト:QIIME 2 2023.9 Amplicon Distribution上のR version 4.2.2
※WSL上でQiime2をactivateし、RでR環境を立ち上げています。
conda activate qiime2-amplicon-2023.9
Rデータの準備
まず、ANCOM-BC2で得られたCSVファイルを読み込みます。
tmp <- read.csv("ancombc_family/ancombc_prim.csv")
de <- tmp[complete.cases(tmp), ]ボルケーノプロットの作成
早速簡単なボルケーノプロットを作ることができます。
#ggplotのインストール
install.packages("tidyverse")
library(ggplot2)
p <- ggplot(data=de, aes(x=lfc_X3groups1day, y=-log10(p_X3groups1day))) + geom_point() + theme_minimal()
pうまくいくと、簡単なボルケーノプロットが表示されます。

有意性の閾値を追加します。ここではlog fold-change 0.6とp値0.05にしていますが、好きな値に設定してください。
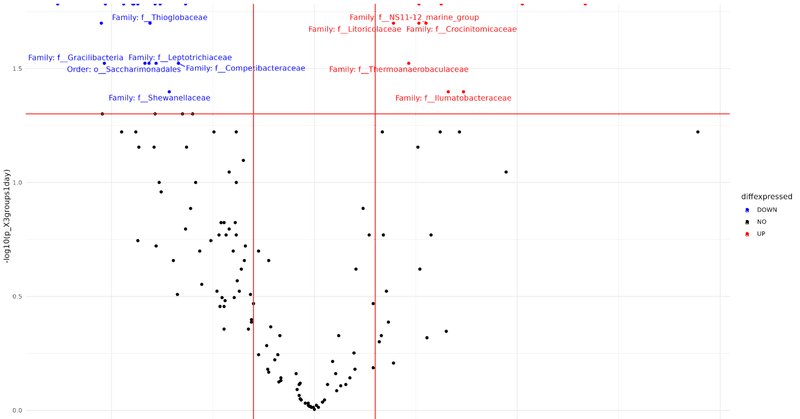
p2 <- p + geom_vline(xintercept=c(-0.6, 0.6), col="red") + geom_hline(yintercept=-log10(0.05), col="red")有意差のある細菌を同定し、図上で分類することもできます。
de$diffexpressed <- "NO"
de$diffexpressed[de$lfc_X3groups1day > 0.6 & de$p_X3groups1day < 0.05] <- "UP"
de$diffexpressed[de$lfc_X3groups1day < -0.6 & de$p_X3groups1day < 0.05] <- "DOWN"
#"diffexpressed"で色付け
p <- ggplot(data=de, aes(x=lfc_X3groups1day, y=-log10(p_X3groups1day), col=diffexpressed)) + geom_point()+ theme_minimal()
p2 <- p + geom_vline(xintercept=c(-0.6, 0.6), col="red") +
geom_hline(yintercept=-log10(0.05), col="red")
色付けとラベリングをすることができます。ggrepelパッケージを用いてラベリングの位置を美しくしています。
library(ggrepel)
ggplot(data=de, aes(x=lfc_X3groups1day, y=-log10(p_X3groups1day), col=diffexpressed, label=delabel)) +
geom_point() +
theme_minimal() +
geom_text_repel() +
scale_color_manual(values=c("blue", "black", "red")) +
geom_vline(xintercept=c(-0.6, 0.6), col="red") +
geom_hline(yintercept=-log10(0.05), col="red")
#pngファイルで保存
ggsave("volcano.png", height=9, width=16, device="png")有意差があり、大きく変化した細菌のみ(左上、右上)を色付けし、ラベリングしています。

まとめ
視覚的に強い印象を与えることができる、ボルケーノプロットを作成しました。
この記事が気に入ったらサポートをしてみませんか?
