Rを使っての重回帰分析(ステップワイズ) scikit-learnの糖尿病の元データを使って
これまではデータを使って、Pythonにて機械学習を行うことにより予測モデルを作成してきました。
機械学習ではなく統計解析を行っていくためにRでのデータ分析を始めることにしました。これで、手元にあるデータがどんな特徴を有しているのか説明することができます。時間に余裕があれば、Rでも機械学習を行っていきます。
今回は以前機械学習の重回帰にて使用したscikit-learnの糖尿病データを用いて統計解析していきます。
scikit-learnのデータは標準化かなにかの作業が施されているので、今回は元データを使うことにしました。
元データの場所
https://www4.stat.ncsu.edu/~boos/var.select/diabetes.html
Rの使用に関しては、Rstudioを使用してます。データ分析の際にPythonで扱うJupyter notebookではコードの実行がshif+Entに対し、Rstudioではctrl+Entなので、まだ慣れないためか結構間違えてしまいます。。。
そんな不満はおいといて、さっそく分析していきます。
Rstudioを立ち上げ、カレントディレクトリをセットします。
データの読み込みを行います。使用するデータはCSVファイルにして、カレントディレクトリにおいてます。CSVファイルの名前は、「diabities2.csv」にしました。
まずはデータの読み込みです。
dataset<-read.csv("diabities2.csv")
pythonでは"="でデータセットをいれますが、Rでは"<-"でデータなどを代数に入力します。
モデル1
重回帰のモデルを組みます。データセットに入っている項目のうち、説明変数には年齢、性別、BMI、血圧、血清のデータ( T細胞、低比重リポタンパク質、高比重リポ蛋白質、甲状腺刺激ホルモン、ラモトリギン、血糖)を入れ、目的変数にはベースラインから一年経過したときの糖尿病の進行度を入れます。
leg<-lm(target~age+sex+bmi+map+tc+ldl+hdl+tch+ltg+glu, data=dataset)目的変数のTarget、~以降は説明変数が入っています。これで重回帰分析のモデル作成完了です。結果を見てみます。
summary(leg)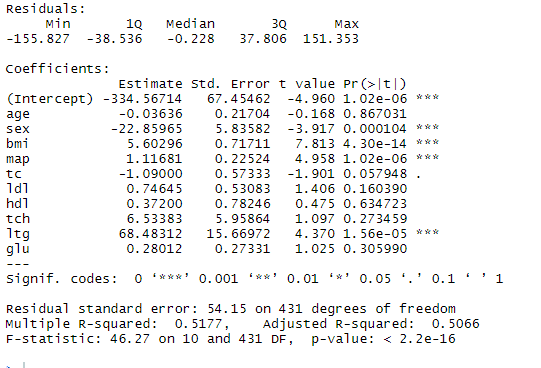
決定係数は0.5177、自由度調整済み決定係数は0.5066でした。
scikit-learnの糖尿病のデータでの重回帰②予測モデルの作成で作成したモデルについても同様に重回帰分析してみます。
モデル2・・・血清脂質を中心に考えたモデル
モデル3・・・甲状腺刺激ホルモンを中心に考えたモデル
モデル4・・・T細胞を中心に考えたモデル
モデル2の結果
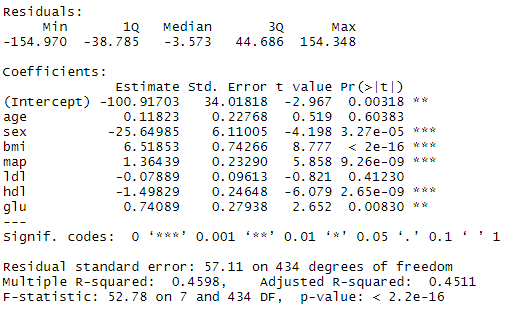
決定係数は0.4598、自由度調整済み決定係数は0.4511でした。
モデル3の結果
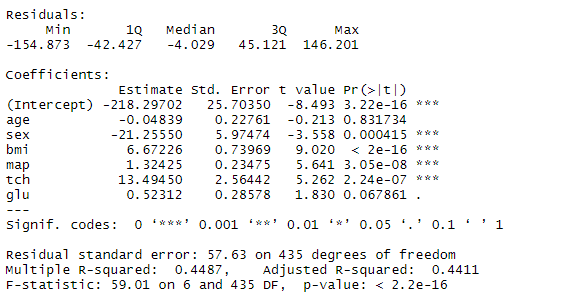
決定係数は0.4487、自由度調整済み決定係数は0.4411でした。
モデル4の結果
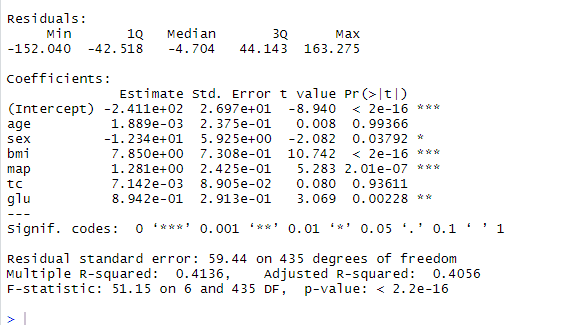
決定係数は0.4136、自由度調整済み決定係数は0.4046でした。
機械学習の時の精度ではすべての説明変数をいれて作成したモデルが一番高かったときと同様に、統計解析でもすべての説明変数をいれて作成した重回帰分析での決定係数が一番大きかったです。
ステップワイズ法
最後にステップワイズ法によって、AICを考慮して作成した重回帰のモデルの結果を見てみます。
ステップワイズ法を行うためにRのライブラリーであるMASSを用いました。
targetのモデルのみのAICと、その他の説明変数を入れた時のAICを見てみます。
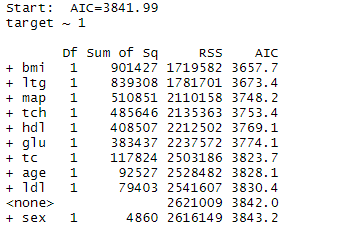
TargetののみではAICは3841.99でした。どうやらBMIを入れた時のAICが一番低いみたいです。
ステップワイズ法によって作成されたモデルの結果を見てみます。

結果、説明変数がBMI、ラモトリギン、血圧、低比重リポタンパク質にいれると一番重回帰のモデルにフィットするようです。
糖尿病の診断基準にあたる血糖が入っていないのが個人的に驚いています( ゚Д゚)。。。
このときの決定係数は0.5149、自由度調整済み決定係数は0.5082。若干ですが、説明変数の全ての項目を入れて行った重回帰分析の決定係数よりも小さいです。
統計分析ならば、交互作用の検定などが必要ですが、、、今回のはここまでにしておきます。
あ、ここまできて思いましたが、統計解析なのでp値などの値も見るべきでした。次から気を付けます。統計解析からはだいぶ離れてしまったので、見る目が相当落ちているようです。。。
同じようなデータでも機械学習とはみる観点が異なったりして、Rでの統計解析も面白いです。今後も継続して勉強していきます。
参考文献・資料
・Rによる多変量解析入門 川端一光ほか著 オーム社
・井出草平の研究ノート Rでステップワイズ回帰 https://ides.hatenablog.com/entry/2019/08/31/171340
