
正準相関分析~多変量同士の相関関係を捉える~
今回の記事では正準相関分析をRのコードを用いてとりあえず実践してみるということを目標としています。
1. 相関分析
まず初めに相関関係について紹介していきます。相関関係といえば、xという変数があり、かつyという変数が存在した時に、その関係性を相関係数であるrを用いて行う相関分析が有名です。相関係数のrは、両変数の共分散を各変量の標準偏差の積で割ると算出できます(ピアソンの積率相関係数)。下図のように、相関係数が大きいほど2つの変数間に強い関係性があることが分かります。また、相関係数がプラスかマイナスかで二つの変数の関係性が分かります。

2. 重回帰分析
それでは、次はxの変数が多数あり、yの変数が1つであった場合にはxとyとの関係性を見る場合にはどうしたらいいでしょうか。代表的な分析方法に重回帰分析がありますね。例えば、xの変数が3つあった場合には、
y = α + β1X1 + β2X2 + β3X3
という式で表すことで変数yとxのそれぞれの変数との関連を調べることができます。xの変数を標準化して上記の式に当てはめれば、βの符号と大きさからyへの関連の強さが分かります。例えば、このβ1という値によって、x1の値が1上がった時に、yにどれくらい影響を与えているかわかります。
3. 正準相関分析
さらに進めると、もしyにおいても複数の変数があった場合にはどうすべきでしょうか。例えば、以下のような関係です。

この時のu(x)とv(y)の関係性を見ようとした際には、先ほど紹介した相関分析や重回帰分析では解析できません。そこで登場するのが今回の主役である正準相関分析です。英語にすると、Canonical Correlation Analysis(CCA)です。名前の響きだけでカッコいいですね。
正準相関分析に関する詳しい説明に関しては、以下の文献に詳しく記載されています。
・赤穂昭太郎. (2013). 正準相関分析入門—複数種類の観測からの共通情報抽出法—. 日本神経回路学会誌, 20(2), 62-72.
・Ishihara, et al., (2022). Association of cardiovascular risk markers and fitness with task-related neural activity during animacy perception. Medicine and Science in Sports and Exercise, 54(10), 1738.
4. 正準相関分析をRを用いてコーディング
今回は、この正準相関分析の中でも、sparse multiple CCAというペナルティを用いた複数の要因が存在する正準相関分析についてRのコードを用いて紹介していきます。
正準相関分析の種類とその用途については、以下の文献に詳しく記載されています。
・Wang, et al., (2020). Finding the needle in a high-dimensional haystack: Canonical correlation analysis for neuroscientists. NeuroImage, 216, 116745.
・Zhuang, et al., (2020). A technical review of canonical correlation analysis for neuroscience applications. Human Brain Mapping, 41(13), 3807-3833.
4.1. パッケージのインストール
まずは必要なパッケージをインストールします。今回はPMAパッケージのmultiCCAという機能を使って正準相関分析を行なっていきます。
PMAパッケージのmultiCCAという機能の説明については、以下のリンクをご参照ください。
・Package ‘PMA’
・multiCCA
rm(list = ls())
library(PMA)
library(ggplot2)
library(ggExtra)4.2. データセットの作成(乱数発生による擬似データ)
次に擬似データ(正規分布による乱数発生)の生成を行なっていきます。今回は、x1、x2、x3、x4という中に50行10列のデータセットが入っています(想定としては、50名のデータセットに10個の説明変数が入っているというイメージです)
# Data set creation
set.seed(123)
x1 <- matrix(rnorm(50 * 10), nrow = 50, ncol = 10)
x2 <- matrix(rnorm(50 * 10), nrow = 50, ncol = 10)
x3 <- matrix(rnorm(50 * 10), nrow = 50, ncol = 10)
x4 <- matrix(rnorm(50 * 10), nrow = 50, ncol = 10)おまけですが、最後に図表にする際に0と1の変数を用いた散布図を作りたいと思うので、以下の変数を作っておきます(イメージとしては、0が女性で1が男性という性別のデータが入っているイメージです)
v6 <- matrix(c(rep(1,25),rep(0,25)),ncol=1)
v6 <- as.factor(v6)4.3. Permutation testのための並べ替え
今後は、正準相関分析に対してPermutation testを行うための並び替えの操作を行います。Permutation testとはノンパラメトリックの検定方法の一つで、サンプルを並び替えて行うことにより、正準相関分析において算出された相関係数が帰無仮説を棄却するかどうか判定するために使います。ここでの帰無仮説は、正準相関分析において算出された相関係数とランダムにサンプリングされた相関係数が一緒ということになります。
Permutation testについての詳しい説明は、以下の文献やサイトをご参照ください。
・Winkler, et al., (2020). Permutation inference for canonical correlation analysis. Neuroimage, 220, 117065.
・Permutation test
#permutation test_並び替え
N <- 10
corPerm_1 <- numeric(length = N)
corPerm_2 <- numeric(length = N)
corPerm_3 <- numeric(length = N)
corPerm_4 <- numeric(length = N)
corPerm_5 <- numeric(length = N)
corPerm_6 <- numeric(length = N)
corPerm_total<- numeric(length = N)
for(i in 1:N)
{
#並び替え
x1_sample <- matrix(sample(c(x1)), nrow = 50, ncol = 10)
x2_sample <- matrix(sample(c(x2)), nrow = 50, ncol = 10)
x3_sample <- matrix(sample(c(x3)), nrow = 50, ncol = 10)
x4_sample <- matrix(sample(c(x4)), nrow = 50, ncol = 10)
#リストに格納
xlist_sample <- list(x1_sample, x2_sample, x3_sample, x4_sample)
#並び替えデータセットにおけるSMCCA
# Execute MultiCCA.permute_sample
perm_out_sample <- MultiCCA.permute(xlist_sample, nperms=10)#10permutations
# Execute MultiCCA_sample
out_sample <- MultiCCA(xlist_sample, penalty=perm_out_sample$bestpenalties)
#回帰モデルへの当てはめ_sample
x_1_sample <- x1_sample%*%out_sample$ws[[1]]
x_2_sample <- x2_sample%*%out_sample$ws[[2]]
x_3_sample <- x3_sample%*%out_sample$ws[[3]]
x_4_sample <- x4_sample%*%out_sample$ws[[4]]
corPerm_1[i] <- cor(x_1_sample,x_2_sample)
corPerm_2[i] <- cor(x_1_sample,x_3_sample)
corPerm_3[i] <- cor(x_1_sample,x_4_sample)
corPerm_4[i] <- cor(x_2_sample,x_3_sample)
corPerm_5[i] <- cor(x_2_sample,x_4_sample)
corPerm_6[i] <- cor(x_3_sample,x_4_sample)
corPerm_total[i] <- corPerm_1[i]+corPerm_2[i]+corPerm_3[i]+corPerm_4[i]+corPerm_5[i]+corPerm_6[i]
}今回の場合、まずfor文で10回並び替え作業を行うことを宣言します。加えて、multiCCAで算出された係数を使った変量の相関係数を格納するデータフレームを作成します(corPerm_)。並び替えはsample関数を用いました。MultiCCA.permuteを使って最適なペナルティを求めて、そのペナルティをMultiCCAに適用しています。x_1_sample <- x1_sample%*%out_sample$ws[[1]]というようにMultiCCAで算出した係数をx_1_sampleに格納しています。corPerm_1[i] <- cor(x_1_sample,x_2_sample)にそれぞれの変数に対する相関係数を入れていきます。corPerm_total[i]にはcorPerm_1からcorPerm_6までの相関係数の総和を格納しています。
4.4. sparse multiple CCAの実行
今回のメインとなるsparse multiple CCAを作成したデータセットに当てはめていきます。
sparse multiple CCAを用いた研究の紹介
Ishihara, et al., (2024). The links between physical activity and prosocial behavior: an fNIRS hyperscanning study. Cerebral Cortex, bhad509.
上記の研究では、身体活動量と向社会的行動との関係を脳機能の介在という点から明らかにしています。非常に興味深い研究知見ですので、是非ともお読みいただくことを心よりお勧めします。
#リストに格納
xlist <- list(x1,x2,x3,x4)
# Execute MultiCCA.permute
perm.out <- MultiCCA.permute(xlist, nperms=10)#10permutations
print(perm.out)
# Execute MultiCCA
out <- MultiCCA(xlist, penalty=perm.out$bestpenalties)
print(out)
#回帰モデルへの当てはめ
x_1 <- x1%*%out$ws[[1]]
x_2 <- x2%*%out$ws[[2]]
x_3 <- x3%*%out$ws[[3]]
x_4 <- x4%*%out$ws[[4]]
#相関係数の合計
cor_1=cor(x_1,x_2)
cor_2=cor(x_1,x_3)
cor_3=cor(x_1,x_4)
cor_4=cor(x_2,x_3)
cor_5=cor(x_2,x_4)
cor_6=cor(x_3,x_4)
cor_total=cor_1+cor_2+cor_3+cor_4+cor_5+cor_6流れとしては、4.3.と同じ流れになっています。まずlistにそれぞれの変量を格納して、MultiCCA.permuteで最適なペナルティを求めたのちに、MultiCCAで係数を求めていきます。x_1 <- x1%*%out$ws[[1]]でMultiCCAで求めた係数とn次方程式(ここではn=50)を作成したのちに、それぞれの変量について相関係数を求めていきます。最後に、その相関係数の総和を計算します。
4.5. Permutation testの実行
並び替えによって求めた相関係数と対象となる相関係数とについてPermutation testを実行します。
#permutation_testの結果図示
results.df <- data.frame(x = unlist(corPerm_total))
ggplot(results.df,aes(x)) +
geom_histogram(color="darkgreen",fill="lightseagreen") +
geom_density(fill='green', alpha=0.3) +
geom_vline(xintercept = cor_total, lwd=1, lty=2) +
xlab("sum of correlation coefficients")+
ylab("Density")+
scale_x_continuous(limits = c(-2, 5)) +
theme_classic()+
theme(axis.text.x = element_text(size = 15),
axis.text.y = element_text(size = 15),
axis.title.x = element_text(size = 15),
axis.title.y = element_text(size = 15))
cor_total#2.249144
corPerm_total
p_value_Cor <- sum(unlist(corPerm_total)>2.249144)/length(corPerm_total)
p_value_Corと下の図が描かれます。
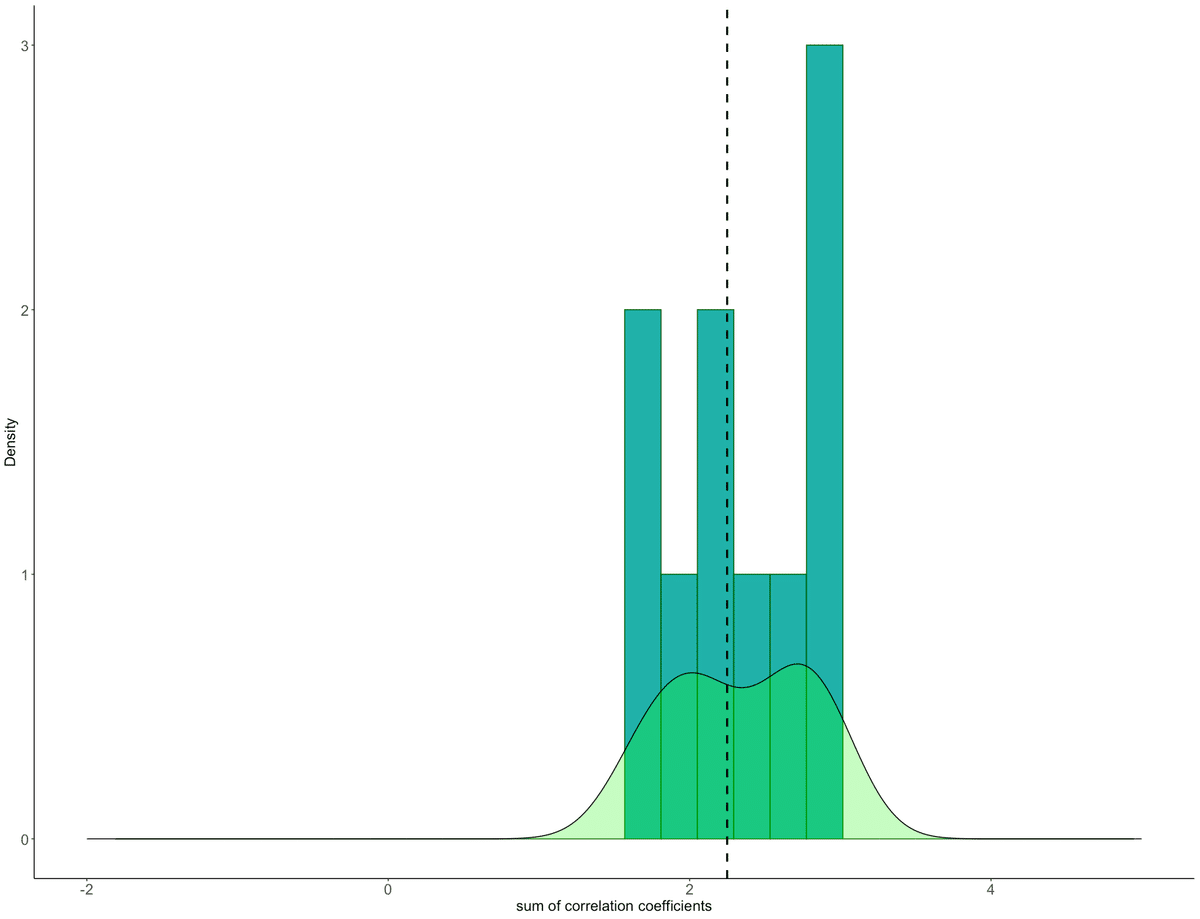
x軸が2.5付近のところにある垂直の点線が対象となる相関係数の総和を示しています。乱数発生によるPermutation testを行っているので、相関係数の総和がヒストグラムの中央に来ています。なので、p_value_Corを求めると、0.5となります。
4.6. おまけ: 変量同士の関係を図示
最後に、MultiCCAによって求めたx_1とx_2の相関図を書いてみます。回帰直線と95%信頼区間を加えます。また、ヒストグラムと確率密度関数の図も加えます。今回、v6という0と1の2値で表した変数を入れて、それぞれに対応する点も図示してみます。
data<-data.frame(x_1,x_2,x_3,x_4,v6)
# 散布図
scatter_plot_color <- ggplot(data,aes(x = x_1, y = x_2))+
geom_point(aes(x=x_1, y=x_2, color = v6)) +
geom_smooth(method = "lm", color = "red",
linetype = "dashed",
se = TRUE,
size = 0.5,
fill = "pink")+
xlab("x_1") +
ylab("x_2") +
theme_classic()
# マージナルヒストグラム
ggMarginal(scatter_plot_color, type = "densigram", groupFill = TRUE)
このコードを実行すると下の図を描くことができます。

5.まとめ
今回の記事では、正準相関分析をRを用いて実際に実装してみました。理論についてはまだまだ勉強中ですので、引き続き正準相関分析について理解を深めていきたいと考えています。また、Permutation testにつてもまだまだ理解不足で引き続き勉強中です。「ここが間違っている」とか「もっとこうした方がいい」というご指摘ありましたら、是非是非是非教えていただきたいです。とりあえず、正準相関分析をRで実装するということが今回の目的なので、上記に示したコードが間違っていましたら大変申し訳ございません。誤っている点が見つかり次第、その都度修正していきます。
最後までお読みいただき誠にありがとうございました。再三の記載になりますが、本記事には誤っている箇所があるかもしれませんので、本コードをご使用の際には適切かどうか確かめていただくようお願いいたします。また、間違っている点や修正点等ございましたら、ぜひご連絡いいただけると幸いです。X(旧:Twitter)のSogachin23(@sogachin2)のDMを解放していますので、ぜひご連絡ください。よろしくお願い申し上げます。
