
【R言語】ggplot2|今まで使っていたデータが使えない?CSVファイルを取り込む
こんにちは。プログラミング超初心者のえいこです。
前回はggplot2をインストールして、サンプルデータを使って描画していきました。
次は、自分のデータを使って!...と意気込んでいままで使っていたデータフレームを使って挑戦したものの、エラーが出て描画できませんでした。
いろいろ調べてみたところ、どうやら今まで使っていたデータを少し書き換えないといけないことが発覚しました。
今回は、ggplot2用にデータの形を整えることをしてみようと思います。
今まで使っていたデータフレームを使うとエラーを吐き出す
ggplot2はRStudioを開発した人が作ったものだし、RStudioで動いていたデータフレームで動くだろうと何も考えずにスタートしてしまいました。
まず、今まで使っているデータを振り返ります。

これを”protein.a”にデータフレームとして格納していました。
> head(protein.a)
Control Treated
1 4.02 5.23
2 3.75 6.12
3 3.68 5.79このデータフレームを使って、グラフを描くときの第一歩である平均値、標準偏差を計算していこうと思ったのですが...
調べたものを打ち込んでもエラーが吐き出されました...
> s.data<-
+ protein.a %>%
+ group_by(colnames(protein.a)) %>%
+ summarize(mean=round(mean(protein.a,),2),S.D.=(sd(mean(protein.a))))
エラー: Column `colnames(protein.a)` must be length 3
(the number of rows) or one, not 2はぁ~~?よくわからない!!
となって、もう少し調べてみたところ。
処理の方法を縦にまとめないと駄目なことが発覚!これ、Rにもともと入っているデータフレームの"iris"の形もそうなっていますね。
処理の方法を縦にまとめるって何?
日本語としてよくわからない表現になってしまったので、具体的なデータの形を見てみようと思います。
> is.data.frame(iris) #irisというデータがあるのか確認
[1] TRUE
> str(iris) #データフレームを表示
'data.frame': 150 obs. of 5 variables:
$ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
$ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
$ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
$ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
$ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
> head(iris) #データフレームの最初の方を表示
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
> irisのデータは実は150行あるのですが、"head()"を使うと最初の6行をコンソール画面に表示させることができます。
ここで大事なのは、列の一番右側が"Species"となっていてサンプルと対応して変数が指定されていることです。
ということは、Rを動かす上ででのデータの形としては、
サンプルに番号をつける→計測データの格納、種類の振り分けを列で行う
が処理しやすいということみたいです。
今まで使っていたデータを書き直す必要がありそう。
エクセルを使ってデータを書き直す
Rの上でいちいち書き直すのはめんどくさい...
ということで、エクセルで作りなおしたデータをRに取り込んでみようと思います。
今回作りなおした表はこんな感じです↓
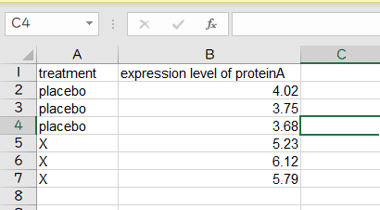
このファイルを".csvファイル(CSV(コンマ区切り))"として保存しておきます。(xtreatment.csvという名前で保存しました)
RStudio上で、カレントディレクトリをCSVファイルを保存した場所に設定しておきます。(そうしないとこんなエラーが出てしまいます)
> rawdata<- read.csv("xtreatment.csv",header=T)
file(file, "rt") でエラー: コネクションを開くことができません
追加情報: 警告メッセージ:
file(file, "rt") で:
ファイル 'xtreatment.csv' を開くことができません: No such file or directory csvファイルのデータを"rawdata"という変数に入れようとしたら「ファイルがありません」と警告されてしまいました。
全然知らなかったのですが、プログラミングの世界では現在地というのがとっても重要なようで、現在地から見えるところからしかファイルを引っ張ってこられないようなのです。
なので、現在地を移動してファイルを見えるようにしてから読み込まなければなりません...
一般的にコードを打ち込めば、カレントディレクトリを移動できるようです。Rにも"setwd"といった関数が用意されているようですが、RStudioでは「Session→Set Working Directory→Choose Directory」で変更することができます。(コードをいろいろ覚えなくても良いのは初心者にとってはありがたい!!)
> setwd("~/R/data_frames")
> #作ったcsvファイルを取り込む
> rawdata<- read.csv("xtreatment.csv",header=T)自動的に"setwd"というコマンドが実行されてファイルを入れていた場所に移りました。"read.csv"を実行しても今回はエラーが出ませんでした。
最後に、取り込んだデータの構造を確認して今回はおしまいにしようと思います。
取り込んだCSVファイルの確認
データフレーム構造を確認するには"str"を使います。
> str(rawdata)
'data.frame': 6 obs. of 2 variables:
$ treatment : Factor w/ 2 levels "placebo","X": 1 1 1 2 2 2
$ expression.level.of.proteinA: num 4.02 3.75 3.68 5.23 6.12 5.79基本的なデータフレームの形は、6つのオブジェクトで、2種類の変数。
treatmentの要素には"placebo"と"X"の二つがあり、expression.level.of.proteinA要素は数値データが入っていますよ。
という情報が読み取れます。
データフレームの最初の方を見るためには、"head()"を使うんでした。headを使って確認すると...
> head(rawdata)
treatment expression.level.of.proteinA
1 placebo 4.02
2 placebo 3.75
3 placebo 3.68
4 X 5.23
5 X 6.12
6 X 5.79おぉ!良い感じ♪
これを使って、ggplot2でグラフを描いていきたいと思います。
それでは、また!
最後までお読みいただきありがとうございます。よろしければ「スキ」していただけると嬉しいです。 いただいたサポートはNGS解析をするための個人用Macを買うのに使いたいと思います。これからもRの勉強過程やワーママ研究者目線のリアルな現実を発信していきます。
