ggplot2: 散布図にラベルを表示
散布図に表示された点(ドット)が、どのデータを示しているのか、ラベルを表示したい時があるかと思います。表示したいラベルが複数の時は、それらの重なりまで考慮すると配置が難しいこともあります。そのような時は、 ggrepel パッケージを使用すると便利です。
使い方は、 ggplot() + geom_point() にプラス(+) で続けて、 geom_text_repel() を指定するだけです。
ボルケーノプロットに遺伝子名を表示する
例として、前回作成したボルケーノプロット上に、変動した遺伝子の名前 (GeneID) を表示させてみましょう。ここでは、変動した遺伝子 (deg = differentially expressed genes) のうち、増加したものから10個、減少したものから10個を選んで用います。
前準備(上位の遺伝子のデータを取得)
前準備として、遺伝子名を表示したいデータを準備します。前回、変動した条件 (PValue, logFC) を満たす遺伝子をフィルタリングして取得しているので、それを用います。 top_n() 関数を使って、上位 10 個を取得できます。ボルケーノプロットでは、 p-value が「小さい」ほど、y軸の値は大きくなるので、マイナスをつけて、取得しています。(小さい方から10個)
top10_deg が増加したほうから10個、bottom10_deg が減少したほうから10個の遺伝子を取得したものです。これらを bind_rows() で連結して、 marker_deg とします。
top10_deg <- up_deg %>% top_n(10, -PValue)
bottom10_deg <- down_deg %>% top_n(10, -PValue)
marker_deg <- top10_deg %>% bind_rows(bottom10_deg)marker_deg テーブルの中身は、下記のようになっています。
> marker_deg
# A tibble: 20 × 5
GeneID logFC logCPM PValue FDR
<chr> <dbl> <dbl> <dbl> <dbl>
1 MTND1P23 9.06 7.65 3.01e-51 8.39e-47
2 CDH3 7.27 5.80 1.44e-31 1.34e-27
3 CPNE7 7.21 4.76 9.51e-25 6.63e-21
4 RP11-750B16 6.26 5.83 1.27e-24 7.08e-21
5 ETV4 5.67 5.82 7.52e-24 3.50e-20
6 CLDN2 8.57 6.92 2.16e-22 7.54e-19
7 FABP6 6.83 3.42 7.92e-19 2.01e-15
8 GRIN2D 5.33 4.53 1.01e-18 2.16e-15
9 CLDN1 4.88 5.53 1.84e-18 3.20e-15
10 RHPN1 4.56 4.74 1.01e-16 1.12e-13
11 CD177 -9.98 8.26 1.74e-32 2.43e-28
12 MT2A -4.63 7.07 7.08e-23 2.82e-19
13 SLC6A19 -8.82 5.15 3.64e-22 1.13e-18
14 BEST4 -7.08 5.66 3.12e-19 8.70e-16
15 MT1H -7.22 4.55 9.67e-19 2.16e-15
16 HSD17B2 -4.50 6.36 1.71e-18 3.20e-15
17 OTOP2 -9.09 5.79 1.83e-18 3.20e-15
18 SFRP1 -8.16 5.24 4.52e-18 7.28e-15
19 RNF152 -4.98 5.12 4.70e-18 7.28e-15
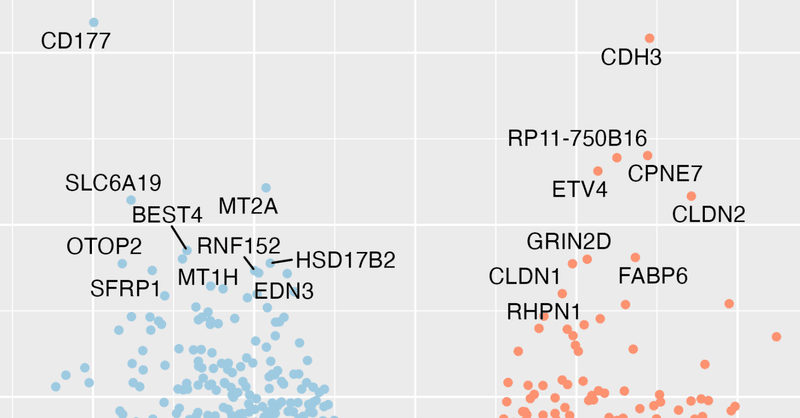
20 EDN3 -4.86 5.18 6.30e-18 9.25e-15geom_text_repel()関数でラベルを表示
前回のコードに追加する形で、geom_text_repel()を記述すると下記のようになります。geom_text_repel() のオプションの data に、前述の marker_deg を指定します。また、 label オプションにラベルに使用する文字列として、marker_deg$GeneID を指定します。max.overlaps のオプションは、ラベルの重なりを回避するためのものです。
g + geom_point(data = non_deg, color = "#969696") +
geom_point(data = up_deg, color = "#FC9272") +
geom_point(data = down_deg, color = "#9ECAE1") +
geom_text_repel(data = marker_deg,
label = marker_deg$GeneID,
max.overlaps = Inf)下記のように、遺伝子名のラベルを追加したボルケーノプロットが表示されます。

この記事が気に入ったらサポートをしてみませんか?
