
「1か月前はイギリスも日本と同じだった!」は本当なのか? データで確認 [Pythonコードあり]
コロナウイルスに関する記述があります。
筆者は感染症の専門家ではありませんので、不正確な情報かもしれません。
データサイエンスの観点のうち、データの取得、グラフの形状的なことにこの記事では集中します。
ーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーー
2020/05/04 追記
2020/04/26から約一週間後にレビューの結果を入れました。
--------------------------------------------------------------------------------------
以前、「日本は3週間前のアメリカ」という動画がありましたが、「増加率の違い」のためにアメリカほどの極端な増加は起こりませんでした。

上のグラフは青の日本の感染者の推移は、その時言われた緑のアメリカの感染者推移のようにはならなかったということは事実として、確認しておく必要があると思います。
感染者数は増加して、危険な状態には間違いないですが、予測された範囲内ので挙動なのではないでしょうか。
今回メディアで「1か月前はイギリスも日本と同じだった!」連休前にニュース番組が注目した“衝撃”の事実」なる記事が出てきました。
ニューヨークの記事の二番煎じの記事で「まあ、でてくるだろうな、、」と思っていました。前回ニューヨークの時と同様に別の記事で粛々と確認していきたいと思います。
前回同様 Kaggle -- COVID-19 Complete Dataset (Updated every 24hrs) のデータを使って確認していきます。
いくつかの記述はこちらの記事を参考にしてください。
「日本は3週間前のアメリカ」という動画は本当なのか?Python で感染者数など比較 [Pythonコードあり]
今回はアメリカのデータではなく、イギリスのデータを使用しますので、いくつか記述の変わる部分もありますので、最初からやっていきます。
Kaggle -- COVID-19 Complete Dataset (Updated every 24hrs)
https://www.kaggle.com/imdevskp/corona-virus-report/data
を利用させていただきました。このサイトに行きますと、以下のような画面になります。

下段には詳細なデータが出ていたりするのですが、ここではダイレクトにノートブックを利用して、自分で解析を行います。
右上の「New Notebook」を押していただき、ダイレクトにデータをGoogle Colaboratoryのように解析できる環境に移動します。


いつも通り[Shift]+[Enter]で実行するとPythonでデータの解析ができます。
データはKaggle Notebooksさんの方で準備してくれています。
/kaggle/input/corona-virus-report/usa_county_wise.csv
/kaggle/input/corona-virus-report/covid_19_clean_complete.csvにあるということなので、それを読み込んで作業の下準備を行います
corona_df = pd.read_csv('/kaggle/input/corona-virus-report/covid_19_clean_complete.csv', index_col=0)
display(corona_df.head(10).append(corona_df.tail(10)))
display(corona_df["Country/Region"].unique())


前回は日本とアメリカのデータでしたが、今回はイギリスのデータを使いたいので、'United Kingdom'のデータだけ抜き出します。
corona_df_JPN=corona_df[corona_df["Country/Region"]=="Japan"].copy()
corona_df_UK=corona_df[corona_df["Country/Region"]=="United Kingdom"].copy()
corona_df_JPN["日付"] = list(map(lambda x: pd.datetime.strptime(x, '%m/%d/%y'),corona_df_JPN["Date"]))
corona_df_UK["日付"] = list(map(lambda x: pd.datetime.strptime(x, '%m/%d/%y'),corona_df_UK["Date"]))
display(corona_df_JPN.head().append(corona_df_JPN.tail()))
display(corona_df_UK.head().append(corona_df_UK.tail()))

前回と違い地方・地域名まで、細かく出ているようです。いったんここでは総合値と思われる「Province/State 」が「NaN」の行だけ取り出したいと思います。
corona_df_JPN=corona_df_JPN.reset_index()
corona_df_UK=corona_df_UK.reset_index()
corona_df_UK=corona_df_UK[corona_df_UK["Province/State"].isnull()]
display(corona_df_UK.head().append(corona_df_UK.tail()))

その二つのデータテーブルを日付を基準にマージします。
corona_df_JPN=corona_df_JPN.set_index('日付')
corona_df_UK=corona_df_UK.set_index('日付')
df_merge=pd.DataFrame(index=corona_df_JPN.index)
df_merge=pd.merge(corona_df_JPN[["Confirmed","Deaths","Recovered"]], corona_df_UK[["Confirmed","Deaths","Recovered"]], on='日付',suffixes=['_JPN', '_UK'])
display(df_merge.head().append(df_merge.tail()))

今回は2020-03-23のイギリスと死者数と2020-04-23の日本の死者数が同じという記事ですので、まずその値が正しいのか確認します。
date1=df_merge.index.get_loc('2020-3-23')
date2=df_merge.index.get_loc('2020-4-23')
display(df_merge.iloc[[date1,date2],:])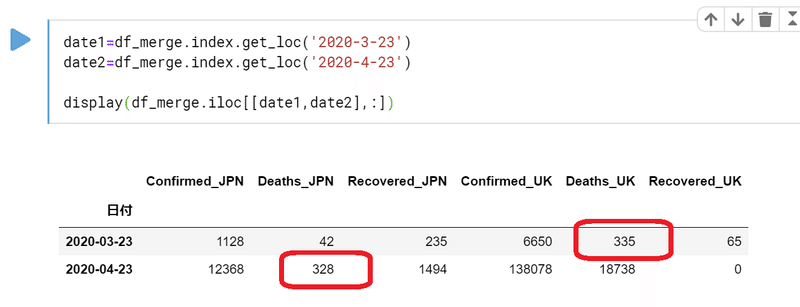
ほぼ同じような数字になっているようです。それでは前回同様日付をイギリスの死者数をシフトした行を一行作ります。単純にシフトすると将来の日付部分が欠損しますので、いったんダミーデータを入れて行を増やしたのちにずらしたデータを入れます。今回は増やす行は31日とします。
import numpy as np
import matplotlib.dates as mdates
from dateutil.relativedelta import relativedelta
for i in range(1,15):
MDtmp=max(df_merge.index) + relativedelta(days=1)
df_merge.loc[MDtmp]=np.nan
df_merge["SHIFT_Deaths_UK"]=df_merge["Deaths_UK"].shift(31)
display(df_merge.head().append(df_merge.tail()))
display(df_merge.iloc[[date1,date2],:])

それでは、これを前回同様プロットします。
import matplotlib.pyplot as plt
import matplotlib.dates as mdates
fig = plt.figure()
plt.rcParams["font.size"] = 18
mmdd=mdates.DateFormatter('%m/%d')
fig, (ax1 ,ax2 )= plt.subplots(2,1,figsize=(10,9),
gridspec_kw = {'height_ratios':[1, 1]},sharex=True)
ax1.plot(df_merge.index,df_merge["Deaths_JPN"],label="Deaths_JPN")
ax1.plot(df_merge.index,df_merge["Deaths_UK"],label="Deaths_UK")
ax1.plot(df_merge.index,df_merge["SHIFT_Deaths_UK"],label="SHIFT_Deaths_UK")
ax1.legend(loc='center left', bbox_to_anchor=(1, 0.5), fontsize=18)
ax1.grid(True)
ax2.plot(df_merge.index,df_merge["Deaths_JPN"],label="Deaths_JPN")
ax2.plot(df_merge.index,df_merge["Deaths_UK"],label="Deaths_UK")
ax2.plot(df_merge.index,df_merge["SHIFT_Deaths_UK"],label="SHIFT_Deaths_UK")
ax2.set_yscale('log')
ax2.legend(loc='center left', bbox_to_anchor=(1, 0.5), fontsize=18)
ax2.grid(True)
ax2.xaxis.set_major_formatter(mmdd)
fig.show()
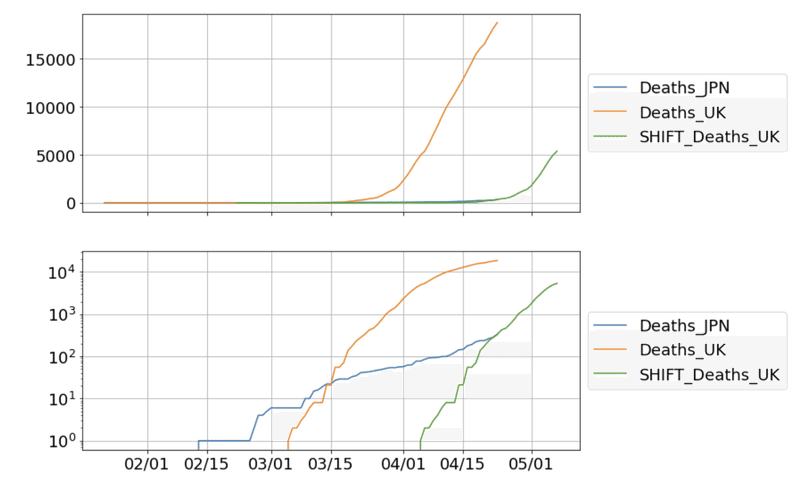
日本の死者数が青、日付をずらしたイギリスの死者数が緑になります。
これからの数日で、どのような推移を描くのか注目していきたいと思います。
ーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーー
2020/05/04追記

記事を書いてから約一週間経ちましたが、やはりというか、予想通りイギリスの死者数のようにはなっておりません。増加率が違いますから、まあ極端な集団感染がない限りは同じにはなりませんよね。
ーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーーー
急激に感染者が増えているのは事実ですし、全国的に外出を控え、医療関係者の負担を減らす必要があると思います。
311の原子力・放射能デマに日本人の多くは振り回されました。
しかも、まだその当時の総括すらできていません。
あの時に人々の不安に付け込んで、恐怖を煽った人たちに科学は負けてはいけないと思います。
放射性物質の半減期のような厳密な物理現象と違って、国民の意識や、行動によって結果が変わる感染症予測などは、正確な予測値を出すのが極めて難しいと思われます。
そのような中で、
「ツールとして利用していただき」、
「いたずらに不安を煽らず」、
「備えるために必要な情報」
としていただければと思います。
サポートしていただき大変ありがとうございます。 励みになります。
