
【R Studio】グループ化した棒グラフに実測値を重ねたグラフの作り方【ggplot2】
概要
本記事では、Rのggplot2を使って次のようなグラフを作成したい人に向けて、コードの書き方を紹介する。
2つの群について、複数の条件で行った実験結果をもとに、平均値を表す棒グラフをグループ化し、エラーバーと生の実測値を重ねてプロットする
最終的に下のようなグラフが作成できる。

背景
複数の群と複数の条件で行った実験の結果を可視化するには、理解しやすいグラフが必要である。方法としては、一つ一つの群と条件について棒グラフで表したり、ファセットを利用したりすることが考えられるが、これらは文字が多くなるとか、やや煩雑な見た目になるといった問題がある。そこで、棒グラフのグループ化を行うことで、直感的な理解がしやすくなる。
近年は、棒グラフや箱ひげ図にデータの実測値を重ねてプロットすることが一般的となってきている。しかし、グループ化した棒グラフと実測値プロットを組み合わせるのは容易ではない(と私は思う)。
本記事では、そのようなグラフを作成する方法を一手順ずつ解説する。
なお、今回は「グループ化した上で棒グラフを描くこと」が主題なので、基本的な棒グラフの作り方については、以前の記事を参照されたい。
データの準備
データは下のExcelファイルのようにしてまとめる。
このダミーデータは、ControlとAという2つの遺伝子型に対して、DMSO(コントロールに相当)、薬剤X、薬剤Yを処理した際に細胞数がどのように変化するかという実験の結果を想定している。
groupの列にはControlとAという2つの群、treatmentの列にはDMSO、X、Yという3つの処理方法、valueの列には各群と処理に対応する細胞数が記録されている。
はじめに、今回用いる3つのパッケージを読み込む。
library(openxlsx)
library(ggplot2)
library(dplyr)データの前処理
データを読み込む。すぐ後にデータの順番を変更するため、ここでは一旦data0という名前で格納する。
data0 <- read.xlsx("sample.xlsx")通常棒グラフはアルファベット順に表示されるので、2本の棒グラフのうち、Aが左側、Controlが右側に表示される。しかし、Controlが左にあったほうが自然なので、あらかじめここで表示順を決めておく。さきほどのdata0をもとに、factorを用いてグループを並べ替える。
アルファベット順のままで困らなければ、この操作は必要ない。
data <- transform(data0, group = factor(group, levels = c("Control", "A")))パイプ演算子を用いて、データの要約を行う。データを群と処理ごとにグループ分けし、平均値を求める。
data.summary <- data %>% group_by(group, treatment) %>% summarize(n = n(), sd = sd(value), value = mean(value)) %>% mutate(se = sd / sqrt(n))> data.summary
# A tibble: 6 × 6
# Groups: group [2]
group treatment n sd value se
<fct> <chr> <int> <dbl> <dbl> <dbl>
1 Control DMSO 10 68.9 409. 21.8
2 Control X 10 44.8 493. 14.2
3 Control Y 10 48.2 416. 15.2
4 A DMSO 10 48.8 186. 15.4
5 A X 10 52.2 224. 16.5
6 A Y 10 66.2 416. 20.9valueの列がそれぞれの平均値を示している。
グラフの作成
処理したデータをもとに、グラフを描いていく。
まず、平均値を示す棒グラフを作る。position = position_dodgeを用いることで、処理ごとにグループ化された棒が描かれる。dodgeの()内は棒の間隔を表す。0.5で間隔なし、1ですべての棒が等間隔になるようである。position = "dodge"と書いた場合も間隔が無くなる。
なお、position = position_dodge部分がないと、積み上げ棒グラフになる。
plot1 <- ggplot(data.summary, aes(x = treatment, y = value, fill = group)) + geom_col(position = position_dodge(0.7), width = 0.5) + theme_classic()
plot1
次に、エラーバー(ここでは標準誤差を表す)を追加する。position = position_dodgeの()内はさきほどの棒と同じ値にすると、棒の中央にエラーバーが表示される。
plot2 <- plot1 + geom_errorbar(aes(ymin = value - se, ymax = value + se), data = data.summary, position = position_dodge(0.7), width = 0.2)
plot2
さらに、生の実測値をプロットする。一般的にこのプロットは必須ではないが、近年の論文では実測値を加えることが多い。詳細なデータがわかるので信頼性が上がるからだろう。
dataで参照しているのがdata.summaryではなく大元のデータである点に注意。
plot3 <- plot2 + geom_point(data = data, aes(x = treatment, y = value, color = group), position = position_jitterdodge(jitter.width = 0.2), shape = 21, fill = "white", size = 3, alpha = 0.5)
plot3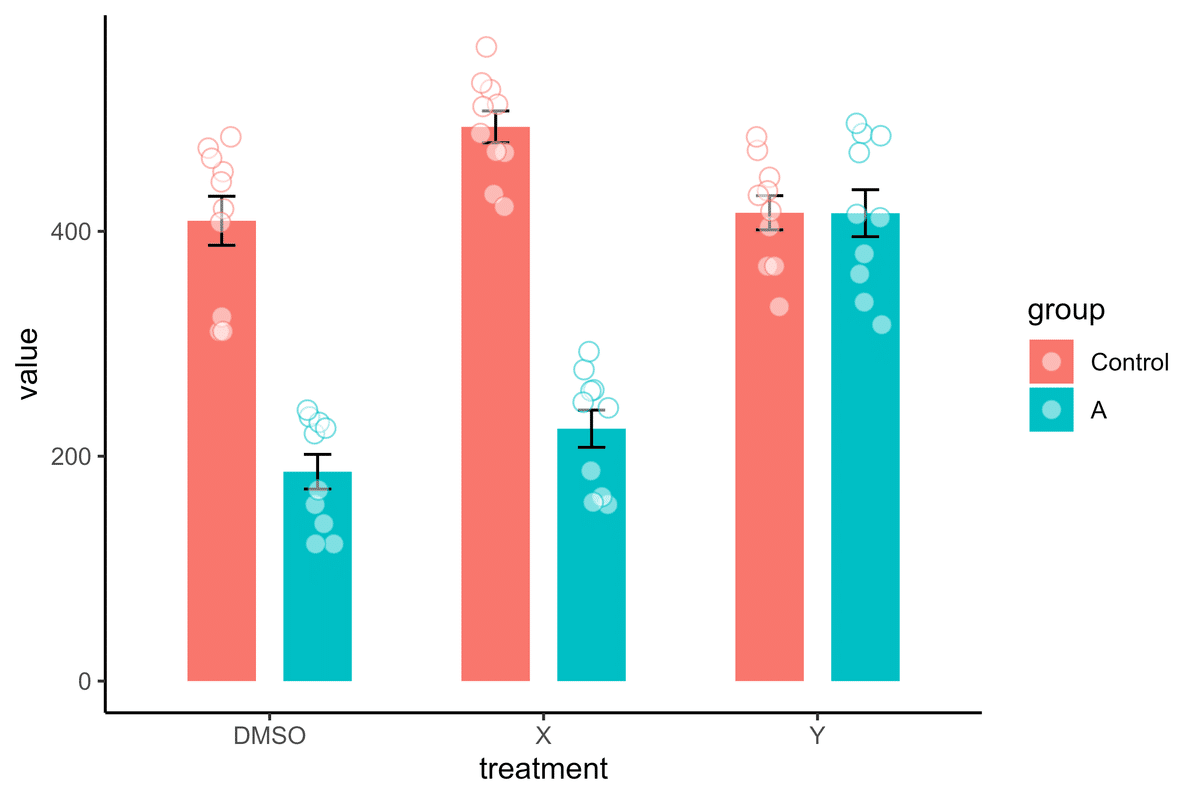
geom_pointはふつう散布図を描く際に用いる関数だが、このように棒グラフなどにも利用できる。なお、geom_jitterを用いても同様の結果を得ることができる。いずれにしても、position = position_jitterdodgeを用いることが重要となる。
position_jitterdodge内でdodge.widthやjitter.height(一般的には0)の値を指定できるが、上記のように指定しなくとも適切な位置にプロットされるようだ。
ここでは点に輪郭があるもの(shape=21)を指定し、透明度を50%にしている(alpha=0.5)。こうしておけば、点が重なったときにも見分けやすい。
ー2024/02/14 追記ー
geom_quasirandom関数でも同様にプロットできた。この場合はposition_jitterdodgeではなくdodge.widthを用いればよいようだ。
library(ggbeeswarm)
plot3qr <- plot2 + geom_quasirandom(data = data, aes(x = treatment, y = value, color = group), dodge.width = 0.7, width = 0.1, shape = 21, fill = "white", size = 3, alpha = 0.5)
plot3qr
こちらのほうがプロットどうしが重なりにくいので見やすいだろう。
ー追記ここまでー
ここまでで基本的なデータの可視化は完了した。以降は主に見た目の調整である。
棒グラフを好みの色に着色し、実測値を表す点の輪郭を黒にする。
plot4 <- plot3 + scale_fill_manual(values = c("darkgrey", "aquamarine2")) + scale_color_manual(values = c("black", "black"))
plot4
凡例のタイトルを削除し、凡例の文字サイズを調整する。タイトル削除の際は、 fillとcolorの両方を指定する必要がある。そうしないと、一方の表示が残ったままになる。
plot5 <- plot4 + guides(fill = guide_legend(title = NULL), color = guide_legend(title = NULL)) + theme(legend.text = element_text(size = 14))
plot5
x軸とy軸のラベルを調整する。今回はタイトルは付けないことにした。
plot6 <- plot5 + xlab("") + ylab("Number of cells") + theme(axis.text.x = element_text(size = 12, color = "black"), axis.text.y = element_text(size = 12, color = "black"))
plot6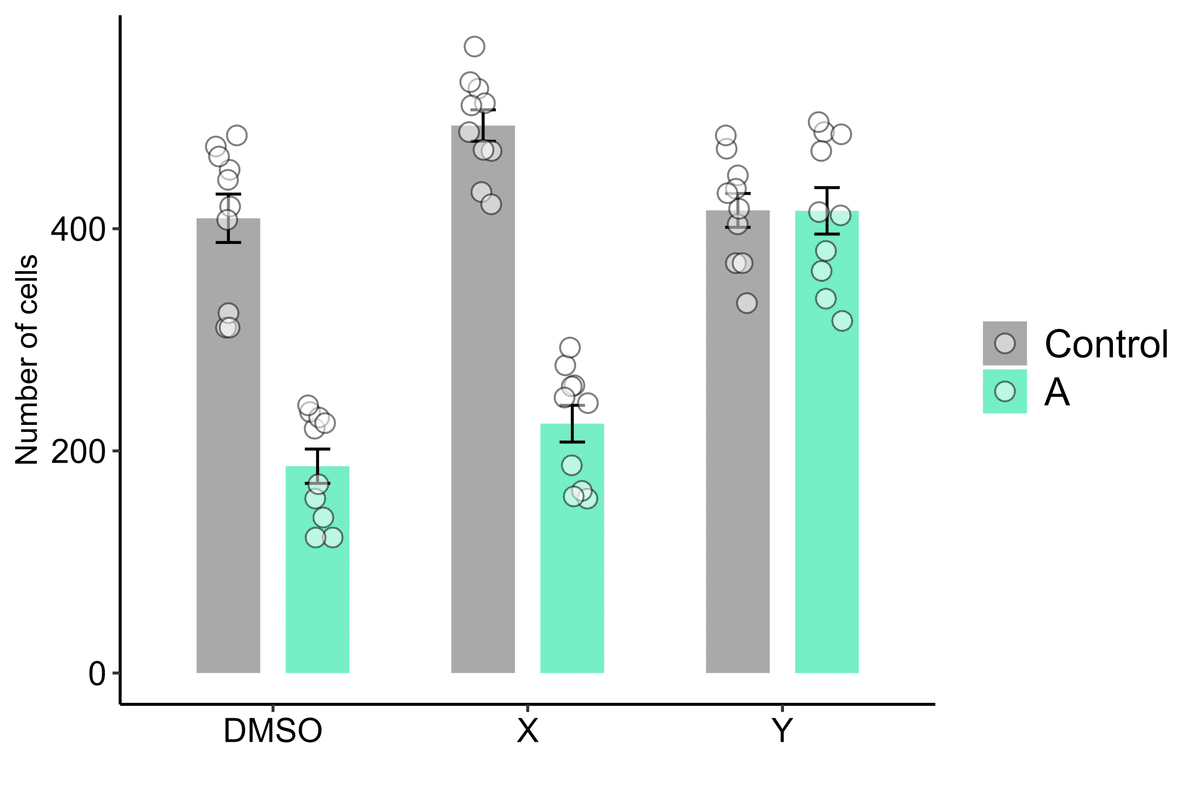
x軸と棒グラフを密着させ、y方向を700まで拡張する(後に有意差を表すアスタリスクを挿入するため)。
plot7 <- plot6 + scale_y_continuous(expand = c(0,0), limits = c(0,700))
plot7
有意差を表すアスタリスクと2群をつなぐ直線を追加する。ここでは統計的仮説検定の説明は省略するが、処理ごとにt検定を行ってControlとAの間で平均値に差があることを確認したものとする。
直線の位置は棒グラフの幅に合わせて適宜調整する必要がある。今回は項目名の中央(つまり、x = 1, 2, 3)から左右に0.15ずつの幅があるとよさそうである。
#DMSOの検定結果
plot8 <- plot7 + annotate("text", x = 1, y = 510, label = "****", size = 7) + annotate("segment", x = 0.85, xend = 1.15, y = 500, yend = 500)
plot8#薬剤Xの検定結果
plot9 <- plot8 + annotate("text", x = 2, y = 610, label = "****", size = 7) + annotate("segment", x = 1.85, xend = 2.15, y = 600, yend = 600)
plot9#薬剤Yの検定結果
plot10 <- plot9 + annotate("text", x = 3, y = 540, label = "N.S.", size = 4) + annotate("segment", x = 2.85, xend = 3.15, y = 520, yend = 520)
plot10
最後に、グラフを画像ファイルで保存する。右側に凡例があるので、縦横比の設定は横長にするとよいだろう。
ggsave(filename = "figure.png",
plot = plot10,
device = "png",
scale = 1,
width = 6, height = 4,
units = c("in"),
dpi = 900)以上で、目的としていたグラフを作成できた。このデザインであれば、有名なジャーナルに論文を投稿しても恥ずかしくないだろう。
本記事がグラフ作成に困っている読者の一助となれば幸いである。
参考にした記事
本記事と概ね同じ内容であるが、この方はstat_summaryを主に用いているのが特徴(私は全く使っていない)。
position_jitterdodgeについて少し参考にさせていただいた。
基本的なコードについては『Rグラフィックスクックブック』を参考にした。
この記事が気に入ったらサポートをしてみませんか?
