クラスClostridia に属するヒト常在腸内細菌を迅速に同定するためのMALDI-TOF MS ライブラリ
57 総閲覧数
記事のaltmetricスコアは3です
ORIGINAL RESEARCH(オリジナル研究)論文
Front. Microbiol.、2023年2月21日
第2部 脊椎動物の消化器官における微生物
第14巻 - 2023年|https://doi.org/10.3389/fmicb.2023.1104707
この記事は、研究トピックの一部です
微生物検出の最新展望。実験室から現場センサーへ




すべての記事を見る
クラスClostridia に属するヒト常在腸内細菌を迅速に同定するためのMALDI-TOF MS ライブラリ
https://www.frontiersin.org/articles/10.3389/fmicb.2023.1104707/full
Paul Tetteh Asare1,2,3,4†‡ Chi-Hsien Lee1,2,3†‡ Vera Hürlimann2,3† Youzheng Teo1, Aline Cuénod4,5†, Nermin Akduman6,7, Cordula Gekeler6,7, Afrizal Afrizal8, Myriam Corthesy9, Claire Kohout10, Vincent Thomas11, Tomas de Wouters12, Gilbert Greub9, Thomas Clavel8, Eric G. Pamer10, Adrian Egli4,5†, Lisa Maier6,7 and Pascale Vonaesch1,2,3*.
1ローザンヌ大学基礎微生物学教室(スイス、ローザンヌ
2スイス熱帯公衆衛生研究所疫学・公衆衛生部、スイス、バーゼル
3バーゼル大学(スイス、バーゼル
4バーゼル大学生物医学部応用微生物学研究室(スイス・バーゼル市
5バーゼル大学病院臨床細菌学・真菌学部門、スイス、バーゼル
6チュービンゲン大学微生物学・感染症医学研究所、ドイツ、チュービンゲン
7クラスター オブ エクセレンス「Controlling Microbes to Fight Infections」、テュービンゲン大学(ドイツ、テュービンゲン市
8ドイツ・アーヘン、RWTH大学病院、医療微生物学研究所、機能的マイクロバイオーム研究グループ
9ローザンヌ大学微生物学研究所、大学病院センター(CHUV)、ローザンヌ、スイス
10デュコソイ・ファミリー研究所、シカゴ大学感染症・グローバルヘルス学部、米国イリノイ州シカゴ市
11バイオアスター社(フランス、パリ
12PharmaBiome AG, スイス、シュリーレン
はじめに 培養から分離された微生物は、16S配列または全ゲノム配列決定によって同定されるが、これには多大なコストと時間、専門知識が必要である。Matrix-assisted Laser Desorption Ionization-time of flight mass spectrometry (MALDI-TOF MS) によるタンパク質フィンガープリンティングは、ルーチン診断における迅速な細菌同定に広く用いられていますが、常在菌については、現在のところデータベースエントリーに制限があり、性能と分解能が低いことが分かっています。本研究の目的は、非病原性ヒト常在菌の迅速な同定を可能にするMALDI-TOF MSプラグインデータベース(CLOSTRI-TOF)を開発することであった。
方法 Clostridia属の47種21属142菌株のマススペクトルプロファイル(MSP)を収録したデータベースを構築した。各菌株のMSPは、microflex Biotyper system (Bruker-Daltonics) で測定した、2つの独立した培養液から得られた20以上の生スペクトルを用いて構築された。
結果 検証のため、配列が確認された58株を使用し、CLOSTRI-TOFデータベースは2つの独立した研究室で、それぞれ98%と93%の株を同定することに成功した。次に、健康なスイス人ボランティアの便から分離された326株に対してこのデータベースを適用したところ、264株(82%)を同定し(Bruker-Daltonicsライブラリのみでは170株(52.1%))、これまで不明だった分離株の60%を分類することに成功しました。
考察 ヒトの腸内細菌叢からクロストリジウム属細菌を迅速かつ正確に同定するための新しいオープンソースのMSPデータベースについて説明する。CLOSTRI-TOFは、MALDI-TOF MSで迅速に同定できる菌種を拡大するものである。
はじめに
クロストリジウムは、グラム陽性で、しばしば厳密に嫌気性の、棒状の、芽胞を形成する細菌の重要なクラスであり、バチロタ門(旧ファームキュート)に含まれるものです。ボツリヌス菌、テタニ菌、ディフィシル菌など、一部のクロストリジウム属細菌はヒトや動物に病原性を示す。しかし、このクラスのほとんどのメンバーは、ヒトの腸管において重要な役割を果たし、特に短鎖脂肪酸である酪酸の生産に貢献している(Beyer-Sehlmeyerら、2003年;Paparoら、2017年;Cruz-Moralesら、2019年)。
腸内細菌叢からの酪酸産生クロストリジウムの枯渇は、すなわち、小児発育不全(Vonaeschら、2018)、炎症性腸疾患(Eeckhautら、2013;Geversら、2014)、大腸がん(Wuら、2013)および心臓代謝疾患などの複数の腸内および腸外疾患(Karlssonら、2012;Wangら、2012;Le Chatelierら、2013)に関連付けられてきた。さらに、酪酸生産者の枯渇は、腸内病原体に対するコロニー形成耐性を低下させることが示されました(Rivera-Chávezら、2016年)。全体として、このように、病気と酪酸生産者の不在の間には明確な関連性があります。健康と疾患における重要な役割のために、これらの細菌を分離し、特徴付けし、最終的にはいわゆる次世代プロバイオティクス(NGP)として使用する努力が重ねられてきた;健康なヒトから直接分離した細菌を、健康促進株として疾患個体に再導入する(オトゥールら、2017;チャンら、2019;リンら、2019)。新規NGPの同定と特徴付けは、培養コレクションに大きく依存しており、したがって、常在菌を迅速に同定できるツールに依存している。
細菌分離株を同定するための現在のゴールドスタンダードは、完全長16S rRNA遺伝子または全ゲノム配列決定(WGS)である(Browne et al.、2016;Lagkouvardos et al.、2019)。WGSは時間とコストがかかり、特定の専門知識が必要です。このため、臨床ルーチン診断における種の同定に使用されることはほとんどありません(Rossen et al.、2018)。そのため、ヒトの腸管から分離された細菌を同定するための、迅速、安価、正確、かつ使いやすい方法への関心が高まっています。
過去10年間で、マトリックス支援レーザー脱離イオン化飛行時間型質量分析法(MALDI-TOF MS)は、迅速かつ信頼性の高い細菌同定のための技術として確立され、主にルーチン診断で使用されています(Bizzini and Greub, 2010; Li et al.) この技術では、細菌のリボソームタンパク質も存在する2000~20000ダルトンの範囲内のマススペクトルプロファイル(MSP)を測定します。通常、MSPは商業データベース内であらかじめ記録された参照スペクトルと比較され、マーカー質量が一致することで種の同定が可能となる(Croxatto et al., 2012; Schumann and Maier, 2014; Seuylemezian et al.、2018)。スペクトルの品質(Cuénod et al., 2021, 2022)と使用するデータベースは、信頼できる同定のための2つの重要な要因である。ルーチン診断で広く利用されている現在入手可能な市販のデータベース[MBT Compass参照ライブラリ(バージョン4.1.100)を使用]は、非病原性クロストリジウムの少数派しかカバーしていない。そのため、適切な菌種同定のためのMALDI-TOF MSの使用は、現状では限られています。このハードルは、過去に他のドメインの研究者が、研究分野に関連する細菌株の社内カスタム基準データベースを作成することで克服されました(Calderaro et al.)
MALDI-TOF MSの堅牢な分類、迅速な取得時間、およびサンプルあたりの低コストを考慮し、我々はこの重要なギャップを埋め、この技術をClostridiaクラスの消化管微生物叢の常在メンバーの同定に拡張することを目的としました。多様な地域から分離された全長16S rRNA遺伝子配列が確認された細菌を用い、21属47種について142種類の質量分析プロファイル(MSP)を作成した。新たに作成したMSPライブラリは、同じ属・種を含むブラインドコードされた完全長16S rRNA配列が確認された58株のClostridiaの分離株を新しいデータベースプラグインで再同定することで精度を評価した。最後に、健康なスイス人ドナーの便から分離された326株に対して新しいMSPライブラリを適用し、ヒトの糞便サンプルから新しい分離株を同定するための可能性を示した。
材料と方法
CLOSTRI-TOFデータベースに登録されている細菌株
使用する質量スペクトルプロファイル(MSP)ライブラリの数と構成が、microflex Biotyperの分類法の割り当て精度に大きな影響を与える。非病原性クロストリジウムのメンバーの同定までカバレッジを広げるために、142の配列決定済み菌株を使用しました。これらの菌株は、Clostridiaクラスのヒト腸内常在菌の主な代表であり(表1;補足表1)、地理的に多様な菌株をできるだけ多く含み、それぞれの種をできるだけ広くカバーするように選択されたものである。21属47種に属する142株は、16S rRNA遺伝子全長配列決定により種同定を確認した。また、新たに作成したMALDI-TOF MSデータベースの検証には、58株の16S rRNA遺伝子全長同定株を使用した。菌株の情報を補足表1(ライブラリ構築に使用した菌株)および補足表2(検証用菌株)にまとめた。
表1
www.frontiersin.org
表1. CLOSTRI-TOFデータベースの構築に使用した細菌株数。
細菌培養
各菌株に使用した培地は補足表1に示すとおりで、以下のものを使用した。Brain Heart Infusion with Inulin (BHII Agar): チコリ由来のイヌリン(Sigma, cat.number I2255, Darmstadt, Germany)を添加したBHI寒天(BD Difco, cat.number 279830, Franklin Lakes, NJ, USA)、1g/l.。Schaedler Agar(Oxoid、猫番号CM0497、アイルランド)、および改変岐阜嫌気性ブロス(mGAM)(HiMedia、猫番号M2079)。すべての培地は製造元の指示に従い調製し、必要に応じて15 g agar/l を添加した。細菌は、10%CO2、5%H2、および85%N2の混合ガスを含む嫌気性ワークステーション(Anaerobic chamber; Coy Laboratories, Ann Arbor, MI, USA)を用いて厳密に嫌気性条件下で培養された。細菌は、MALDI-TOF MS分析の前に37℃で3-4日間培養した。培養条件は、補足表1(ライブラリ構築に使用した株)および補足表2(検証用株)にまとめた。
DNA精製と16S rRNA遺伝子配列の決定
ビーズビート装置(SpeedMill BeadBeater、30秒またはボルテックスアダプターで最大速度、10分)を用いて細菌培養物からゲノムDNAを抽出し、その後、WizardゲノムDNA精製キット(Promega、カタログ番号A2920、Dübendorf、スイス)を用いて製造元の指示に従って精製した。NanoDrop® ND-1000 Spectrophotometer (Witec AG, Littau, Switzerland) を用いて260 nmの吸光度を測定し、Total DNAを定量した。DNAサンプルは、さらなる分析まで20℃で保存した。
分離株の同一性を確認するために,FIREPol® DNA polymerase (Solis BioDyne, Tartu, Estonia) を用いて,プライマー27F (5′-AGA GTT TGA TCC TGG CTC AG-3′) と1492R (5′-GGT TAC CTT GTT ACG ACT T-3′) で16S rRNA遺伝子完全領域を以下の条件でPCRにより増幅させた.95℃、3分間;95℃、30秒間、50℃、30秒間、72℃、30秒間の35サイクル;そして72℃、7分間(Frankら、2008年)。PCR産物は、標準的なゲル電気泳動(1×Tris-acetate-EDTA [TAE] バッファー中1%アガロース)を用いて100Vで30分間可視化し(Bio-Rad, Cressier, Switzerland)、製造者のプロトコルに従ってPromega Wizard™ SV Gel, and PCR Cleanup Systemを用いて精製し、Sanger sequencing at Microsynth (Balgach, Switzerland) でほぼ完全長の16S rRNA遺伝子配列を取得すべく双方向に配列決定された。最も近いホモログを同定するために、得られた配列をBasic Local Alignment Search Tool (BLAST) (Altschul et al., 1990) を用いてアライメントした。配列の相同性99%以上を種一致、配列の類似性97%以上を属一致と定義した(Becker et al., 2004)。系統樹を構築するために、MUSCLEを用いて16S rRNA遺伝子の配列を整列・トリミングし、MEGA 11 (Tamura et al., 2021) を用いて1000ブートストラップレプリカで最尤系統樹を構築した。
MSP作成のためのMALDI-TOF MSスペクトルの調製
ブルカー・ダルトニクスが新規ライブラリー作成に推奨するように、エタノール-ギ酸抽出でMSP作成用のサンプルを調製した。ギ酸は、マトリックスの酸性pHがリボソームタンパク質の抽出を向上させるため、細菌学でMALDI-TOFを使用する際に日常的に使用されています(Croxatto et al.、2012)。簡単に説明すると、2~3個のコロニーを300μlの高圧液体クロマトグラフィー(HPLC)グレードの水(Sigma-Aldrich, Buchs, Switzerland)に懸濁し、900μlの100%エタノール(Sigma-Aldrich, Buchs, Switzerland)と混合した。15,000×g で 2 分間遠心分離した後、上清を除去し、ペレットを乾燥させた。サンプル抽出のため,50μlのギ酸(水中70%)を菌体ペレットに加え,チューブをボルテックスで十分に混合し,50μlのアセトニトリル(シグマ)を添加した.この混合物を15,000×gで2分間遠心分離した。各菌株について、菌体抽出物を含む上清1μlを96スポット鋼板(ブルカー・ダルトニクス社製)に15反復でスポットし、室温で乾燥させた。各プレートの2つのMALDIターゲットスポットに1マイクロリットルの細菌試験標準(BTS、ブルカー・ダルトニクス)をピペッティングし、取得および処理中の較正を可能にした。その後、測定前に1μlのα-シアノ-4-ヒドロキシ桂皮酸(HCCA)マトリックスでサンプルを重ね、再度風乾させた。Bacterial Test Standard (BTS) は、各取得セッションの前に、装置の較正に使用されました。
MALDI-TOF MSのデータ取得
MALDI-TOF MSは、Bruker-Daltonics microflex LT/LHベンチトップ型質量分析計で実施されました。サンプルのタンパク質質量スペクトルは、flexControl 3.4プログラム(Bruker-Daltonics)およびレーザー周波数20Hz、線形ポジティブモードを使用して、質量範囲2000〜20000Daで取得された。デフォルトの動作条件は、イオン源1、20kV、イオン源2、18.25kV、パルスイオン抽出、370nsであった。各スペクトルは、240レーザーショット(40レーザーショット/ポジション×6種類のポジション)まで合計された。各菌株について、2つの独立した培養から30個のシングルスペクトルを作成し、15個の技術的複製を作成した。すべての生スペクトルはZenodoに寄託されており、アクセス番号は10.5281/zenodo.7573939である。
MALDI-TOF MS品質管理およびカスタムMSPライブラリー作成
生スペクトルの品質はflexAnalysis 3.4 software (Bruker-Daltonics) を用いて評価しました。生スペクトルの品質チェックを容易にするために、平滑化パラメータとベースライン減算パラメータを適用した。ベースライン減算には、マルチポリゴン、最小、ペナルティ付き最小二乗、ペナルティ付きBスプラインベースライン減算法を用い、平滑化には、Savitzky-Golay平滑化法を使用した。各分析日において、細菌試験標準(BTS)スペクトルの8つの既知の質量は、質量精度を証明するために+/- 300 ppmの許容範囲内になるように制御された。フラットライン、外れ値、劇的な質量シフト、および異常値を示すスペクトルは削除されました。フラットラインスペクトルは完全にフラットなラインを持つスペクトルと定義され、アウトライナーは同じ株の他のスペクトルでは検出されないユニークなピークを持つスペクトルと定義された。マスシフトは 6,000 Da から 7,000 Da の間のランダムなピークで評価し、500 ppm 以下のピークシフトを持つスペクトルのみを受理した。生スペクトルを処理した後、1株あたり最低27の高品質スペクトルを選択し、MALDI Biotyper Compass Explorerソフトウェア(ブルカー・ダルトニクス社製)を使用してマージし、質量スペクトルプロファイル(MSP)を作成した。MSPを構成する菌株ごとのマージされたスペクトルは、log scoreが2.7より大きく、ピーク頻度が75%より大きいことが確認された。最終的には、142株のMSPをもとにライブラリーを作成した(図1A)。MALDI Biotyper Compass Explorer 4.1を用いて、ユークリッド距離測定と平均連結アルゴリズムによりMSPデンドログラムを構築した。最終的なライブラリはZenodoに寄託されており、アクセス番号は10.5281/zenodo.7573939です。
図1
www.frontiersin.org
図1. CLOSTRI-TOFの作成(A)、検証(B)、MALDI-TOF MSによる微生物同定ワークフローの主要ステップ(C)。
CLOSTRI-TOFデータベースの検証
CLOSTRI-TOFデータベースの検証のため、2つの共同研究室で16S rRNA遺伝子配列が決定された58の完全長分離株のブラインドテストを実施しました(図1B;補足表2)。検証は、eDT(extended direct transfer)法を用いて行った。簡単に言うと、数個の細菌コロニーをMALDI-TOF MSターゲットに直接塗抹し、ルーチンの細菌同定に一般的に推奨されているように、1μlの飽和HCCAマトリックス溶液で覆った。スペクトルが検出されない場合は、エタノール-ギ酸抽出法(既述)を用いて分析を繰り返した。生成された生スペクトルを2つの結合データベース (Bruker-Daltonics MALDI Biotyper Compass MSP library v. 4.1.100 および CLOSTRI-TOF) と比較した (図1B)。同定結果は、Bruker が提案したスコア値で分類されました。スペクトルの最も高いマッチングスコアは、高い信頼性の同定 (2.000-3.000) 、低い信頼性の同定 (1.700-1.999) 、生物の同定が不可能 (0.000-1.699) を表す範囲で表現されています。
健康なスイス人ボランティアから分離された326の臨床分離株セットの同定
これまで知られていなかった分離株のセットでライブラリをテストするために、オリジナルの Bruker-Daltonics MALDI Biotyper Compass MSP library v. 4.1.100 と組み合わせた CLOSTRI-TOF データベースを使って、EnrichBut プロジェクト (Swiss Ethics Number: BASEC ID 2021-00199) で募集したスイス人健康者の便サンプルから分離した合計326の嫌気性培養物を特定しました(図1C)。簡単に説明すると、サンプル採取の2週間前に抗生物質を摂取していない30歳から60歳の自己申告の健康なスイス人成人4名から便サンプルを採取した。各便提供者には、嫌気条件下で酸素感受性細菌群を保存できる特注の便サンプリングキットが提供された。便サンプルは、処理前に室温で嫌気的に保存された。受領後(採取後48時間以内)、便サンプルを嫌気性リン酸緩衝生理食塩水(PBS)に再懸濁(1:10 w/v)させた。この懸濁液をPBSで連続的に希釈し、100μLをColumbia Blood、BHII、Schaedler、mGAM寒天培地にプレーティングした。プレートは37℃で5日間インキュベートされた。各培地から異なるコロニー形態を示す20〜30個のコロニーを摘出し、分離株の純度を確認するために新鮮な寒天プレートに3回再圧入した。分離株は液体ブロスで培養し、20%グリセロールと混合して-80℃で保存した。
分離株は,eDT(extended direct transfer)法を用いてMALDI-TOF MS同定用に調製し,3回の技術的複製に基づいて同定した。得られたスペクトルは、市販のBruker-Daltonicsライブラリ、またはBruker-DaltonicsとCLOSTRI-TOFの結合ライブラリのいずれかと比較された。
結果
MSPライブラリーの作成
解析した142株について、2,130の標的位置と合計4,260の個別のマススペクトルを作成した。また、MSP生成前に1株あたり平均1-3個の生スペクトルを除去しており、スペクトルの質は高かった。このように、47 種の生物種に対してロバストなライブラリーを作成することができた。
MSPライブラリーの性能評価
新たに構築したライブラリーを検証するため、ライブラリー構築に用いた株群には含まれないが、16S rRNA 遺伝子の配列に基づいて事前に同定した58株(検証株、補表 S2)を追加して MALDI-TOF MS に供した。Bruker データベースと CLOSTRI-TOF データベースプラグインを組み合わせることにより、A 研究所では 57/58 株(98.3%)、B 研究所では 54/58 株(93.1%)を確実に種レベルで同定できた(図 2)。なお,Blautia 属に属する複数の菌株は,高スコア(≧2.0)で同定されたが,誤って種レベルで同定された。また,Coprococcus eutactusは両研究室で正しく同定されたが,スコアは1.7~2.0であった.このように,Bruker データベースと CLOSTRI-TOF データベースを組み合わせることで,検証株のほとんどを種レベルで,全株を属レベルで再同定することができた.
図 2
www.frontiersin.org
図2. ラボA(A)とラボB(B)で行った、新たに作成したCLOSTRI-TOF MSを用いたクロストリジウム属菌のブラインドテスト。スペクトルの最高一致スコアは、高信頼性同定(2.000-3.000)、低信頼性同定(1.700-1.999)、生物同定不可(0.000-1.699)を示す範囲で表された。
16S rRNA遺伝子に基づく系統樹とMALDI-TOF MSプロファイリング
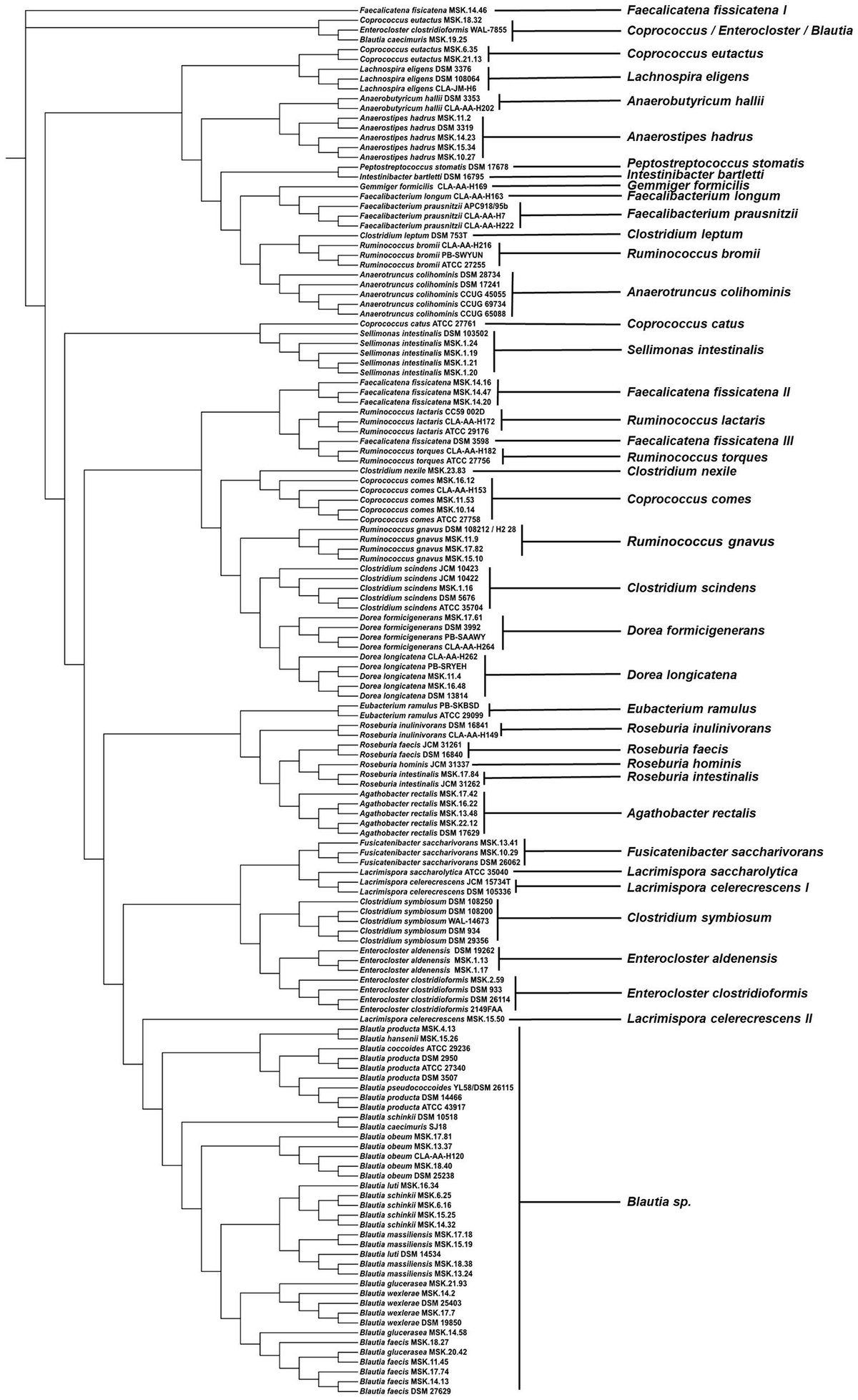
microflex Biotyperと16S rRNA遺伝子ベースのシーケンスの分類学的割り当て関係を調べるために、これらの手法のどちらかで作成したデンドログラムを比較しました。ほとんどの場合、両手法は同じ種に属する株がクラスター化する同一の結果を示した(図3、4)。しかし、Agathobacter rectalis、Blautia spp.、Faecalicatena fissicatena、Lacrimispora celerecrescensの異なる株はMALDI-TOFベースの2~3個の異なるクラスタを形成した(Figure 3)。同様に,16S rRNA遺伝子配列に基づく最尤系統樹では,Agathobacter rectalis,Blautia spp.,Faecalicatena fissicatena,Lacrimispora celerecrescensの同一種の菌株をクラスタリングできなかった(図4).
図3
www.frontiersin.org
図3. MSPデンドログラムクラスター解析による、スペクトルプロファイルが類似している異なる種の分布。
図4
www.frontiersin.org
図4. CLOSTRI-TOFデータベースの構築に使用した菌株の16S rRNA遺伝子完全配列に基づく最尤法系統樹。
CLOSTRI-TOFで強化したMALDI-TOF MSによる嫌気性腸内細菌分離株の同定
健康なスイス人ボランティアの糞便から分離された326株(補足表5)について、microflex Biotyperを用いた解析を行った。326 株のうち,170 株(52.1%)がオリジナルの Bruker データベースで同定され,156 株(47.9%)がスコア > 1.7 でライブラリで同定できない/一致しないものであった.オリジナルライブラリと CLOSTRI-TOF ライブラリプラグインを組み合わせることで,スコア > 2.0 で同定された株は 264 株(80.9%)に増加し(図 5),60%の未知株が同定されたことになる.
図 5
www.frontiersin.org
図5. Bruker-Daltonics MALDI Biotyper Compass MSP library v. 4.1.100 と CLOSTRI-TOF データベースプラグインを併用した場合と併用しない場合の細菌 326 株の同定率。
考察
今日まで、16S rRNA 遺伝子配列の決定は、ヒトの消化管から分離された未知の微生物を同定するために最も広く使用されている技術の 1 つです。ここでは、MALDI-TOF MSのライブラリプラグインであるCLOSTRI-TOFを提案し、迅速、正確、かつ経済的な代替法としてMALDIバイオタイピングの能力を強化します。
MALDIバイオタイピングはスペクトルデータベースに強く依存しているため、ある種のスペクトル情報を既に持っている分類群のみを同定することができます。このハードルは、特定の微生物グループのための社内カスタムリファレンスデータベースを作成することで克服されています。例えば、これまでの研究では、Borrelia属 (Calderaro et al., 2014), Burkholderia属 (Fergusson et al., 2020) またはVibrio属 (Moussa et al., 2021), Clostridium tyrobutyricumの異なる株 (Burtscher et al., 2020) および蠕虫 (Sy et al., 2022) のためのデータベースプラグインが生成されています。
今回発表したCLOSTRI-TOFライブラリプラグインでは、ライブラリプラグインに含まれる47の細菌種をすべて属レベルで、大部分は種レベルまで同定することができた。さらに、オリジナルのBrukerライブラリーとCLOSTRI-TOFライブラリーのプラグインを組み合わせることで、健康なスイス人ボランティアの糞便細菌分離コレクションから同定された菌株の数が大幅に増加することを示すことができた。また,ライブラリに含まれる1種(Coprococcus eutactus)は,検証用菌株を使用しても信頼性の高い同定を得ることができませんでした.また,C. eutactus は CLOSTRI-TOF ライブラリでは 2 株のみであった.種内多様性を考慮し、信頼性の高い同定を可能にするためには、データベースに含まれる菌株の数が重要であることが、以前に示されている(Erler et al.、2015)。そのため、今後のCLOSTRI-TOFデータベースの更新では、菌株カバー率が低い種については、含まれる菌株数を拡大する必要がある。
我々のデータベースプラグインは、Blautiaの属レベルでの同定は可能であるが、種レベルでの信頼性の高い割り当てを可能にするものではなかった。Blautia属の菌株は16S rRNA遺伝子の系統樹でも多系統であったため、この属内の系統関係をより詳細に比較することができれば興味深い。ここで紹介するライブラリプラグインは、ヒトの消化管に生息するClostridiaクラスの主要なメンバーに焦点を当てています。重要な代謝物である酪酸の生産に頻繁に関与し(Louis and Flint, 2009)、栄養不足(Vonaesch et al., 2018)、潰瘍性大腸炎(Machiels et al., 2014)、2型糖尿病(Wang et al., 2012)などのディスバイオティクス疾患で減少するため、このグループの微生物を選びました。データベースをClostridiaのクラスを超えて、ヒトマイクロバイオームの他の頻繁なメンバーに拡張することは、ヒトの糞便サンプルから分離されたすべての細菌株を迅速に同定するための鍵となるであろう。
注目すべきは、MALDI バイオタイピングは細菌の同定を迅速に行うことができますが、全ゲノム配列決定で得られるような各単離株の情報量を得ることはできない点です。したがって、MALDI による同定は、全ゲノム配列決定のようなより高度な方法を用いてより詳細な特徴を得る株を選択するための、低コストで迅速な事前同定ツールとして捉える必要があります。
結論
我々のデータは、MALDI-TOF MSが常在菌の分離株を迅速に同定するための有望なツールであることを示している。今回開発したオープンソースのライブラリプラグインは、属まで含めたすべての分類群、および種レベルまで含めたほとんどの分類群を識別することが可能です。
データの利用可能性に関する声明
本研究で紹介したデータセットは、Zenodoの以下のリンクから入手可能である。https://zenodo.org/deposit/7573939#。
著者による貢献
PA、LM、PV:コンセプト立案。VT、TW、GG、TC、EP、AE:リソース。PA、CL、VH、YT、AC、NA、CG、AA、MC、CK、および PV:方法論。PA、CL、PV:データキュレーション、形式分析、可視化、原著論文の執筆。PA, CL, GG, TC, EP, AE, LM, and PV: 査読と編集。全著者が論文に貢献し、提出されたバージョンを承認した。
資金提供
この研究は、Bill and Melinda Gates Foundation(PVとLMへの助成番号INV-004352)、Basel大学Forschungsfonds(PV)、スイス国立科学財団のEccellenza Fellowship(助成番号PCEFP3_194545)からPVに資金提供されたものである。VonaeschとGreubの研究室は、スイス国立科学財団(助成番号180575)の助成を受けた国立研究能力センターであるNCCR Microbiomesに所属している。TCはドイツ研究財団(DFG)、プロジェクトID 403224013-SFB 1382、プロジェクトID 395357507-SFB 1371、プロジェクトID 460129525-NFDI4Microbiotaから資金援助を受けている。NA, CG, LMはDFG (CMFI Cluster of Excellence EXC 2124 and Emmy Noether Program)の支援を受けています。
謝辞
VonaeschおよびMaier研究室のメンバー、Sören Becker、Johanna Burtscher、Jennifer Keiserには、有益な議論とフィードバックをいただいた。さらに、Bruker MALDI-TOF MSの操作にご協力いただいたUniversity Hospital BaselとCentre Hospitalo-Universitaire Vaudois (CHUV) のClinical Bacteriology and Mycologyのラボ技術者に感謝します。図1はBioRenderで作成した。
利益相反について
著者らは、本研究が利益相反の可能性があると解釈される商業的または金銭的関係がない状態で実施されたことを宣言する。
出版社からのコメント
本論文で述べられたすべての主張は、著者個人のものであり、必ずしも所属団体、出版社、編集者、査読者の主張を代表するものではありません。本論文で評価される可能性のある製品,あるいはそのメーカーが行う可能性のある主張は,出版社によって保証または承認されたものではない.
補足資料
本論文の補足資料は、https://www.frontiersin.org/articles/10.3389/fmicb.2023.1104707/full#supplementary-material に掲載されています。
参考文献
Altschul, S. F., Gish, W., Miller, W., Myers, E. W., and Lipman, D. J. (1990). 基本的なローカルアライメント検索ツール。J. Mol. Biol. 215, 403-410.
クロスレフフルテキスト|Google Scholar
ベッカー、K.、ハームセン、D.、メルマン、A.、マイヤー、C.、シューマン、P.、ピーターズ、G.、その他 (2004). 16S リボソーム DNA に基づくブドウ球菌種の同定のための品質管理されたリボソーム配列データベースの開発および評価。J. Clin. Microbiol. 42, 4988-4995.
PubMed Abstract|CrossRef Full Text|Google Scholar
Beyer-Sehlmeyer, G., Glei, M., Hartmann, E., Hughes, R., Persin, C., Böhm, V., et al.(2003)。酪酸は、食物繊維源の腸内細菌叢を介した発酵中に生成されるいくつかの成長阻害物質の一つに過ぎない。酪酸は、食物繊維源の腸内フローラを介した発酵中に生成されるいくつかの成長阻害物質の一つに過ぎない。
PubMed Abstract|CrossRef Full Text|Google Scholar
Bizzini, A., and Greub, G. (2010). Matrix-assisted laser desorption ionization time-of-flight mass spectrometry, a revolution in clinical microbial identification(マトリックス支援レーザー脱離イオン化飛行時間型質量分析、臨床微生物同定における革命)。Clin. Microbiol. Infect. 16, 1614-1619. doi: 10.1111/J.1469-0691.2010.03311.X.
PubMed Abstract | CrossRef Full Text | Google Scholar
Browne, H. P., Forster, S. C., Anonye, B. O., Kumar, N., Neville, B. A., Stares, M. D., et al. (2016). 培養不可能な」ヒト微生物群の培養により、新規の分類群および広範な胞子形成が明らかになった。ネイチャー 533, 543-546.
PubMed Abstract|CrossRef Full Text|Google Scholar
Burtscher, J., Küller, F., Dreier, M., Arias-Roth, E., Drissner, D., and Domig, K. J. (2020). 3つの異なる型別技術を用いたClostridium tyrobutyricum株の特性評価。Microorganisms 8, 1-17. doi: 10.3390/microorganisms8071057
PubMed Abstract|CrossRefフルテキスト|Google Scholar
Calderaro, A., Gorrini, C., Piccolo, G., Montecchini, S., Buttrini, M., Rossi, S., et al.(2014)。社内MALDI-TOF MSデータベース作成後のボレリア種の同定。PLoS One 9:e88895. doi: 10.1371/journal.pone.0088895.
PubMed Abstract|CrossRef Full Text|Google Scholar
Chang, C. J., Lin, T. L., Tsai, Y. L., Wu, T. R., Lai, W. F., Lu, C. C., et al. (2019). 疾患改善における次世代プロバイオティクス。J. Food Drug Anal. 27, 615-622. doi: 10.1016/j.jfda.2018.12.011.
PubMed Abstract|CrossRefフルテキスト|Google Scholar
Croxatto, A., Prod'hom, G., and Greub, G. (2012). 臨床診断微生物学におけるMALDI-TOF質量分析法の応用。FEMS Microbiol. REV. 36, 380-407.
クロスレフフルテキスト|Google Scholar
Cruz-Morales, P., Orellana, C. A., Moutafis, G., Moonen, G., Rincon, G., Nielsen, L. K., et al.(2019).。クロストリジウムの進化と分類の再検討、系統学的な更新。Genome Biol. Evol. 11, 2035-2044. doi: 10.1093/gbe/evz096
PubMed Abstract|CrossRefフルテキスト|Google Scholar
Cuénod, A., Aerni, M., Bagutti, C., Bayraktar, B., Boz, E. S., Carneiro, C. B., et al.(2022)。ルーチン診断における MALDI-TOF 質量スペクトルの品質: 47 の困難な細菌株を使用した 12 カ国 36 研究所を含む国際外部品質評価による結果。Clin. Microbiol. Infect., 190-199.
PubMed Abstract|CrossRef Full Text|Google Scholar
Cuénod, A., Foucault, F., Pflüger, V., and Egli, A. (2021). 臨床ルーチン診断における種同定のMALDI-TOF質量スペクトル品質と関連する要因。Front. Cell. Infect. Microbiol. 11:646648.論文番号: 10.3389/FCIMB.2021.646648
PubMed Abstract|CrossRef Full Text|Google Scholar
Eeckhaut, V., Machiels, K., Perrier, C., Romero, C., Maes, S., Flahou, B.、他 (2013). 炎症性腸疾患におけるブチリコッカス・プルリカエコラム。Gut 62, 1745-1752. doi: 10.1136/gutjnl-2012-303611
クロスレフフルテキスト|Google Scholar
Erler, R., Wichels, A., Heinemeyer, E. A., Hauk, G., Hippelein, M., Reyes, N. T., et al. (2015). VibrioBase: a MALDI-TOF MS database for fast identification of Vibrio spp. that are potentially pathogenic in humans. Syst. Appl.Microbiol. 38, 16-25. doi: 10.1016/j.syapm.2014.10.009
PubMed Abstract|CrossRefフルテキスト|Google Scholar
Fergusson, C. H., Coloma, J. M. F., Valentine, M. C., Haeckl, F. P. J., and Linington, R. G. (2020).を参照。カスタムマトリックス支援レーザー脱離イオン化飛行時間型質量分析データベースによるBurkholderia属および近縁属の環境分離株の同定。Appl. Environ. Microbiol. 86. doi: 10.1128/AEM.00354-20
PubMed Abstract|CrossRef Full Text|Google Scholar
フランク・J・A・ライヒ・C・I・シャーマ・S・ワイズバウム・J・S・ウィルソン・B・A・オルセン・G・J・(2008年). 細菌 16S rRNA 遺伝子の増幅に一般的に使用される 2 種類のプライマーの批判的評価(Critical evaluation of two primers commonly used for amplification of bacterial 16S rRNA genes. Appl. Environ. Microbiol. 74, 2461-2470.
PubMed Abstract|CrossRefフルテキスト|Google Scholar
Gevers, D., Kugathasan, S., Denson, L. A., Vázquez-Baeza, Y., Van Treuren, W., Ren, B., et al.(2014年)。新規発症のクローン病における治療未受診のマイクロバイオーム。セルホストマイクロビー 15, 382-392.
PubMed Abstract|CrossRefフルテキスト|Google Scholar
Karlsson, F. H., Fåk, F., Nookaew, I., Tremaroli, V., Fagerberg, B., Petranovic, D., et al.(2012年)。症候性アテローム性動脈硬化症は、腸内メタゲノムの変化と関連している。Nat. Commun. 3:1245.論文番号: 10.1038/ncomms2266
PubMed Abstract|CrossRef Full Text|Google Scholar
Lagkouvardos, I., Lesker, T. R., Hitch, T. C. A., Gálvez, E. J. C., Smit, N., Neuhaus, K., et al. (2019). Muribaculaceaeの配列および培養研究により、このまだ記述されていないファミリーの新種、宿主嗜好性、および機能的可能性が明らかになった。マイクロバイオーム 7, 28-15. doi: 10.1186/s40168-019-0637-2
PubMed Abstract|CrossRef Full Text|Google Scholar
Le Chatelier, E., Nielsen, T., Qin, J., Prifti, E., Hildebrand, F., Falony, G., et al.(2013)。ヒト腸内細菌群の豊富さは、代謝マーカーと相関している。ネイチャー500, 541-546.
クロスリフトフルテキスト|Google Scholar
Li, Y., Shan, M., Zhu, Z., Mao, X., Yan, M., Chen, Y., et al. (2019). 嫌気性菌の迅速な同定へのMALDI-TOF MSの適用。BMC Infect. Dis. 19, 941-911. doi: 10.1186/s12879-019-4584-0
PubMed Abstract|CrossRef Full Text|Google Scholar
Lin, T.-L., Shu, C.-C., Lai, W.-F., Tzeng, C.-M., Lai, H.-C., and Lu, C.-C. (2019). 次世代プロバイオティクスの疾病改善への投資 - strains do matter. Med. Microecol. 1-2:100002. doi: 10.1016/j.medmic.2019.100002.
クロスレフフルテキスト|Google Scholar
Louis, P., and Flint, H. J. (2009). ヒト大腸由来の酪酸産生菌の多様性、代謝および微生物生態学。FEMS Microbiol. 294, 1-8. doi: 10.1111/j.1574-6968.2009.01514.x
PubMed Abstract|CrossRef Full Text|Google Scholar
Machiels, K., Joossens, M., Sabino, J., de Preter, V., Arijs, I., Eeckhaut, V., et al.(2014)。酪酸産生種Roseburia hominisとFaecalibacterium prausnitziiの減少は、潰瘍性大腸炎患者におけるディスバイオーシスを規定する。Gut 63, 1275-1283.
PubMed Abstract|CrossRefフルテキスト|Google Scholar
Moussa, M., Cauvin, E., Le Piouffle, A., Lucas, O., Bidault, A., Paillard, C., et al.(2021)。海産軟体動物に病原性を有するビブリオ属菌の迅速な同定のためのMALDI-TOF MSデータベース. Appl.Microbiol. Biotechnol. 105, 2527-2539. doi: 10.1007/s00253-021-11141-0
PubMed Abstract|CrossRef Full Text|Google Scholar
オトゥール,P. W., マルケージ,J. R., およびヒル,C. (2017). 次世代プロバイオティクス:プロバイオティクスからライブバイオセラピューティックまでのスペクトラム。Nat. Microbiol. 2:17057. doi: 10.1038/nmicrobiol.2017.57
PubMed Abstract|CrossRefフルテキスト|Google Scholar
Paparo, L., Calignano, A., Tocchetti, C. G., Di Scala, C., Russo, R., Bonaduce, D., et al.(2017)。The influence of fiber on gut microbiota: butyrate as molecular player involved in the beneficial interplay between dietary fiber and cardiovascular health(食物繊維と心血管系の健康との間の有益な相互作用に関与する分子プレイヤーとしての酪酸). Amsterdam: Elsevier Inc.
Google Scholar
Rivera-Chávez, F., Zhang, L. F., Faber, F., Lopez, C. A., Byndloss, M. X., Olsan, E. E., et al. (2016). 腸内細菌叢から酪酸産生クロストリジウムを枯渇させると、サルモネラの好気性ルーミナル拡大が促進される。Cell Host Microbe 19, 443-454. doi: 10.1016/j.chom.2016.03.004.
PubMed Abstract|CrossRefフルテキスト|Google Scholar
Rossen, J. W. A., Friedrich, A. W., and Moran-Gilad, J. (2018). ルーチン診断微生物学における全ゲノムシーケンス導入の実際的な問題点。Clin. Microbiol. Infect. 24, 355-360. doi: 10.1016/j.cmi.2017.11.001.
PubMed Abstract|CrossRefフルテキスト|Google Scholar
Schumann, P., and Maier, T. (2014). MALDI-TOF Mass Spectrometry Applied to Classification and Identification of Bacteria(細菌の分類と同定に適用されるMALDI-TOF質量分析). 1st Amsterdam Elsevier Ltd.
Google Scholar
Seuylemezian, A., Aronson, H. S., Tan, J., Lin, M., Schubert, W., and Vaishampayan, P. (2018).を参照。宇宙船および関連表面から収集された細菌分離株の種レベルの同定のためのカスタムMALDI-TOF MSデータベースの開発。Front. Microbiol. 9:780. doi: 10.3389/fmicb.2018.00780.
PubMed Abstract|CrossRef Full Text|Google Scholar
Sy, I., Conrad, L., and Becker, S. L. (2022). 蠕虫の同定のためのMALDI-TOF質量分析法の最近の進歩と将来の潜在的なアプリケーション。診断 12, 3035.
PubMed Abstract|CrossRef Full Text|Google Scholar
このような場合、「MEGA11」と「MEGA12」の2種類があります。MEGA11: 分子進化遺伝学解析バージョン11. Mol. Biol. 38, 3022-3027. doi: 10.1093/molbev/msab120
PubMed Abstract|CrossRefフルテキスト|Google Scholar
Vonaesch, P., Morien, E., Andrianonimiadana, L., Sanke, H., Mbecko, J. R., Huus, K. E., et al. (2018). 小児期の成長阻害は、消化管のデコンパートメント化と中咽頭分類群の過成長と関連している。Proc. Natl.Acad. 115, E8489-E8498.
PubMed Abstract|CrossRef Full Text|Google Scholar
Wang, J., Qin, J., Li, Y., Cai, Z., Li, S., Zhu, J.、他 (2012). 2型糖尿病における腸内細菌叢のメタゲノム-ワイド関連研究。ネイチャー490, 55-60.
PubMed Abstract|CrossRef Full Text|Google Scholar
Wu, N., Yang, X., Zhang, R., Li, J., Xiao, X., Hu, Y.、他 (2013). 大腸がん患者における糞便微生物叢のディスバイオーシス・シグネチャー(Dysbiosis signature of fecal microbiota in colorectal cancer patients. Microb. Ecol. 66, 462-470.
PubMed Abstract|CrossRefフルテキスト|Google Scholar
キーワード:ヒト腸内細菌叢、クロストリジウム、MALDI-TOF MS、常在菌、嫌気性、細菌同定、カルチュロミクス、次世代プロバイオティクス
引用元 Asare PT, Lee C-H, Hürlimann V, Teo Y, Cuénod A, Akduman N, Gekeler C, Afrizal A, Corthesy M, Kohout C, Thomas V, de Wouters T, Greub G, Clavel T, Pamer EG, Egli A, Maier L and Vonaesch P (2023) a MALDI-TOF MS library for rapid identification of human commensal gut bacteria from a class Clostridia. Front. Microbiol. 14:1104707.doi: 10.3389/fmicb.2023.1104707
Received: 2022年11月21日; Accepted: 2023年1月31日
公開:2023年2月21日
編集者
パルミロ・ポルトロニエリ(イタリア国立研究評議会(CNR)、イタリア
査読者:Hoh Boon-Peng, International Research Council, Italy
Hoh Boon-Peng, 国際医科大学, マレーシア
Pawan Kumar Kanaujia, Mahayogi Gorakhnath University, インド
Sidharth Prasad Mishra, 南フロリダ大学, 米国
Copyright © 2023 Asare, Lee, Hürlimann, Teo, Cuénod, Akduman, Gekeler, Afrizal, Corthesy, Kohout, Thomas, de Wouters, Greub, Clavel, Pamer, Egli, Maier and Vonaesch. 本論文は、Creative Commons Attribution License (CC BY) の条件の下で配布されているオープンアクセス論文です。原著者および著作権者のクレジットを表示し、本誌の原著を引用することを条件に、他のフォーラムでの使用、配布、複製を許可する。本規定に従わない使用,配布,複製は認めない.
*通信欄 Pascale Vonaesch, ✉ pascale.vonaesch@unil.ch
†Present addresses: Paul Tetteh Asare, Gnubiotics Sciences AG, SuperLab Suisse, Lausanne, Switzerland.
Chi-Hsien Lee, Louvain Drug Research Institute, Université Catholique de Louvain, Brussels, Belgium
Vera Hürlimann ファーマバイオームAG、スイス・シュリーレン
Adrian Egli、Aline Cuénodチューリッヒ大学医学微生物学研究所(スイス、チューリッヒ
4.本作品への貢献は同等です。
免責事項:本論文で述べられているすべての主張は、著者個人のものであり、必ずしも所属団体、出版社、編集者、査読者のものを代表するものではありません。この記事で評価される可能性のある製品、またはそのメーカーが行う可能性のある主張は、出版社によって保証または承認されるものではありません。
こんな人も見ています
モレル(Morchella sextelata)の栽培ライフサイクルにおける土壌微生物相のダイナミクス
Chen Zhang, Xiaofei Shi, Jiexiong Zhang, Yesheng Zhang, Wen Wang(チェン・ジャン、シャオフェイ・シー、ジェシオン・ジャン、ウェン・ワン
全身性エリテマトーデスにおける腸内真菌の変化-パイロット研究
李宝珠、王華、李賢宝、張乾留、黄栄桂、呉洪、王義宇、李凱迪、朱秀傑、曹昂偉、周浩岳、方新玉、凌瑞学、ファン陰光、田金輝、周宗文、葉東欽
編集部 病原体ゲノミクス。感染症サーベイランスとアウトブレイク調査の強化
マーク・J・ストルーレンス、ヴィタリ・シンチェンコ
呼吸器検体の種類に応じた結核診断用微生物検査の性能:10年間のレトロスペクティブスタディ
Marc-Olivier Boldi, Justin Denis Lessard, Rina Neziri, René Brouillet, Christophe Von Garnier, Valérie Chavez, Jesica Mazza-Stalder, Katia Jaton, Gilbert Greub および Onya Opota
自閉症児と定型発達児の血漿および尿サンプルにおける代謝プロファイルの比較。予備的研究
徐新潔、蔡暁娥、孟範超、宋天佳、王小西、魏益珍、蔡福俊、ボーロン、王俊、尤新、張榮
フッター
ガイドライン
探索
アウトリーチ
接続
フォローする
© 2023 Frontiers Media S.A. All rights reserved
個人情報保護方針
|
利用規約
この記事が気に入ったらサポートをしてみませんか?
