食餌性タンパク質が腸管CD4+ T細胞のプロファイルとレパートリーを形成する
記事|2023年5月16日
食餌性タンパク質が腸管CD4+ T細胞のプロファイルとレパートリーを形成する
エインズリー・ロックハート
,
オーブリー・リード(Aubrey Reed
,
ティアゴ・レゼンデ・デ・カストロ
,
カルビン・ハーマン
,
マリア・セシリア・カンポス・カネッソ
,
ダニエル・ムシダ
著者・論文情報
J Exp Med (2023) 220 (8): e20221816.
https://doi.org/10.1084/jem.20221816
記事履歴
スタンダードビュー
別ウィンドウでPDFを開くにはここをクリックPDFfor
共有
ツール
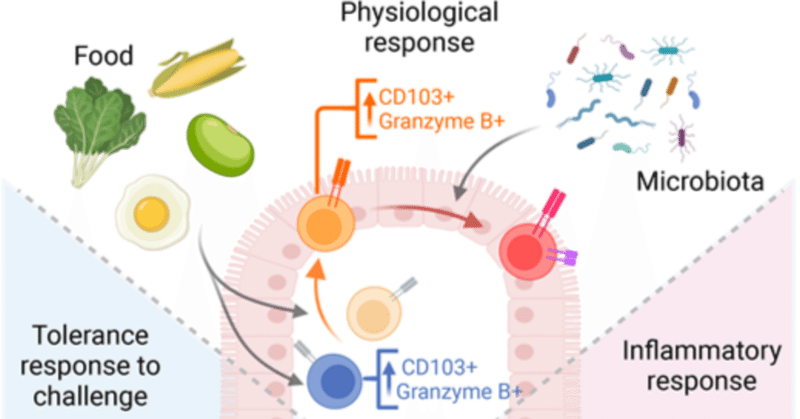
腸管免疫系は、アレルギーを回避するために食物抗原を許容する必要があり、そのプロセスにはCD4+ T細胞が必要である。抗原を定義した食事とグノトビオティックモデルを組み合わせることで、食事と微生物叢が腸内CD4+ T細胞のプロファイルとT細胞受容体レパートリーに明確に影響を与えることを示す。微生物叢とは無関係に、食物タンパク質は、腸上皮における抗原経験CD4+ T細胞の蓄積とクローン選択に寄与し、従来型および制御型CD4+ T細胞(Tregs)の両方に細胞毒性遺伝子を含む組織特異的転写プログラムを刷り込んだ。食物に対するこの定常状態のCD4+ T細胞応答は、炎症性チャレンジによって破壊され、この文脈における食物アレルギーからの保護は、Tregクローンの拡大および炎症性遺伝子発現の減少に関連していた。最後に、食物抗原を認識する定常状態の上皮適応CD4+ T細胞と寛容誘導Tregの両方を同定し、食物に対する不適切な免疫反応を防止するために、両方の細胞タイプが重要である可能性が示唆されました。
グラフィカルアブストラクト
大きく表示する スライドをダウンロードする
テーマ
粘膜免疫学、トレランス
はじめに
食物アレルギーを防ぐためには、免疫系が許容しなければならない食物由来の抗原が、毎日大量に腸から吸収される。抗原の経口投与により、その抗原に対する局所的および全身的な耐性が生じる重要なメカニズムである経口耐性には、特に制御性T細胞(Tregs;Garsideら、1995;Hadisら、2011;Josefowiczら、2012;Mucidaら、2005;PabstおよびMowat、2012)を含むCD4+ T細胞が必要です。しかし、食事性抗原に対するT細胞応答は、主にモノクローナルT細胞受容体(TCR)トランスジェニックシステムを用いて特徴づけられており、生理学的な免疫応答を表すものではありません。TCR特異的選択と機能分化を含む食物に対するポリクローナルCD4+ T細胞応答は、ほとんど解明されておらず、寛容とアレルギーのメカニズムを理解する上で重要である。
CD4+T細胞は、腸の2つの主要な隣接組織区画、前庭(LP)と上皮(IE)を占め、これらは基底膜によって分離され、免疫学的に区別されている。腸の炎症に重要なメディエーターであるトレグは、LPには豊富に存在するが、選択性の高いIEには比較的少ない(Sujino et al.、2016)。IEに移動すると、従来のCD4+ T細胞とTregの両方が、組織残留性(CD103、CD69)、細胞毒性(グランザイム)、ナチュラルキラー機能(NKG7)、およびCD8+ T細胞系(Runx3、ロンドンら、2021、ムチダら、2013、レイスら、2013、スジノら、2016)関連の遺伝子をアップレギュレートしながら特殊な転写プログラムを段階的に獲得し得る。この分化プロセスの終点において、IE適応CD4+ T細胞はCD8ααホモダイマーをアップレギュレートし、これは高い活性化閾値をもたらすTCRシグナルを減衰させることが提案されています。(Cheroutre and Lambolez, 2008)。我々の最近の知見は、IE適応CD4+ T細胞がTregに対して補完的な抗炎症の役割を果たし、Tregがまれな腸上皮において重要な調節機構を提供することを示唆している(Bilateら、2016、2020;Bousbaineら、2022;Sujinoら、2016年)。Tregの生成(Bouziatら、2017;Josefowiczら、2012;Torgersonら、2007)または上皮T細胞プログラミング(Reisら、2013;Sujinoら、2016)の破壊は、腸の炎症につながる。さらに、IE適応CD4+ T細胞は、食事性抗原に対する免疫制御に貢献することができる(Sujino et al.、2016)。しかし、IE適応CD4+ T細胞は、炎症誘発能を有し、セリアック病に見られるような食事性抗原に対する調節不全反応において病的な役割を果たすこともある(Abadie et al., 2012, 2020; Costes et al., 2019; Fina et al., 2008).
ここで、我々は、食事性タンパク質に対するポリクローナル腸CD4+ T細胞応答を特徴付け、腸における彼らの一般的な定常状態の運命は、細胞毒性関連遺伝子の発現を含む上皮居住に関連した転写プロファイルの獲得であることを実証する。さらに、食物に対する腸管T細胞応答が、活動的な耐性またはアレルギーにおいて、それぞれTregまたは炎症性Tヘルパー細胞の組織流入を促進するように変化することを実証した。これらの知見は、トレッグに加えて、上皮に適応したCD4+ T細胞が食物に対する恒常的な免疫反応に寄与していることを示唆している。
結果
食事シグナルは、腸上皮における成熟CD4+ T細胞の蓄積を促進する
食物タンパク質に対する腸管T細胞の応答を特徴付けるために、我々は遊離アミノ酸を含むタンパク質抗原なし固形飼料(AA)を開発した。AA食はポリペプチドを含まないが、単純および複合炭水化物(スクロース、コーンスターチ)、脂質(コーンオイル)、繊維(セルロース;表S1)を含む他の食物高分子を含んでいる。対照的に、標準的なチャウ食は非常に複雑で、数百種類の異なるタンパク質と多様な食餌代謝物を含む全食品成分(小麦、トウモロコシ、大豆など)で構成されています。したがって、食事療法は、食事による抗原特異的なTCR刺激、食事代謝物による免疫コスト刺激(一部は微生物叢に依存する)、食事による微生物叢の変化による二次刺激を通じて、腸内T細胞に影響を与える可能性があります。
AA食で離乳させた特定病原体フリー(SPF)C57BL/6マウスは、体重増加、便中リポカリン-2による腸の炎症、血清栄養バイオマーカーにおいて標準的なチャウ食マウスと同様だった(図S1、AおよびB)。8週齢のAA食マウスでは小腸の長さが減少していたが(図S1 C)、組織学的検査では組織の損傷や炎症は認められず、組織構造にもほとんど差がなかった(図S1 DおよびE)。最後に、SPF AA食マウスとチャウ食マウスの腸管ミエロイド細胞頻度に有意差は見られなかった(図S1 F)。総じて、AA食マウスは健康であり、腸の損傷や炎症の証拠はない。
我々は、フローサイトメトリーにより、2つの主要な腸内T細胞コンパートメント、IEとLPを評価し、AA食と比較して、チャウに離乳した8週齢マウスの小腸IEでTCRαβ+ CD4+T細胞の増加を発見した(図1 A)。総TCRαβ+ T細胞は、チャウ食マウスの小腸LPにおいても増加した;しかしながら、チャウ食はこのコンパートメントにおけるCD4+ T細胞の頻度の増加を促進しなかった(図S2 A)。組織適応したプレCD8α+(CD103+ CD8αα-)およびCD8α+サブセット(Londonら、2021)を含む抗原経験CD44+ CD62L-細胞は、チャウ食への曝露により誘導されたIE CD4+T細胞の大部分を占めた(図1、BおよびC)。IE Tregの絶対数もチャウ食でわずかに増加したのに対し、それらはCD4+のうち相対的な頻度で減少したことから、食餌シグナルへの曝露がTregよりもIE適応サブセットを優先することが示唆された(図1、B、およびC)。さらに、このことは、AA食マウスにおいて、TregのCD4+ CD8αα+ T細胞への上皮転換が損なわれている可能性を示唆しているのかもしれない(Sujino et al., 2016)。
LP CD4+ T細胞のうち、チャウ食マウスは、AA食と比較して、CD44+細胞の絶対数がわずかに増加したが、Tregは増加しなかった(図S2 B)。さらに、無菌環境においてチャウ食に反応すると報告されている(Kim et al., 2016)Rorγt-末梢性誘導Treg(pTregs)の増加は、IEまたはLPのいずれにおいても見られなかった(図S2 C)。我々は、食物吸収の主要部位である小腸において、食物タンパク質の影響が最も大きいと予想した。これと一致して、CD44+ CD4+、プレCD8αα+(CD103+ CD8α-)、またはTregを含む大腸IE CD4+ T細胞のチャウ関連増加は認められず、CD4+ CD8αα+ T細胞はわずかに増加しただけだった(Fig. S2 D). 全体として、これらのデータは、複雑な食事が小腸CD4+ T細胞の成熟と上皮適応を促進し、一方、大腸とLPは比較的影響が少なく、食物に対する明確で局所的なT細胞応答を示していることを実証しています。
T細胞の表現型に対する食事性抗原の影響を高度に制御された方法で評価するため、次に、タンパク質が多様な食事に含まれる単一の食物タンパク質の量をモデル化することを目的として、AA食に低用量のOVA(飲料水中0.1%、約5 mg/日)を添加した。この用量で飲料水中に 2 日間供給した OVA は、in vivo で腸管排泄リンパ節において OVA 特異的 TCR トランスジェニック CD4 T 細胞を刺激するのに十分だった(図 S2 E)。AA + OVAを与えたSPF C57BL/6マウスは、頻度は変わらなかったが、AA食マウスと比較して、総IE CD4+T細胞、CD44+ CD4+ T細胞、およびCD4+ CD8αα+ T細胞の絶対数がわずかに、しかし有意に上昇した(図S2 F)。プレCD8αα+(CD103+ CD8αα-)、IEからのTreg、または試験したLP T細胞サブセットに差は見られなかった(図S2、FおよびG)。異なる、あるいはより多様な食物タンパク質が、腸内CD4+ T細胞にさらに影響を与えるかどうかを調べるため、SPFマウスを、牛乳カゼインまたはカゼイン、グルテン、大豆タンパク質の混合物を補充したAA食に離乳させた。近位小腸におけるIE CD4+CD8αα+T細胞とTregの頻度を比較したところ、AAとカゼインでは差がなかったが、カゼイン-グルテン-大豆食ではCD4+CD8αα+T細胞が増加しTregが減少しており、TregからCD4+CD8αα+への分化経路(Sujino et al, 2016)がより多様なたんぱく質の摂取を行うマウスにおいて誘導されていると考えられた(図S2 H).これらの結果は、食事性タンパク質がSPFマウスにおいてIE CD4+T細胞の蓄積と成熟を促進し、カゼイン-グルテン-大豆または標準的なチャウ食のような食事性タンパク質の多様性を高めると、相加効果がある可能性を示している。
食事シグナルは、微生物に依存しないCD4+ T細胞の上皮および細胞傷害性プログラミングを促進する
食事は腸内細菌叢に大きな影響を与え、それが局所免疫細胞への間接的な下流効果につながる可能性がある(Sonnenburg and Bäckhed, 2016). 実際、SPF AAとチャウ食マウスの16S rRNA配列決定により、腸内細菌叢の組成が異なることが明らかになり、チャウ食は小腸と盲腸の両方で微生物の多様性の増加を促進した(図2、A-C;図S2、IとJ;および表S2)。免疫細胞に対する微生物に依存しない食事の影響をより明確にするために、無菌(GF)マウスと、マウス腸内の主要な細菌群のメンバーを代表する12種類の常在菌の安定した垂直伝播性のコンソーシアムであるOligo-MM12でコロニー形成したgnotobioticマウス(Brugiroux et al, 2016)で食事モデルを確立しました。GF AA-dietマウスは、外来タンパク質抗原への曝露がなく、微生物叢に依存しない食事の影響を評価する理想的なモデルを提供し、Oligo-MM12は、腸内抗原の多様性と複雑性を制限しつつ、常在菌からの何らかの共刺激を伴うより生理的なモデルを提供します。
SPFと同様に、GF AA食マウスでは、腸の長さがチャウ食と比較して減少し(図S1 C参照)、組織には損傷、炎症、大きな形態変化の証拠は認められなかった(図S1 DおよびE参照)。GF chow dietマウスでは、LPマクロファージの頻度が上昇したが、骨髄コンパートメント内では他の食餌間の差は観察されなかった(図S1 Fを参照)。
GFおよびOligo-MM12マウスは、SPF AA食マウスで見られたレベルと同様に、腸内CD4+ T細胞が非常に減少しており(図2 D;および図S2、KおよびL)、定常状態の腸内T細胞蓄積には、複合食と複合微生物叢の両方が必要であることを示唆している。GFまたはOligo-MM12マウス内で、チャウ食はAA食と比較してIE TCRαβ+T細胞を再び増加させた(図2 Dおよび図S2 K)。CD4+T細胞頻度は影響を受けなかったが、チャウ食はCD44+、プレCD8αα+(CD103+ CD8αα-)、CD8α+ CD4+ T細胞サブセットの頻度を増加させ(図2 Dおよび図S2 K)、食事シグナルが微生物叢とは独立してIE CD4+ T細胞の成熟と組織適応を促進することが示された。
食事が腸内CD4+ T細胞の機能的遺伝子発現経路にどのように影響するかをより詳細に調べるため、AA、AA + OVA、またはチャウを与えたGFまたはOligo-MM12マウスのIEおよびLPからCD4+ T細胞の単一細胞RNAシーケンス(scRNAseq)をChromium 10X (10X Genomics) プラットフォームを用いて行った(図S3 A).配列決定された細胞は、上位の差次的発現遺伝子に基づいて定義した11の主要な不偏クラスターに割り当てられた(図2 E、図S3 B、および表S3)。特にGFまたはAA食マウスでは、ナイーブ細胞やNKT細胞が高頻度に観察された(図2 F)。逆に、IEでは3つのクラスターを形成し、LPではTh1型遺伝子で定義される単一の主要クラスターを形成する成熟細胞は、Oligo-M12またはchow食マウスの頻度が高くなっていた(図2 Fおよび図S3 C)。
IEへの段階的CD4+ T細胞転写適応に関連するシグネチャー遺伝子の発現を評価し(London et al., 2021)、我々のIE成熟クラスターがシグネチャー遺伝子獲得の3段階を表すことを発見した(図2 G, 図S3 D, および表S3)。IE 1細胞は上皮への適応度が最も低く、Nkg7、Ccl5、Cd160を発現し、これらは3つのクラスターすべてで発現していた(図2 G)。IE 2は、CD103(Itgae)、および顆粒酵素(Gzma、Gzmb)を発現する、我々のプレCD8αα+集団と一致し、コロニー形成に関わらずAAまたはAA + OVAに対してチョウ食で富化した(図2、FおよびG;ならびに図S3 E)。IE 3は、完全にIEに適応したCD4+ CD8αα+T細胞を表し、さらにCd8a、Lag3、Prf1(Perforin)、Cd7を発現し、すべての配列決定グループで稀であり、複合微生物叢への依存性を確認した(図2、FおよびG、および図S3 E).IEシグネチャー遺伝子の発現を条件間で比較すると、IE1遺伝子(Nkg7、Ccl5、Cd160)はより均一に発現していたのに対し、IE2遺伝子(Itgae、Gzma、Gzmb)はGFとOligo-MM12の両方のchow dietマウスでほぼ独占的に発現していた(図2 H、それぞれのコロニー内で食事により著しく発現上昇する遺伝子は*で示した)。実際、10個のIEホールマーク遺伝子すべてを用いて遺伝子シグネチャーを作成したところ、GFおよびOligo-MM12の両方で、チャウ食マウスのCD4+ T細胞はAAまたはAA + OVA食マウスよりも高いスコアを示した(図2 I)。GFおよびOligo-MM12におけるAA、AA + OVA、またはチャウ食マウスの差分遺伝子発現の偏りのない三者比較により、イトガエおよび顆粒酵素を含むIEシグネチャー遺伝子がチャウ食マウスのトップアップレギュレーション遺伝子に含まれていることがさらに確認されました(図2のJおよび表S4)。
マウスに単一の食物タンパク質(AA+OVA)を与えても、フローサイトメトリー(データ示さず)またはscRNAseq(図2 F;および図S3、C-E参照)のいずれによっても腸内CD4+T細胞サブセットの頻度は変化しなかったが、Oligo-MM12 AA+OVAマウスからのIE CD4+ T細胞はAAと比較してItgaeを有意に上昇させ、Gzmaの増加傾向を示した(P = 0.007; 図2 H).この差はGFでは見られなかったことから、単一食品タンパク質の低用量は、微生物叢からのコスト刺激を必要とするかもしれない方法で、CD4+ T細胞の何らかの上皮適応を促進するのに十分であることが示唆される。
総Treg頻度は、GFまたはOligo-MM12において、食餌間で大きく変化しなかった(図2 Fおよび図S3 F参照)。しかし、Rorγt- pTregは、GFおよびOligo-MM12においてAA食と比較してchow食で増加し(図S3 F)、これらの細胞が食物に応答するという以前の報告(Kimら、2016)を裏付ける。次に、scRNAseqデータセット内のTreg転写プログラムを評価し、IEシグネチャー遺伝子(Nkg7、Gzma、Gzmb;図S3、GおよびI;ならびに表S5)を上昇させるものを含む8つのサブクラスターを特定した。IEシグネチャーTregは、コロニー形成に関係なくチョウ食で拡大し、Oligo-MM12からAA + OVAマウスで増加する傾向にあった(図3、AおよびB)。これらの細胞は、CD4+ CD8αα+分化に向かう軌道にある以前に同定されたpTregと転写的に類似していた(Bilate et al.、2020)。対照的に、Il10-high Tregは、Oligo-MM12においてほぼ独占的に見出され(図3 A及び図S3 J)、Rorc発現はTregの中でもこの集団に限定されていた(データは示さず)。したがって、この集団は、微生物叢に依存することが知られているRorγt+ pTregs(Sefikら、2015;Yangら、2016)を表す可能性がある一方、IEシグネチャーpTregsは、食事に依存すると報告されているRorγt- pTregs(Kimら、2016)を表すかもしれない。実際、コロニー形成状態にかかわらず、チャウ食TregはIE2シグネチャー遺伝子(Itgae、Gzma、Gzmb)をアップレギュレートし、IE遺伝子シグネチャー全体で高いスコアを示した(図3、CおよびD)。GFおよびOligo-MM12におけるAA、AA + OVA、またはチャウ食トレグ間の差分遺伝子発現の偏りのない三者比較により、さらにチャウ食はIEシグネチャー遺伝子のアップレギュレーションを引き起こしたことがわかった(Itgae、 Gzma、Gzmb、およびLag3)およびコアTreg抑制機能遺伝子(Il10、Ctla4)が上昇する一方で、AAまたはAA + OVA食では、異なる天然Treg関連転写プロファイル(Gata3、Nrp1、Cd81、およびKlrg1)が濃縮されていた(Fig. 3 E および Table S6).
総IE CD4+T細胞における我々の知見と同様に、低用量のOVAはGFマウスにおけるTreg転写プロファイルに影響を与えなかったが、Oligo-MM12におけるItgae発現を促進するには十分であった(図3 C)。これに対応して、全CD4+ T細胞とTregの両方において、Oligo-M12は、同じ食餌のGFマウスよりもGzmaとItgaeを含むIEシグネチャー遺伝子の発現を促進したが(図2 Hおよび図3 C;Ȩで示す)、これは食物タンパク質の存在に依存した。したがって、食物タンパク質への曝露は、抗原を経験したCD4+ T細胞を腸管上皮に呼び込み、微生物のコスト刺激によって増幅される形で、組織常在性の細胞傷害性遺伝子発現シグネチャーを刷り込んでいる。
複雑な食事は、腸内T細胞におけるグランザイムBの発現を誘導する
scRNAseqで得られた知見をさらに検証するために、フローサイトメトリーで腸管T細胞のグランザイムBの発現を評価した。実際、チョウ食は、GFとSPFの両方で、IE CD4+T細胞とTregの合計でグランザイムBの発現を促進した(図4 A)。さらに、CD8αβ+ T細胞、胸腺TCRγδ+およびCD8αα+ TCRαβ+T細胞では、グランザイムBの発現がより高かった(図4 B)。食事によって誘導されたCD4+およびCD8αβ+ T細胞のグランザイムB発現は、微生物叢の存在下で高められたが、胸腺由来のT細胞サブセットへの影響は微生物叢に依存しなかった。したがって、複雑な食事は腸内T細胞におけるグランザイムBの発現を広く誘導するが、誘導の経路はサブセットによって異なる可能性がある。
腸内CD4+ T細胞に対する食事の刷り込みの動態を特徴付けるために、SPFマウスを3週(離乳前)、8週、または12週で分析した。8週齢までAAまたはチャウ食を与えていた一部のマウス(すなわち、先行実験の標準プロトコルに従う)を、12週齢の終点分析まで逆の食餌に切り替えた(図4のC)。この実験セットアップにより、成体マウスから食餌シグナルを除去した場合(Chow から AA)、または複合食への曝露を成体まで遅らせた場合(AA から Chow)、腸 CD4 T 細胞の運命を評価することができました。離乳前(3週齢)のマウスは、CD8αα+またはGranzyme B+ CD4+T細胞の頻度が非常に低かった(図4 D)。その後、チャウ食に切り替えたマウスは、8週齢、そして12週齢までにこれらの細胞の頻度が徐々に増加したが、AA食マウスは離乳期のような未熟なIE CD4+T細胞プロファイルを維持した(図4 D)。8週齢でAA飼料をチャウ飼料に切り替えると、CD4+ CD8αα+ T細胞頻度は回復しなかったが、グランザイムB発現は回復した(図4 D、赤四角)。逆に、8週目にAA食に切り替えたマウスでは、チャウで誘導されたCD4+ CD8αα+ T細胞は持続したが、グランザイムBの発現は減少した(図4 D、青四角)。これは、離乳期および発育期に、食事が腸内CD4+ T細胞に上皮シグネチャーを刻み込む重要なウィンドウを示す可能性がある。一方、CD4+ CD8α+が発達すると、チャウ食なしで少なくとも4週間は持続することができる。対照的に、グランザイムBの発現は、複雑な食餌に最近さらされたかどうかに依存している。
我々のこれまでの研究で、CD4+ T細胞によるIE適応が、グランザイムBのアップレギュレーションと関連していることが示されている(Bilate et al., 2020; London et al., 2021; Mucida et al., 2013; Reis et al., 2013)。しかし、これらの食餌スイッチ実験では、Chow-to-AAマウスが、もはやグランザイムBを発現しないIE適応CD4+ T細胞を有するように、グランザイムB発現とIE適応が切り離された(図4 E)。全体として、これらのデータは、食事シグナルと微生物叢が、CD4+ CD8αα+ の分化と腸内CD4+ T細胞におけるグランザイムBの発現の両方を促進することを示しているが、時間的ダイナミクスとしたがって誘導の経路は異なるようだ。
食事性タンパク質への曝露は、腸内CD4+ T細胞のクローン選択を促す
次に、AA、AA + OVA、またはチャウを与えたGFおよびOligo-MM12マウスのscRNAseqからTCRを分析することにより、食事性タンパク質への日常的な曝露が、腸内CD4+ T細胞のTCRレパートリーをいかに形成するかを評価しました。IEおよびLP成熟CD4+ T細胞は、複雑な微生物叢がないにもかかわらず、全体的に高度にクローン的に拡大し、一方、ナイーブ細胞およびNKT細胞(不変のTCRαを使用するが多様なTCRβ)は、予想通り高度に多様でした(図5 A)。レパートリーを0(最も多様性が低い)から0.5(最も多様性が高い)までスコア化したD50を用いてサンプル群間のレパートリー多様性を推定したところ、GF AAまたはAA + OVAでは他の群に比べクローン多様性が高い傾向が見られた(図5 B)。しかし、成熟した(IE1、IE2、IE3、LP Th1)CD4+T細胞またはTregでは、サンプルグループ間でレパートリーの多様性に大きな違いは見られませんでした(図5 B)。これは、大きな外来抗原にさらされない腸では、成熟CD4+T細胞の蓄積は少ないものの、蓄積したものは依然としてクローン的に拡大していることを実証しています。我々は最近、GF AA食マウスにおいて腸関連胚中心が非常に減少していることを報告したが、これらの胚中心からのB細胞は依然としてクローン選択と拡大を示しており(Nowosadら、2020)、腸のB細胞についても並行してメカニズムがあることを示唆している。
食物抗原が腸管CD4+ T細胞のクローン選択を駆動できるかどうかを決定するために、同一のペアTCRαおよびTCRβ CDR3アミノ酸配列によって定義される、マウス間の共有(パブリック)クローンを調べた(図5、CおよびD)。外来抗原の主な供給源が食物であるGFマウスのうち、AA食マウスの間ではクローンの重複が見られず、AA + OVAマウスの間では低レベルのクローン共有が見られ、chow食マウスの間では広範なクローン共有が見られ、食物抗原がクローン選択を促進している可能性が示された。さらに、AA+OVAマウスとチャウ食マウスの間に少量のクローン重複を認めたが、これは自己または未認識の環境抗原によって駆動されている可能性がある。Oligo-MM12マウスでは、微生物抗原の認識や微生物叢からの共刺激効果によるものと思われ、全般的にクローンの重複が見られた。しかし、Oligo-MM12の食餌間では、GFと同様の傾向がみられ、AA食マウス間ではクローンの共有はなく、AA + OVAマウス間では低レベルの共有、chow食マウス間ではより高度な共有が認められた。GFマウスとOligo-MM12マウスでクローンの重複を比較したところ、AA + OVAマウス間では追加の共有が見られたが、chow dietマウス間ではクローンの共有度が高いことがわかった。これらのデータは、食事性抗原による腸管CD4+ T細胞のクローン選択を示唆しており、単一の食物タンパク質の低用量への曝露は低レベルのクローン選択をもたらし、複雑なチャウの文脈で多様な食物タンパク質に曝露すると高レベルの選択をもたらす。この場合、多様な食物代謝物の存在は、食物抗原特異的T細胞の分化と拡大を促進する付加的なアジュバント効果を有すると考えられる。
食物タンパク質に対する腸管CD4+ T細胞応答を形成する曝露の文脈
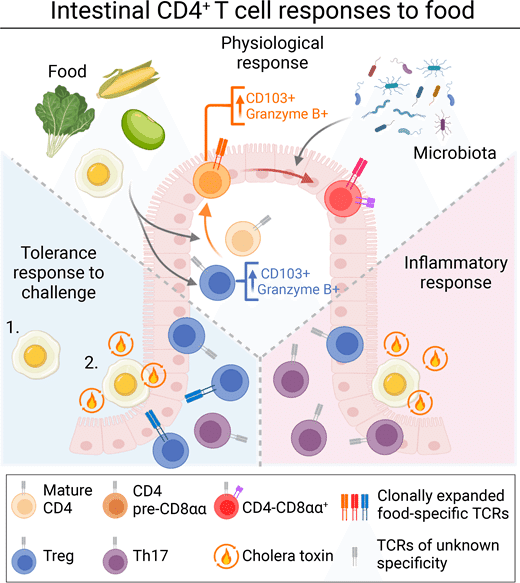
食物誘導腸CD4+ T細胞が寛容にどのように寄与するか、また食物アレルギーの文脈でこれらの応答がどのように撹乱されるかをよりよく理解するために、ナイーブT細胞運命マッピングマウスモデル(iSellTomato;Merkenschlagerら、2021)と食物アレルギーコレラ毒素(CT)モデルマウス(Jimenez-Saizら、? 2017)。CD62L+ナイーブ細胞をタモキシフェン投与時にTomato蛍光で永久標識するiSellTomatoでは、タモキシフェン標識時以降に成熟して腸に入った「元ナイーブ」(Tomato+ CD62L-)T細胞を同定できる(Parsa et al., 2022)。我々はこの戦略を利用して、3つの文脈で食事性タンパク質に応答する腸管CD4+ T細胞を濃縮した: (1) 摂食、ここではマウスにOVAを与え、食物に対する定常状態の応答を表す;(2) アレルギー、ここではOVAへの一次曝露がCTとともに行われ、アレルギー感作をもたらす;および (3) 耐性、ここではマウスにOVA/CTの前にOVAのみを与え、経口耐性およびアレルギーに対する保護をもたらす(図6のA)。したがって、我々の治療プロトコルの26日目に分析したトマト+ CD62L-細胞は、4週間の治療プロトコルの間に成熟して腸に入ったすべての細胞を含み、OVAに反応する細胞に富んでいるが、CD4+ T細胞が腸に絶えず募集されているので、CT、微生物叢および他の食物抗原に反応する細胞も含まれるかもしれない。26日目の時点で、アレルギー群のマウスは、血清総IgEおよびOVA特異的IgG1の上昇を示し、アレルギー感作を示すが(図6 B)、腸組織損傷または炎症に観察可能な増加はない(図S4 A)。OVA/CTの4wk用量についてこのプロトコルを継続すると、全身性OVAチャレンジは、アレルギーマウスにおいてアナフィラキシーをもたらすが(Jimenez-Saizら、2017)、耐性グループのマウスは保護される(図6 C)。
最初のタモキシフェン投与から4wk後、OVA給餌プロトコルのSPFマウスからの小腸IEまたはLP CD4+T細胞の約15%がTomato+ CD62L-であり(図6 D)、定常状態で腸へのCD4+T細胞流入のベースラインを表している。両組織において、CD4+ T細胞流入はOVAアレルギーマウスで2倍以上増加し、一方OVA耐性は摂食とアレルギーの間で低下しました(図6 D)。対照的に、大腸では、寛容マウスまたはアレルギーマウスにおいてCD4+ T細胞流入の増加はなく(図S4 B)、炎症性の文脈における食物への応答は、小腸に局在したままであることを示唆した。トマト(既存の非ナイーブ)CD4+ T細胞のうち、各処理群のマウスは、フローサイトメトリー分析に基づくIE2、IE3、Treg、およびIL-17AまたはIL-4を産生する細胞の頻度が類似していた(図6 E; および図S4、CおよびD)。しかしながら、浸潤するTomato+ CD4+T細胞のプロファイルは、OVA摂食、耐性、およびアレルギー条件において異なっており、iSellTomatoが、異なる文脈で食物タンパク質に応答するポリクローナル腸内CD4+ T細胞を濃縮し特徴付けるための有効なシステムであることが示唆される。OVA耐性では、Tomato+ CD4+T細胞でTreg頻度が高かったが、OVAアレルギーでは、IL-17A発現が増加し(図6 Eおよび図S4 D)、CTが微生物叢依存的に腸内Th17を誘導するという先行報告(Zhaoら、2017)と一致する。C57BL/6マウスは、CTアレルギーモデルにおいて典型的に強いTh2応答を有しておらず、アレルギーマウスからのTomato+ CD4+ T細胞におけるIL-4産生の有意な増加は見られなかった(図S4、CおよびD)。3つの処理グループすべてからのIE Tomato+ CD4+ T細胞は、4wkの過程で上皮プロファイルの部分的な獲得のみを示し、約25%がCD103(IE2)を発現したが、CD8αα(IE3;図S4 C)を発現する細胞はほとんどなかった。
摂食、耐性、アレルギーの文脈でOVAに対するCD4+ T細胞応答を転写的に特徴付けるために、Tomato+ CD62L-(fat-mapped元ナイーブ)およびTomato- CD62L-(既存の非ナイーブ) CD4+ T細胞(約1の比率でプール)でscRNAseqを実行しました: 治療プロトコルの26日目にSPF iSellTomatoマウスのIEおよびLPから、Tomato+細胞を均一に濃縮するために2 Tomato+:Tomato-でプールした(図6 A参照)。6 A)。scRNAseqは、2つの独立した実験および配列決定ランにわたってプールされた条件ごとに4〜5匹のマウスを用いて、10X Genomicsプラットフォームで実施した(図S4、EおよびF)。scRNAseqデータで定義されたTomato-およびTomato+集団は、真の既存の成熟細胞対元ナイーブ細胞を濃縮するが、運命マッピングマウスモデルの不完全な効率、TdTomato/Stopの低い検出、または分化して腸に入る可能性のあるSell+中枢記憶細胞の標識のために、各集団の中にいくつかの汚染細胞が存在し得る。
配列決定された細胞は、上位の差次的発現遺伝子に基づいて定義した11の主要な不偏クラスターに割り当てられた(図7 A; 図S4、G、H; および表S7)。トマト+細胞はすべてのクラスターに存在したが、CCR7+移動性細胞とTregの間で特に濃縮され、IE適応クラスターでは最も低かった(後述;図S4 F)。C57BL/6 CTアレルギーモデルにおける弱いTh2応答と一致して、ホールマークTh2遺伝子によって定義される遺伝子発現クラスターは同定されなかった(図S5 A)。IEシグネチャー遺伝子の発現に基づき、GF/オリゴ-MM12データセットと同等のIE2およびIE3クラスターを同定したが、IE1クラスターはなかった。これは、CD4+ T細胞IE適応に対するチャウ食と完全コロニー化の相加効果という考え方をさらに補強した(図7 A、図S5 B、および表S7)。追加のIEクラスターが同定され(IE4)、これはIE3のシグネチャーに加え、NK機能(Fcer1g、Tyrobp、Klrd1)、Treg機能(Ikzf2)、およびアポトーシス(Tox、Bcl2)に関連する追加の遺伝子を発現し、Cd4レベルは低下した(図S5のB、表S7のB)。トマトの標識頻度は、IE2(40%)→IE3(15%)→IE4(5%)と徐々に減少し、4週間の経過で上皮性プロファイルの部分的獲得のみが起こることが確認された(図S5 C)。グループ間のクラスター分布を比較すると、IE4細胞はほぼTomato+のみで、寛容マウスまたはアレルギーマウスのものであった(図S5 Dおよび図7 B)。実際、IE Tomato-細胞間の三元差分遺伝子発現解析では、IE4遺伝子が摂食群と比較して寛容群およびアレルギー群で非常に濃縮されていることが示された(図S5 Eおよび表S8)。これらのデータは、炎症(CT曝露)状態において、既存の組織常在IE3細胞でIE4遺伝子発現プログラムがオンになったことを示唆している。したがって、IE4は、以前に示唆されたように(Bilate et al., 2020)、自然受容体を介して抗原非依存的に機能することができるIE適応CD4+ T細胞の活性化状態を表していると考えられる。注目すべきは、IE4転写プログラムは、腫瘍と定常状態の腸上皮の両方で見られる胸腺由来(CD4-)CD8αα+ TCRαβ+細胞と類似している(Chou et al., 2022; Denning et al., 2007)、ただしIkzf2の添加があり、何らかの調節能力を示唆していると考えられる。
OVA摂食、耐性、またはアレルギーの際に成熟して腸に入るCD4+ T細胞が異なる特性を持つかどうかを評価するために、これらのグループ間でTomato+細胞のクラスター分布を比較しました。フローサイトメトリーによる我々の観察と一致し、OVA耐性マウスはTregの頻度が増加したのに対し、アレルギーマウスはIE Tomato+細胞の中でTh17が増加した。LPでも有意ではないが同様の傾向が見られた(図7 Bおよび図S5 F)。三元配置による遺伝子発現の差分から、OVA摂取マウスのIEおよびLP Tomato+細胞は、組織残留性およびホーミング(Zfp683、Ccl5)、メモリー(Ccr7、Satb1、Bach2)、I型免疫応答(Ly6a、Ifi47)に関わる遺伝子を発現上昇させた。対照的に、OVAアレルギーTomato+細胞は、Th17関連遺伝子(Il17a、Il22、Rorc、Them176a;図7 C、表S9、およびS10)をアップレギュレートした。OVA耐性マウスは、そのアップレギュレートされた遺伝子の大部分を共有し、摂食またはアレルギーマウスはその中間炎症表現型を示した。しかし、LPでは、耐性のみがTreg関連遺伝子(Foxp3、Ikzf2、Itgav、Cd83;図7 C)をアップレギュレートした。最後に、耐性とアレルギーの両方から得られたLPトマト+細胞は、このシーケンスデータセットでTregで主に発現していたGata3を上昇させた(図7 Cおよび図S5 A)。
次に、Tregの転写プログラムを詳細に評価し、上位の異なる発現遺伝子に基づいて5つのサブクラスターを特定した(図S5 Gおよび表S11)。Tomato+ Tregsの中で、サブクラスタの頻度は、条件によって変化した。定常状態(OVA摂食)では、流入トレグの約50%がIl10高pTregであったが、OVA耐性またはアレルギー条件では約20%に低下した(図7 D)。これらの環境における流入トレグは、代わりにHelios+(Ikzf2)細胞が濃縮される傾向にあり(摂食 vs. アレルギー P = 0.059)、これらは主にアレルギーではGata3-high、耐性ではGata3-intermediateだった(図7のD)。トマト+トレグもまた、条件間で主要な機能性分子の発現が異なっていた(図7 E)。定常状態と比較して、耐性とアレルギーの両条件では、Ctla4が抑制され、Ikzf2が発現上昇した。耐性トマト+トレグは、他の2つの条件と比較して、Il10とGzmbを低下させたが、アレルギーではGzmbが促進された。アレルギーではさらに、Cd83が減少し、Socs2が増加することが特徴的であった。したがって、寛容またはアレルギーの活動時に腸に浸潤するトレグは、それぞれの機能的結果に寄与すると考えられる、異なる転写プロファイルを有している。
これらの知見は、食事と微生物叢の両方からのシグナルによって刷り込まれた定常状態のCD4+ T細胞の腸内浸潤と組織適応が、炎症性チャレンジによって破壊され、エフェクターCD4+ T細胞反応を引き起こすことを実証しています。炎症性チャレンジの間、食物タンパク質に対する免疫寛容の維持は、Tregの流入の増加と関連している。一方、OVA/CTアレルギーモデルでは、炎症性Th17の流入の増加が観察された。寛容とアレルギーの両方において、流入するトレッグの機能的遺伝子発現プログラムの変化と、常在するIE CD4+T細胞の転写プログラムの変化が見出された。これらのデータは、定常的な食物への曝露と、炎症性チャレンジによる食物への積極的な寛容の間には、異なる腸管免疫応答が存在することを実証しています。
食物に対する寛容および炎症性CD4+ T細胞応答のクローン動態と抗原特異性
摂食、耐性、またはアレルギーの文脈における、食品タンパク質に対するCD4+ T細胞応答のポリクローナル・レパートリー動態を理解するために、次に、scRNAseqデータセットからTCRを評価しました。定常状態では、ほとんどのクラスターで高度のクローン拡大が見られたが、TregとFR4高細胞はよりクローン的に多様だった(図8 A)。Tomato-細胞のTCRレパートリーは条件によって同じように多様であったが、Tomato+細胞はOVA耐性またはアレルギーでクローン拡大が増加する傾向にあった(図8、AおよびB)。最後に、耐性群ではTregのクローン拡大が増加し、アレルギー群ではTh17細胞の拡大が増加する傾向が見られたことから(図8 C)、これらのサブセットはそれぞれの条件下で同時に募集・拡大されていると考えられる。
最後に、scRNAseqデータセットで同定されたTCRが食事性タンパク質に特異的であるかどうかを評価しました。AA+OVA食を与えたGFマウス(すなわち、OVAが唯一の外来抗原源である)、またはOVA食または耐性条件のTomato+細胞から31個のTCRを選択し(表S12)、NFAT-GFPハイブリドーマ(Iseら、2010)において発現した。NFAT-GFPアッセイにおいて、2つのTCRがOVAに反応し、1つはOVA摂食から(TCR 18)、1つはOVA耐性から(TCR 316;図8 D)である。さらに、これら2つのTCRについて、重複するOVAペプチドライブラリーでの反応性を試験したところ、それらは両方とも、タンパク質の非切断シグナルペプチド内にあるエピトープOVA 26:40 (Fig. S5 H)に結合することがわかった。一方、免疫によってOVAに応答して生成されたOT-II TCRは、OVA 323:339 (Fig. S5 H) に結合する。このことは、食物タンパク質の経口曝露により生成されたT細胞応答が、免疫応答とは異なるエピトープを優先的に使用することを示唆していると考えられる。
トマト+OVA摂食マウスに由来するTCR 18は、高度に拡大し、主にIE2、IE3、およびTh1クラスタ内に見出された(図8 D)。Tomato+ OVA耐性に由来するTCR 316は、中程度に拡大し、Tregおよびサイクリングクラスター内にのみ見出された(図8 D)。さらに、これらのTregは主にIl10+サブクラスター由来であったことから、Il10+ Tregの浸潤は定常状態と比較して耐性では減少するが、これらの細胞は依然として経口耐性の維持において抗原特異的な機能的役割を担っていることが示唆される。これらの結果は、完全ポリクローナルシステムにおいて、食物抗原特異的T細胞応答は、活発な口腔内寛容だけでなく、自然摂食の過程でも生成されることを示すものである。
考察
ここで我々は、食物に対する腸管CD4+ T細胞のクローン動態と機能分化の包括的な解析を提供した。我々は、定常状態では、食物からのシグナルが、腸内CD4+ T細胞のクローン選択、上皮適応、および細胞毒性プログラミングを促進し、この経路は、微生物叢からのシグナルによってさらに後押しされることを発見した。食物に対する炎症反応の制御におけるトレグの要件は十分に確立されており(Mucidaら、2005;Torgersonら、2007)、食物タンパク質に対するLP Treg反応は、TCRモノクローナルモデル(Hadisら、2011)、MHCクラスIIテトラマー(Hongら、2022)、および抗原なし食物モデル(Kimら、2016)を使用して説明されている。我々のデータはさらに、食事で誘導された腸内のTregが、定常状態で細胞毒性および上皮残留に関連する遺伝子をアップレギュレートすることを示す。以前、我々はIE Tregが微生物叢依存的にCD4+ CD8αα+ T細胞に分化できること(Sujino et al., 2016)、そしてIEシグネチャー遺伝子を発現するTregはこの運命に向かう軌道にあると考えられることを報告した(Bilate et al., 2020)。これらのデータは、腸内の食事シグナルが、従来のCD4+T細胞だけでなく、Tregにおいても細胞傷害性IE適応の表現型を促進することを示唆している。
定常状態の食事シグナルはTregのGranzyme B発現を促進するが、アレルギーモデルで流入したTregはGranzyme Bをさらに上昇させ、寛容マウスでは抑制することが示され、この経路の制御がTregを介した免疫制御に重要である可能性が示唆された。活性化したエフェクター細胞のグランザイム依存的な細胞溶解は、Treg抑制機能の一つのメカニズムとして説明されており(Grossman et al.、2004)、細胞毒性が炎症促進および抗炎症反応の両方に寄与することが示唆されている。細胞傷害性特性を持つCD4+ T細胞は、ウイルス感染、自己免疫、または癌による慢性的な免疫刺激の設定でも記述されている(Cenerentiら、2022年)。我々の研究は、食物や常在菌への継続的な恒常的な曝露が、腸内で同様のCD4+ T細胞表現型を駆動することを示唆している。疾患状態で記述される細胞傷害性CD4+ T細胞と定常的な腸刺激によって誘導される細胞傷害性CD4+ T細胞との本質的な違いの一つは、TCR活性化の閾値を上げることが提案されているCD8ααの発現です(Cheroutre and Lambolez, 2008; Mucida et al., 2013)。したがって、IE適応CD4+ CD8αα+T細胞は細胞傷害性機械を備えているが、細胞傷害性メカニズムを活性化するためには、定常状態レベルを超える刺激が必要なのかもしれない。我々のデータは、炎症シグナル(コレラ毒素)と同時に食物タンパク質にさらされると、細胞傷害性遺伝子のアップレギュレーションを含む、組織に常駐するIE適応CD4+ T細胞の過活動状態を駆動することを示している。セリアック病では、IE適応CD4+ T細胞が炎症性の高いTh1様応答を示し、組織病理に寄与するが、これは食事からグルテンを除去すると消失する(Abadieら、2012;Costesら、2019;Finaら、2008)。したがって、Tregに加えてIE CD4+T細胞の調節不全または機能不全が、無害な腸管抗原に対する炎症反応を出現させるメカニズムの1つであると考えられる。しかしながら、細胞傷害性CD4+ T細胞に関する現在の機能的理解は限られている。例えば、CD4+ T細胞の細胞傷害性がMHC IIを介した同族抗原提示にどの程度依存するか、また、CD4を介した細胞溶解の正確な細胞標的は依然として不明である(Takeuchi and Saito, 2017)。CD4+T細胞細胞傷害が腸の寛容に寄与するのか、逆に調節不全に陥って組織損傷に寄与するのかを定義するためには、さらなる研究が必要である。
我々は、炎症シグナルが定常状態のCD4+ T細胞腸内適応を破壊し、エフェクター表現型に偏った細胞の流入を増加させることを示す。食物タンパク質に対する経口耐性が炎症性チャレンジの前に確立されていた場合、炎症性チャレンジ中に同じ食物タンパク質に曝露すると、炎症性CD4+ T細胞遺伝子発現の低下と食物アレルギーからの保護に関連する腸トレッグのクローン拡大が起こる。しかし、食物タンパク質の一次曝露が炎症刺激と同時に起こる場合、腸内ではTreg反応の低下と炎症性CD4+ T細胞遺伝子発現の増加が観察され、食物アレルギーの発症に関連することが分かっています。経口耐性にはpTregが必要であることが知られているが(Garside et al., 1995; Hadis et al., 2011; Mucida et al., 2005)、経口耐性時のTreg流入は、腸管抗原に反応しない胸腺Tregと典型的に関連するマーカーであるHeliosの発現増加と、IL10減少によって特徴づけられることがわかった。それにもかかわらず、耐性条件下で同定されたOVA特異的トレグは、Il10+クラスターのものであった。このことは、Il10+ pTregがポリクローナルな設定で抗原特異的耐性に寄与する一方で、増加したHelios + Tregは、抗原非特異的に組織保護に広く寄与する可能性を示す。ヒトのアレルギーは、一般的にアレルゲンに対するTh2反応と関連しているが、我々および他の研究者は、OVA/CTマウスモデルにおける腸管CD4+T細胞応答が、炎症性Th17に偏っていることを見出し、この効果は微生物叢に依存することが示されている(Zhao et al.、2017)。Th17がC57BL/6マウスのアレルギーに寄与するエフェクター細胞なのか、それとも単に粘膜アジュバント存在下での微生物叢に対する並行反応なのかは、依然として不明である。
iSellTomatoのデータは、食事性抗原に応答するT細胞の分化に重要なレパートリー動態が伴うことを実証しており、これまでTCRモノクローナルシステムの普及によって除外されていた結論です(Mucidaら、2005;Weinerら、2011)。さらに、食事性タンパク質への定常的な曝露が、腸管CD4+ T細胞のクローン選択に寄与し、食事性タンパク質を認識する組織由来のTCRクローンを同定する証拠を提供するものであった。我々の発見は、MHCクラスIIテトラマーを用いて、自然のポリクローナル環境における食事性抗原特異的CD4+ T細胞応答の生成を示した最近の研究を補完するもので、LPにおける主要な運命はTreg分化であった(Hongら、2022年)。我々のアプローチはエピトープ特異的な発見に依存しておらず、我々が同定したクローンは、免疫化によって以前に同定された免疫優性OVAエピトープに結合せず(Robertsonら、2000)、むしろ両方ともOVA 26:40に結合します。このことは、自然経口経路における食物抗原によるクローン選択のダイナミクスが異なることを示唆しているのかもしれない。定常食から同定された食物抗原特異的クローンは、IE適応細胞またはTh1細胞由来であったが、経口耐性クローンはTreg由来であった。したがって、経口耐性は抗原特異的なTreg反応によって特徴づけられるが、定常状態ではIE適応型CD4+ CD8α+細胞への分化を含むことができることを示すことができる。したがって、食物タンパク質は、腸上皮への適応を含む、抗原特異的な腸CD4+ T細胞における多様な表現型の結果を駆動することができます。
全体として、我々の発見は、定常状態の腸における食物反応性CD4+ T細胞の一般的な運命は上皮への適応であり、一方、炎症環境における経口耐性の維持は、Tregの流入とクローン拡大の増加に相関していることを示唆しています。我々や他の研究者は、食事に対する反応の文脈で(Sujinoら、2016)、あるいは大腸炎や感染の文脈で(Basuら、2021;Reisら、2013)、上皮適応CD4+ T細胞の重要な制御活性を実証している。さらに、IE CD4+T細胞の調節不全は、無害な腸管抗原に対する炎症反応が出現するメカニズムの一つである可能性がある。したがって、Tregに加えて上皮に適応したCD4+ T細胞を高度に制御して維持することは、食物に対する不適切な免疫応答とその後の疾患を予防するために重要であると考えられる。
材料と方法
動物
動物の世話と実験は、米国国立衛生研究所のガイドラインに合致し、ロックフェラー大学のInstitutional Animal Care and Use Committeeの承認を得た。すべてのマウスは、ロックフェラー大学の動物施設で飼育された。無菌C57BL/6Jマウスは、Sarkis Mazmanian(California Institute of Technology, Pasadena, CA, USA)から入手し、無菌アイソレーターで繁殖・維持した。SPF C57BL/6Jマウスは、糞便の単回ガベージでGFから再コロニー化し、SPF条件下で繁殖・維持させた。GFの子孫をSPF実験に使用し、GFアイソレーターでの遺伝的ドリフトを制御した。Oligo-MM12コンソーシアムは、K. McCoy (University of Calgary, Calgary, Canada)から贈られたものである。Oligo-M12の単回投与でGF C57BL/6Jブリーダーをコロニー化し、定量ポリメラーゼ連鎖反応(qPCR;下記参照)による個々の細菌メンバーの特異的増幅によってコロニー化(連続世代での全コンソーシアムの存在を含む)をモニターしました。その後、Oligo-M12マウスを繁殖させ、アイソレーターで維持し、垂直にコロニー形成された子孫をすべての実験に使用した。SellCre-ERT2マウスは、M. Nussenzweig(Merkenschlager et al., 2021)から提供され、Jackson LaboratoryからのRosa26CAG-LSL-tdTomato-WPRE(007914)マウスと交配し、SPF条件下に維持された。CD45.1 OT-II TCR-トランスジェニックマウスは、もともとTaconic Farmsから購入し、我々の施設で維持した。実験プロトコルが7週齢で開始され、11週齢でエンドポイント解析が行われたiSellTomatoを除き、ほとんどの実験でマウスは8週齢で使用された。遺伝子発現への性差の影響を避けるため、雌(飼料用)または雄(iSellTomato用)のみのマウスを使用したscRNAseqを除き、すべての実験に雄雌両方のマウスが使用された。
無菌状態およびOligo-MM12モニタリング
無菌状態は、ユニバーサル16S rRNAプライマー(fwd: 5′-ACTCCTACGGAGGCAGCAGT-3′; rev: 5′-ATTACCGCGCTGCTGGC-3′ )を用いたqPCR分析により確認した。Oligo-MM12コンソーシアムによるマウスのコロニー形成は、以前に記載されたように、各種に特異的なプライマー対を使用して、qPCRにより世代を超えて確認および監視した(Nowosadら、2020)。DNA は、ZR Fecal DNA kit (Zymo Research) を用いて、製造者の説明書に従って糞便サンプルから抽出した。定量的PCRは、Power SYBR Greenマスターミックス(Applied Biosystems)を用いて実施した。2つのテクニカルレプリケートの平均サイクル閾値(Ct)値を用いて、ユニバーサル16S rRNAプライマーをサンプル間のコントロールとして、∆∆Ct法を用いて各生物種の16SリボソームRNAの相対存在量を定量した。相対的存在量は、各生物種の16S rRNAのゲノムコピー数に応じて補正した。
実験用飼料
すべての食事実験において、ブリーダーは標準的なチャウ食で維持され、飼育ケージは食物タンパク質への早期曝露を防ぐため、子犬が1週齢になった時点でアミノ酸食に切り替えられた。その後、マウスは3週齢で実験食に離乳させ、特に指示がない限り、8週齢の終点分析までその食餌で維持された。遊離アミノ酸を含むタンパク質抗原を含まない固形飼料(5%セルロースを含むModified TestDiet 9GCV;組成の詳細は表S1にある)は、45kGy以上で照射して無菌状態を確保し、無菌状態を維持した。GF、Oligo-MM12、およびex-GF SPFチャウ食コントロールマウスには、オートクレーブ中の損失を補うために余分な栄養素で強化したオートクレーブ標準チャウ食(5K54;LabDiet)を与えた。AA+OVAについては、オバルブミン(OVA)グレードIII(A5378;Sigma-Aldrich)を飲料水中で1mg/mlで提供し、無菌のためにオートクレーブ処理した。カゼインおよびカゼイン・グルテン・大豆食は、AA食からアミノ酸を50%減らし、カゼインまたはカゼイン、グルテン、大豆タンパク質を50%均一に混合したものに変更した(9GU1、9GU2;TestDiet)。
血清生化学
離乳期からAA食またはchow食を与えた8週齢のマウスから、2時間の絶食後に血清を採取した。血清分析はIDEXX社(米国)により標準的なプロトコルで行われた。
組織学的検査
十二指腸、回腸、結腸の代表的な部分を4%パラホルムアルデヒドで固定し、標準的なプロトコールに従ってパラフィンに包埋した。5μmの切片をスライドガラスに取り付け、ヘマトキシリンとエオシン(H&E)で染色した。画像は、10×0.3/14.50mmの対物レンズを用いた倒立顕微鏡Keyence BZ-X800で、明視野照明で取得した(Keyence)。腸管病理の盲検定量評価は、確立された方法(Erbenら、2014年)に従って実施した。簡潔に言えば、各組織切片は、炎症性細胞浸潤の程度(0〜7)および上皮(0〜23)または粘膜(0〜15)構造の変化について顕微鏡的に評価された。これらのスコアの合計は、各組織について報告された病理学的スコアの合計を表し、40が最大スコアで、スコア<10は炎症が最小から軽度の正常組織を示す。組織メトリクスのさらなる定量化は、ImageJで実施した。
腸管T細胞の単離
上皮内リンパ球およびLPリンパ球を、以前に記載されたように単離した(Bilateら、2016年;Reisら、2013年)。簡潔に言えば、小腸または大腸(盲腸および結腸)を採取し、PBSおよび1mMジチオスレイトール(DTT)、次いで30mM EDTAで洗浄した。DTTおよびEDTA洗浄の上清から上皮内細胞を回収し、Percollを用いた勾配遠心分離により単核球を分離した。LPリンパ球は、組織をコラゲナーゼ消化した後に得た。
抗体およびフローサイトメトリー解析
蛍光色素結合抗体は、BD Biosciences、BioLegend、Ebioscience(Thermo Fisher Scientific)、またはR&D Biosciencesから購入した。以下のクローンを使用した:抗CD4 RM4-5; 抗CD8α 53-6.7; 抗CD8β YTS 156.7.7; 抗CD11b M1/70; 抗CD11c N418; 抗CD44 IM7; 抗CD45 30-F11; 抗CD45・1 A20; 抗CD62L MEL-14, G8.8; 抗CD69 H1. 2F3; 抗CD103 2E7; 抗CD117(cKit) 2B8; 抗F480 BM8; 抗FceR1 MAR-1; 抗Foxp3 FJK-16 s; 抗グランザイムB GB11; 抗IL-4 11B11, 抗IL-17A TC11-18H10; 抗Ly6C AL-21、抗Ly6G RB6-8C5、抗Nrp1 BAF566、抗Rorγt Q31-378、抗Siglec F E50-2440、抗TCRβ H57-597、抗TCRγδ GL3、抗TCR Vα2 B20。 1. Live/dead fixable dye Aqua(Thermo Fisher Scientific)は、製造者の指示に従い使用した。Foxp3の核内染色およびGranzyme Bの細胞内染色は、Foxp3 Mouse Regulatory T Cell Staining Kitを用い、キット説明書(eBioscience)に従って実施した。IL-4およびIL17-A産生の解析のために、細胞を100 ng/ml phorbol 12-myristate 13-acetate (PMA, Sigma-Aldrich) 200 ng/ml ionomycin (Sigma-Aldrich), Monensin (2 mM, BD Biosciences) を含むGolgi stop solutionと37℃で4時間インキュベートした。細胞内サイトカイン染色は、Fix/Perm buffer(BD Biosciences社製)で固定・透過処理後、Perm/Wash bufferでキットの説明書に従って実施した。フローサイトメトリーデータは、LSR-IIまたはSymphonyフローサイトメーター(Becton Dickinson)で取得し、FlowJoソフトウェアパッケージ(Tri-Star)を使用して解析した。細胞選別実験のために、リンパ球はFACS Aria II装置で図解に示すように選別された。AccuCheck Counting Beads(Thermo Fisher Scientific)は、絶対細胞数のカウントに使用した。フローサイトメトリー解析では、以下のゲーティング戦略を用いて細胞集団を同定した。T細胞:単一の、生きた、リンパ球(FSC、SSC、および生死確定色素Aqua染色に基づく)、CD45+、TCRgδ+(gδT細胞について)、またはTCRβ+(他のすべてのT細胞について);CD4+T細胞: CD4+、CD8β-。CD8αβ+ T細胞: CD4-, CD8β+. CD8αα+ T細胞: CD4-、CD8β-、CD8α+。骨髄系細胞:シングル、ライブ、CD45+;好酸球: シグレックF+、CD11b+、マスト細胞:cKit+、FCer1+、マクロファージ: CD11b+, CD11c+, F480+; 単球: CD11b+ CD11c- Ly6c+;好中球: Ly6G+ CD11b+。
OTIIトランスファー実験
CD45.1 OTII TCRトランスジェニックマウスの脾臓およびリンパ節からナイーブCD4 T細胞を、CD8α、CD25、CD11c、CD11b、TER-119、NK1.1、B220に対するバイオチン化抗体および抗ビオチンMACSビーズ(Miltenyi Biotec)を用いたネガティブセレクションにより分離した。イソフルランガス麻酔下でCD45.2宿主に眼窩後注射により1×106個の細胞を移植した。宿主マウスには、通常の飲料水または1 mg/ml OVA grade III (A5378; Sigma-Aldrich)を添加した飲料水を与えた。48時間後、腸間膜リンパ節を採取し、CD45.1+ TCRVα2+ CD4+ OTII細胞をCD69の発現により活性化について分析した。
タモキシフェン投与
タモキシフェン(Sigma-Aldrich)をコーン油(Sigma-Aldrich)および10%エタノールに溶解し、37℃で30分-1時間振とうし、50mg/mlの2用量(5mg/用量)を治療プロトコル開始の3日前および1日前に、経口ガベージによりマウスに投与した.
OVA/コレラ毒素アレルギーモデル
OVA grade III (A5378; Sigma-Aldrich)を飲料水中に0.1%で供給し、3日間オートクレーブで無菌化し(摂食、耐性)、またはしない(アレルギー)ことで耐性を初期化した。その後、すべてのマウスに通常の飲料水を1週間提供した。0.2M重炭酸ナトリウム中の1mgのOVA(摂食)または0.2M重炭酸ナトリウム中の1mgのOVA+20μgコレラ毒素(100B;リストバイオロジー)(耐性、アレルギー)を3週間にわたり週1回投与し、最終投与2日後に終点分析を行った。血清は、エンドポイント解析の1日前にELISA用に採取された。アナフィラキシー実験については、以下の修正を除き、上記のプロトコルに従った: OVAまたはOVA/CTを週1回、4週間投与し、その後、最終投与から7日後にチャレンジした。移植可能な電子温度プローブ(Avidity IPTT-300)は、チャレンジの1日前にs.c.注入された。マウスに5mgのOVAをi.p.投与し、10分ごとに50分間体温を測定した。アナフィラキシー実験に使用したマウスは、下流のシークエンス解析には使用しなかった。
ELISA
リポカリン-2は、Chassaingら(2012)によって記載されたように、Lcn-2 ELISAキット(R&D Biosciences)を用いて分析した。IgEおよびOVA特異的IgG1 ELISAは、以前に記載されたように行った(Atarashi et al.、2011)。
16S rRNAの塩基配列決定
腸内容物は、終点分析時に全小腸または盲腸から新鮮な状態で採取した。母体の影響をコントロールするために、すべての16S実験にリターメイトコントロールを使用した。ケージ効果をコントロールするため、実験グループごとに少なくとも4つの異なるケージから試料を採取した。サンプルは、Earth Microbiome Project (Caporaso et al., 2011)の16S Illumina Ampliconプロトコルに従って16S rRNA配列決定用に準備した。ライブラリーは、15% PhiXスパイクを使用してMiseq 2 × 150で配列決定した。配列処理と解析はRで行った。簡単に言うと、アンプリコン配列バリアント(ASV)へのリードアセンブリとSilvaデータベースでの分類学的割り当てをDada2(v.1.2.6)を使って行った。少なくとも20%のサンプルで10回以上見られない分類群は、解析から除外した。ASVの定量化と解析はPhyloseq (v1.42; McMurdie and Holmes, 2013)を用いて実施した。
scRNAseqライブラリー調製
小腸上皮またはLPから単離したリンパ球を上記のように単離し、TotalSeqC Hashtag(BioLegend)細胞表面抗体でインデックス化し、より深い多重化のためにサンプルあたり2つのバーコードを使用した。全CD4+ T細胞をソートし、プールし、直ちにChromium Controller(10×Genomics) にロードしました。SellTomato では、選別された Tomato+ と Tomato-(CD62L-、CD4+ T)細胞は、約1:3の比率でプールされた。5′遺伝子発現、VDJ、Feature Barcodeライブラリーは、Chromium Single Cell 5′ v2 Reagent Kit(10×Genomics)を用いて、The Rockefeller UniversityのGenomics coreで製造者のプロトコルに従って準備した。ライブラリーはIllumina NextSeq500またはNovaSeq 6000で配列決定した。ハッシュタグインデックスは、シーケンスデータの多重化を解除し、遺伝子バーコードマトリックスを生成するために使用した。
scRNAseqおよびシングルセルTCRseqライブラリーのデータ処理
当社の10Xライブラリからの生の.FASTQファイルは、10X Genomicsの構築済みマウスリファレンス(v3.0.0 mm10;ダイエット用)またはAi9Tomato配列プラスミドを人工染色体として含みAi9TomatoおよびSTOP配列が特徴として注釈されたカスタマイズマウスゲノム(mm10)(SellTomato用)を用いてCellranger count (v6.2.0) で処理しました。解析はR 4.2.2で行った。品質管理は、ミトコンドリア固有分子識別子(UMI)含有量が高い(抗原除去食ライブラリでは10%以上、SellTomatoでは5%以上)細胞を除去することで実施した。TracまたはCd4を発現していない細胞は、解析から除外した。tdTomatoとStopのUMIカウントを正規化し(Tomato+: tdTomato > Stop; Tomatoo-: tdTomato < Stop)、tdTomato = Stopの曖昧な細胞を除外して、シーケンス後にTomato+とTomato-細胞を定義しました。UMIカウントの行列は、Seurat (v41.-4.3.)パッケージ(Hafemeister and Satija, 2019)のSCTransform関数を通じて利用できる、負の二項分布の回帰モデルを適用して正規化した。上位3,000個の可変遺伝子をまず、スケーリングされたデータを用いてPCAによる次元削減に使用した。最初の30個の主成分はさらに、Manifold Approximation and Projection(UMAP)および細胞クラスタリングを用いた可視化に使用された(Hafemeister and Satija, 2019; Stuart et al, 2019)。TCRコンティグとアノテーションは、10X GenomicsのCellranger vdjワークフローとprebuildマウスリファレンス(v3.1.0 mm10)で実施しました。コンティグのフィルタリング、クロノタイプコーリング、および下流のTCR解析は、scRepertoire (v1.5.2; Borcherding et al., 2020)を使用して実施した。さらなる処理、統計解析、および可視化は、ggplot2 (v3.4.1; Wickham, 2016) および rstatix (v0.7.2) を使用して実行した。
シグネチャースコア算出
IEシグネチャースコアは、UCell(v2.2; Andreatta and Carmona, 2021)を用いて、以下の遺伝子を入力として計算しました: Nkg7、Ccl5、Cd160、Itgae、Gzma、Gzmb、Cd7、Prf1、Lag3、およびCd8a。
3Dボルケーノ
3Dボルケーノプロットは、volcano3D (v1.2-2.0.8; Lewis et al., 2019)を使用して生成した。簡単に言うと、3つのグループ間で差次的に発現した遺伝子は、任意のグループの細胞の少なくとも10%で発現した遺伝子のみを考慮して、FDR調整クラスカル-ウォリス検定(P-adj < 0.05)により特定した。各グループの発現量は、さらにFDR調整したWilcoxon順位和検定(P-adj <0.05, Log2FC > 0.5)により決定した。プロット上の各ポイントは、遺伝子が有意にアップレギュレートされたグループによって色分けされ、有意でない遺伝子は色分けされていない。各遺伝子の原点からの距離は、3つのグループ間のクラスカル・ウォリス比較から得られた-log10 P値に基づいて計算されたzスコアを表し、原点から遠い遺伝子ほど高い有意性を持つことを示している。ある条件における遺伝子のアップレギュレーションの程度は、条件ラベルのついた軸に対するプロット上の角度で示される。フォールドチェンジを除くすべての計算には、スケールされた遺伝子発現データが使用された。各プロットには注目すべき遺伝子を選択し、ラベルを付けている。
サルコスプロット
TCR共有(クローンオーバーラップ)はCircosを使用して可視化し、円形プロットの美学を作成した(Krzywinski et al.、2009)。各セグメントは図に示すようにマウスを示し、バンドは対になったTCRαとTCRβのアミノ酸配列に基づくクローン共有を表している。NKT細胞および対になるTCRαとTCRβの配列がない細胞は、解析に考慮しなかった。
統計解析
統計解析は、GraphPad Prism v.9を使用して実施した。フローサイトメトリー解析は、FlowJoソフトウェアを用いて実施した。グラフのデータは平均値±SEMを示し、P値<0.05は有意とみなした。レパートリーの多様性は、ペアCDR3全体の累積50%を占める支配的なクローンの割合としてRで計算したDiversity 50(D50)により分析した。グラフにはGraphPadPrism v.9を、図の組み立てと編集にはAdobe Illustrator 2021を使用した。
TCRハイブリドーマの作製
scRNAseqデータセットから選択したTCRを合成し(P2Aで連結したTCRαおよびTCRβ)、pMSCV-mCD4ベクター(Twist Bioscience)にクローニングした。レトロウイルス産生にはPhoenix-Eco細胞(CRL-3214;Addgene)を使用した。Phoenixは、10cmの培養皿に2mlの0.01%ポリ-L-リジン(A-005-M;Sigma-Aldrich)をプレコートしたDMEM中で60〜80%コンフルエンスまで成長させ、8ml 0. 1%ゼラチン(1040700500;Sigma-Aldrich)、そして5μg pMSCV-mCD4-TCRプラスミド、2μg pEco(12371;Addgene)、72μl 1mg/mlポリエチレンアミン(23966;Polysciences)、600μl DMEMを含む混合物でトランスフェクションした。培地は24時間で交換し、48時間で収穫してウイルス導入に使用した。NFAT-GFPハイブリドーマ細胞を、15μgのレトロネクチン(T100A;Takara)でプレコートした6ウェルプレートにおいて、5μg/mlのポリブレン(TR-1003-G;Sigma-Aldrich)を用いたウイルス上清中に1×106/mlプレーティングした。プレートは、2,500rpm 32℃、90分間の遠心分離によりスピンフェクションした。次に、細胞を、TCR+CD3+細胞のFACS選択の前に、IMDM+10%FBS+Pen/Strep/L-グルタミン+50μM β-me+1mM ピルビン酸ナトリウムで2〜7日間培養した。
OVA特異性のためのTCR試験
樹状細胞をマウス脾臓(130-092-465;Miltenyi Biotec)から採取し、96ウェルUボトムプレートで37℃で4時間、50μg OVA grade III(A5378;Sigma-Aldrich)と5×105/ml共培養して抗原を負荷するか、ネガティブコントロールのために負荷しないままにした。2.5×104個のTCR発現NFAT-GFP細胞を各ウェルに添加し、一晩インキュベートした。ポジティブコントロールとして、0.2μlの抗CD3(553057;BD)を、NFATと負荷していないDCのあるウェルに添加した。TCR刺激に対するNFAT-GFPの応答は、フローサイトメトリーで測定した。エピトープ特異性を試験するために、OVAの長さを5-aaシフトしてカバーする粗15-aaペプチドを合成し(LifeTein)、ウェルあたり1μMで上記のようにアッセイで使用した。TCR刺激は、培養上清のIL-2 ELISAで測定した(555148;BD)。
図の作成
図はPrismまたはRで作成し、Adobe Illustratorでコンパイル・整形した。グラフィカルアブストラクトはBioRender(グラフィカルライセンスLO25BBHSCS)を用いて作成した。
オンライン補足資料
図S1は、体重、糞便リポカリン、血清栄養バイオマーカー、腸の長さ、腸の組織学、および腸のミエロイド細胞の定量化を含むAAダイエットマウスの健康状態の検証を示す図である。図S2は、図1および2を支持する、食餌タンパク質に応答する腸CD4+T細胞蓄積の追加分析および16S rRNA配列決定;および図S3は、図2および3を支持する、AA、AA+OVA、またはチョウ食を与えたGFおよびOligoMM12マウスの追加scRNAseq分析;および図S4、A〜Dは、iSellTomatoマウスにおけるOVA摂食、耐性およびアレルギーモデルに関する追加の組織学およびフローサイトメトリ特性化。図S4、E-Hは、iSellTomatoマウスのscRNAseq解析の裏付けを示す。図S5 A-Gは、OVA摂食、耐性、およびアレルギーにおけるiSellTomatoマウスの追加のscRNAseq解析を示す。図S5 Hは、候補TCRに対するOVA重複ペプチド反応性試験を示す。表S1は、AA食の組成を示す。表S2は、AA飼料またはチョウ飼料を与えたSPFマウスの16S rRNA配列決定結果を示す。表S3は、AA、AA+OVA、またはチャウ食を与えたGFマウスおよびOligoMM12マウスの全CD4+T細胞UMAPクラスタにわたる差次的発現遺伝子を示す。表S4は、AA、AA+OVA、またはチャウ食マウスからのIE CD4+T細胞間の三元差分遺伝子発現試験の結果を示す。表S5は、AA、AA+OVA、またはチャウ食を与えたGFマウスおよびOligoMM12マウスからのTreg UMAPクラスター間で差次的に発現した遺伝子を示す。表S6は、AA、AA+OVA、またはチョウ食マウスのTreg間の三元差分遺伝子発現試験の結果を示す。表S7は、OVA摂食、耐性、およびアレルギーにおけるiSellTomatoマウスからの全CD4+T細胞UMAPクラスターにわたる差次的発現遺伝子を示す。表S8は、OVA摂食、耐性、およびアレルギーにおけるiSellTomatoマウスからのTomato-IE CD4+T細胞間の3方向差分遺伝子発現試験からの結果を示す。表S9は、OVA摂食、耐性、およびアレルギーにおけるiSellTomatoマウスからのTomato+ IE CD4+T細胞間の三元差分遺伝子発現試験からの結果を示す。表S10は、OVA摂食、耐性、およびアレルギーにおけるiSellTomatoマウスからのTomato+ LP CD4+T細胞間の三元差分遺伝子発現試験による結果を示す。表S11は、OVA摂食、耐性、およびアレルギーにおけるiSellTomatoマウスからのTreg UMAPクラスター間で差次的に発現した遺伝子を示す。表S12は、OVA特異性について試験したscRNAseqデータセットで同定されたTCRに関する補足情報を示す。
データの利用可能性
AA、AA+OVA、またはチャウ食を与えたGFまたはOligo-M12マウス、およびOVA摂食、耐性、またはアレルギープロトコルのSPF iSellTomatoマウスの腸内CD4+T細胞のscRNAseqデータは、Gene Expression Omnibusアクセッション番号GSE231351で一般公開されています。その他のデータは、公開された論文およびオンライン補足資料で入手できます。
謝辞
Mucida Labのメンバーおよびロックフェラー大学の職員の皆様の継続的なご協力に感謝します。特に、マウスのメンテナンスにA. RogozおよびS. Gonzalez、配列決定にRU Genomics core、組織学的準備にTri-I Laboratory of Comparative Pathology、ソーティングに K. Gordon, K. Chhosphel, J.P. Truman、図の作成に B. Reis、原稿にCritical Readingをしていただいた A. Bilate, G. Donaldson および R. Parsa に感謝します。また、VictoraとLafailleの研究室には、実りある議論をさせていただいたことに感謝する。
この研究は、Howard Hughes Medical Instituteの助成金R01DK093674、R01DK113375、R21AI144827、Food Allergy FARE/FASI Consortiumから支援を受けた。
著者の貢献 A. Lockhartは、実験の開始、設計、実施、分析を行い、原稿を執筆した。A. Reed、C. Herman、M.C. Campos Canessoは、実験の設計と実施を行った。T. Rezende de Castroは解析を行った。D. Mucidaは、研究の構想、開始、設計、監督を行い、原稿を執筆した。著者全員が原稿と図表を修正・編集した。
参考文献
アバディ
,
V.
,
V.
ディセポロ
を、そして
B.
ジャブリ
.
2012
.
セリアック病免疫病理における上皮内リンパ球の存在
.
Semin. Immunopathol.
34
:
551
566
.
https://doi.org/10.1007/s00281-012-0316-x
Google Scholar
クロスフィルム
PubMed
アバディ
,
V.
,
S.M.
キム
,
T.
Lejeune
,
B.A.
パランスキー
,
J.D.
アーネスト
,
O.
タステット
,
J.
ヴォワジーヌ
,
V.
ディセポロ
,
E.V.
マリエッタ
,
M.B.F.
ハワッシュ
, et al.
2020
.
IL-15、グルテン、HLA-DQ8がセリアック病の組織破壊を促進する
.
ネイチャー
.
578
:
600
604
.
https://doi.org/10.1038/s41586-020-2003-8
Google Scholar
クロスフィルム
パブコメ
アンドレッタ
,
M.
アンドレッタ , そして
S.J.
カルモナ
.
2021
.
UCell: ロバストでスケーラブルなシングルセル遺伝子シグネチャースコアリング
.
Comput. Struct. バイオテクノロジー(Biotechnol. J.
19
:
3796
3798
.
https://doi.org/10.1016/j.csbj.2021.06.043
Google Scholar
クロスフィルム
パブコメ
アタラシ
,
K.
,
T.
田之上
,
T.
志摩
,
A.
今岡
,
T.
桑原
,
Y.
百瀬
,
G.
Cheng
,
S.
山崎
,
T.
斉藤
,
Y.
大場
他
2011
.
土着のクロストリジウム種による大腸制御性T細胞の誘導
.
サイエンス
.
331
:
337
341
.
https://doi.org/10.1126/science.1198469
Google Scholar
クロスフィルム
パブコメ
芭蕉
,
J.
,
B.S.
Reis
,
S.
ペリ
,
J.
Zha
,
X.
Hua
,
L.
Ge
,
K.
Ferchen
,
E.
ニコラ
,
P.
Czyzewicz
,
K.Q.
Cai
他
2021
.
成熟CD4+ T細胞のアイデンティティと機能の維持におけるThPOK自己調節ループの重要な役割
.
Nat. Immunol.
22
:
969
982
.
https://doi.org/10.1038/s41590-021-00980-8
Google Scholar
クロスフィルム
パブコメ
ビラート
,
A.M.
,
D.
ブスバイン
,
L.
メサン
,
M.
アグデロ
,
J.
リューベ
,
A.
クラッツァート
,
S.K.
ドウガン
,
G.D.
ビクトラ
と
H.L.
Ploegh
.
2016
.
クローン性T細胞前駆体からの制御性T細胞および上皮内T細胞の組織特異的な出現
.
サイ・イムノル.
1
:eaaf7471.
https://doi.org/10.1126/sciimmunol.aaf7471
Google Scholar
ビラート
,
A.M.
,
M.
ロンドン
,
T.B.R.
カストロ
,
L.
メシン
,
J.
Bortolatto
,
S.
コングトーン
,
A.
ハルナゲル
,
G.D.
ビクトラ
を、そして
D.
ムシダ
.
2020
.
腸管CD4+上皮内リンパ球の分化にはT細胞受容体が必要であるが、維持には必要ない
.
免疫力
.
53
:
1001
1014.e20
.
https://doi.org/10.1016/j.immuni.2020.09.003
Google Scholar
クロスフィルム
パブコメ
ボルチェリング
,
N.
,
エヌエル.
ボルマン
を、そして
G.
クラウス
.
2020
.
scRepertoire: 単一細胞免疫受容体解析のためのRベースのツールキット
.
F1000 Res.
9
:
47
.
https://doi.org/10.12688/f1000research.22139.1
Google Scholar
クロスレフ
ブスバイン
,
D.
,
L.I.
フィッシュ
,
M.
ロンドン
,
P.
バグチャンダニ
,
T.B.
レゼンデ・デ・カストロ
,
M.
ミーミー
,
S.
Olesen
,
B.S.
Reis
,
D.
ヴァンインスベルヘ
,
J.
ボートラット
他
2022
.
保存されたバクテロイデス抗原は抗炎症性腸管Tリンパ球を誘導する
.
サイエンス
.
377
:
660
666
.
https://doi.org/10.1126/science.abg5645
Google Scholar
クロスフィルム
パブコメ
ブージア
,
R.
,
R.
ヒンターライトナー
,
J.J.
ブラウン
,
J.E.
ステンセル・ベーレンワルド
,
M.
イキズラー
,
T.
Mayassi
,
M.
マイゼル
,
S.M.
キム
,
V.
ディセポロ
,
A.J.
Pruijssers
, et al.
2017
.
レオウイルス感染が食事性抗原に対する炎症反応とセリアック病の発症の引き金となる
.
サイエンス
.
356
:
44
50
.
https://doi.org/10.1126/science.aah5298
Google Scholar
クロスフィルム
PubMed
ブリュギルー(Brugiroux
,
S.
,
M.
ボイトラー
,
C.
プファン
,
D.
ガルゼッティ
,
H.J.
ルシェウェイ
,
D.
リング
,
M.
Diehl
,
S.
Herp
,
Y.
Lötscher
,
S.
Hussain
, et al.
2016
.
Salmonella enterica serovar Typhimuriumに対するコロニー形成抵抗性を付与する定義されたマウス微生物叢のゲノムガイド付きデザイン
.
Nat. Microbiol.
2
:
16215
.
https://doi.org/10.1038/nmicrobiol.2016.215
Google Scholar
クロスフィルム
パブコメ
カポラソ
,
J.G.
,
C.L.
ラウバー
,
W.A.
ウォルターズ
,
D.
ベルグ=リオンズ
,
C.A.
ロズポーン
,
P.J.
ターンボー
,
N.
フィーア
を、そして
R.
ナイト
.
2011
.
サンプルあたり数百万配列の深さでの16S rRNA多様性のグローバルパターン
.
Proc. Natl. Acad. Sci.USA
.
108
:
4516
4522
.
https://doi.org/10.1073/pnas.1000080107
Google Scholar
クロスレフ
チェネレンティ
,
M.
,
M.
Saillard
,
P.
ロメロ
を、そして
C.
ジャンドゥス
.
2022
.
細胞傷害性CD4 T細胞の時代
.
Front. Immunol.
13
:
867189
.
https://doi.org/10.3389/fimmu.2022.867189
Google Scholar
クロスフィルム
パブコメ
シャッセイング
,
B.
,
G.
スリニヴァーサン
,
M.A.
デルガド
,
A.N.
ヤング
,
A.T.
ゲヴィルツ
を、そして
M.
ヴィジャイ・クマール
.
2012
.
腸内炎症の非侵襲的バイオマーカーである便中リポカリン2の高感度かつ広範な動的特性
.
PLoS One
.
7
:e44328.
https://doi.org/10.1371/journal.pone.0044328
Google Scholar
シェルトル
,
H.
を、そして
F.
ランボレズ
.
2008
.
CD8ααのTCRコアレセプター機能を疑う
.
イミュニティ
.
28
:
149
159
.
https://doi.org/10.1016/j.immuni.2008.01.005
Google Scholar
クロスフィルム
パブコメ
チョウ
,
C.
,
X.
Zhang
,
C.
クリシュナ
,
B.G.
ニクソン
,
S.
ダディ
,
K.J.
キャピストラーノ
,
E.R.
カンスラー
,
M.
スティール
,
J.
ハン
,
A.
Shyu
をはじめとする。
2022
.
自己反応性自然様T細胞介在型がん免疫のプログラム
.
ネイチャー
.
605
:
139
145
.
https://doi.org/10.1038/s41586-022-04632-1
Google Scholar
クロスフィルム
パブコメ
コステス
,
L.M.M.
,
D.J.
リンデンベルグ=コートルヴェ
,
L.A.
ヴァン・ベルケル
,
S.
ヴィーンベルゲン
,
H.R.C.
ラートギープ
,
Y.
サイモンズ=オステルホイス
,
D.H.
ヴァン・ハフテン
,
J.J.
カーリッチ
,
J.C.
エッシャー
,
M.
Groeneweg
, et al.
2019
.
IL-10シグナルは、グルテン依存性の上皮内CD4+細胞傷害性Tリンパ球の浸潤と小腸の上皮障害を予防する
.
Mucosal Immunol.
12
:
479
490
.
https://doi.org/10.1038/s41385-018-0118-0
Google Scholar
クロスフィルム
パブコメ
デニング
,
T.L.
,
S.W.
グレンジャー
,
D.
ムシダ
,
R.
グラディ
,
G.
Leclercq
,
W.
チャン(Zhang
,
K.
ハニー
,
J.P.
Rasmussen
,
H.
Cheroutre
,
A.Y.
ルデンスキー
を、そして
M.
クローネンベルク
.
2007
.
マウスTCRαβ+CD8αα上皮内リンパ球は、抗原反応性をダウンレギュレートし、免疫応答を抑制する遺伝子を発現する
.
J. Immunol.
178
:
4230
4239
.
https://doi.org/10.4049/jimmunol.178.7.4230
Google Scholar
クロスフィルム
パブコメ
エルベン
,
U.
,
C.
Loddenkemper
,
K.
ドアーフェル
,
S.
シュピーカーマン
,
D.
ハラー
,
M.M.
ハイメサット
,
M.
ツァイツ
,
B.
ジークムント
を、そして
A.A.
キュール
.
2014
.
マウスモデルにおける腸管炎症の組織形態学的評価ガイド
.
Int. J. Clin. Exp. Pathol.
7
:
4557
4576
.
Google Scholar
パブコメ
フィナ
,
D.
,
M.
サーラ
,
R.
カルーソ
,
G.
デル・ベッキオ・ブランコ
,
F.
パローネ
,
T.T.
MacDonald
を、そして
G.
モンテレオーネ
.
2008
.
セリアック病における粘膜Tヘルパー細胞1型反応に寄与するインターロイキン21
.
腸
.
57
:
887
892
.
https://doi.org/10.1136/gut.2007.129882
Google Scholar
クロスフィルム
パブコメ
ガーサイド
,
P.
,
M.
スチール
,
F.Y.
リュー
と
A.M.
モワット
.
1995
.
CD4+ではなくCD8+T細胞が経口耐性の誘導に必要であること
.
Int. Immunol.
7
:
501
504
.
https://doi.org/10.1093/intimm/7.3.501
Google Scholar
クロスフィルム
パブコメ
グロスマン
,
W.J.
,
J.W.
バーブスキー
,
W.
バーシェット
,
M.
コロンナ
,
J.P.
アトキンソン
を、そして
T.J.
レイ
.
2004
.
ヒトT制御細胞はパーフォリン経路を利用して自己の標的細胞死を引き起こすことができる
.
免疫力
.
21
:
589
601
.
https://doi.org/10.1016/j.immuni.2004.09.002
Google Scholar
クロスフィルム
パブコメ
ハディス
,
U.
,
B.
ワール
,
O.
シュルツ
,
M.
ハルトケ=ウォレンスキー
,
A.
シッパーズ
,
N.
ワグナー
,
W.
ミュラー
,
T.
スパルヴァッサー
,
R.
フェルスター
を、そして
O.
パブスト
.
2011
.
腸管寛容には、腸管ホーミングとFoxP3+制御性T細胞のlamina propriaでの拡大が必要である
.
免疫力
.
34
:
237
246
.
https://doi.org/10.1016/j.immuni.2011.01.016
Google Scholar
クロスフィルム
パブコメ
ハーフェマイスター
,
C.
を、そして
R.
サティヤ
.
2019
.
正則化負の二項回帰を用いた単一細胞RNA-seqデータの正規化と分散安定化
.
ゲノム・バイオル.
20
:
296
.
https://doi.org/10.1186/s13059-019-1874-1
Google Scholar
クロスフィルム
パブコメ
ホン
,
S.W.
,
P.D.
Krueger
,
K.C.
オサム
,
T.
Dileepan
,
A.
ハーマン
,
D.L.
ミューラー
と
M.K.
ジェンキンス
.
2022
.
食物の免疫寛容はCD4+ T細胞機能不全の層によって媒介される
.
自然界
.
607
:
762
768
.
https://doi.org/10.1038/s41586-022-04916-6
Google Scholar
クロスフィルム
パブコメ
伊勢
,
W.
,
M.
小山
,
K.M.
ナッチ
,
H.M.
リー
,
A.
スーリ
,
E.R.
ウナヌエ
,
T.L.
マーフィー
を、そして
K.M.
マーフィー
.
2010
.
CTLA-4は、細胞内在性および細胞外性メカニズムによって自己抗原特異的T細胞の病原性を抑制する
.
Nat. Immunol.
11
:
129
135
.
https://doi.org/10.1038/ni.1835
Google Scholar
クロスフィルム
パブコメ
ヒメネス-サイス
,
R.
,
D.K.
Chu
,
T.S.
マンドゥール
,
T.D.
ウォーカー
,
M.E.
ゴードン
,
R.
チャウダリー
,
J.
ケーニッヒ
,
S.
サリバ
,
H.J.
ガリポー
,
A.
アトレー
, et al.
2017
.
食物アレルギーにおいて、生涯記憶応答が体液性TH2免疫とアナフィラキシーを永続させる
. J
Allergy Clin Immunol.
140
:
1604
1615 e1605
.
https://doi.org/10.1016/j.jaci.2017.01.018
Google Scholar
クロスフィルム
ヨセフォビッチ
,
S.Z.
,
R.E.
ニエック
,
H.Y.
キム
,
P.
トレウティング
,
T.
知念
,
Y.
Zheng
,
D.T.
梅津
と
A.Y.
ルデンスキー
.
2012
.
粘膜外に生成された制御性T細胞は粘膜TH2炎症を制御する
.
ネイチャー
.
482
:
395
399
.
https://doi.org/10.1038/nature10772
Google Scholar
クロスフィルム
パブコメ
キム
,
K.S.
,
S.W.
ホン
,
D.
ハン
,
J.
Yi
,
J.
Jung
,
B.G.
ヤン
,
J.Y.
リー
,
M.
リー
と
C.D.
Surh
.
2016
.
食餌性抗原は小腸の制御性T細胞を誘導することで粘膜免疫を制限する
.
サイエンス
.
351
:
858
863
.
https://doi.org/10.1126/science.aac5560
Google Scholar
クロスフィルム
パブコメ
クシヴィンスキー
,
M.
,
J.
シャイン
I.
ビロル
,
J.
コナーズ
,
R.
Gascoyne
,
D.
ホースマン
,
S.J.
ジョーンズ
ジョーンズ、および
M.A.
マーラ
.
2009
.
サーカス: 比較ゲノミクスのための情報美学
.
Genome Res.
19
:
1639
1645
.
https://doi.org/10.1101/gr.092759.109
Google Scholar
クロスフィルム
パブコメ
ルイス
,
M.J.
,
M.R.
バーンズ
,
K.
ブリッヒ
,
K.
ゴールドマン
,
S.
ラナ
,
J.A.
ハックニー
,
N.
ラマモオルティ
,
C.R.
ジョン
,
D.S.
ワトソン
,
S.K.
クマーフェルド
, et al.
2019
.
早期関節リウマチの分子ポートレートは、臨床と治療反応の表現型を特定する
.
セルレップ.
28
:
2455
2470.e5
.
https://doi.org/10.1016/j.celrep.2019.07.091
Google Scholar
クロスフィルム
パブコメ
ロンドン
,
M.
,
A.M.
ビラート
,
T.B.R.
カストロ
,
T.
スジノ
を、そして
D.
ムシダ
.
2021
.
上皮内リンパ球プログラムの段階的なクロマチンおよび転写の獲得
.
Nat. Immunol.
22
:
449
459
.
https://doi.org/10.1038/s41590-021-00883-8
Google Scholar
クロスフィルム
パブコメ
マクマーディ
,
P.J.
とは
S.
ホームズ
.
2013
.
phyloseq:マイクロバイオームセンサスデータの再現性の高いインタラクティブな解析とグラフィックスのためのRパッケージ
.
PLoS One
.
8
:e61217.
https://doi.org/10.1371/journal.pone.0061217
Google Scholar
メルヘンシュラガー
,
J.
,
S.
フィンキン
,
V.
ラモス
,
J.
クラフト
,
M.
チポッラ
,
C.R.
ノウォサド
,
H.
ハルトウィガー
,
W.
チャン(Zhang
,
P.D.B.
オリナレス
,
A.
ガズィーミャン
, et al.
2021
.
胚中心反応におけるTFH選択の動的な制御
.
ネイチャー
.
591
:
458
463
.
https://doi.org/10.1038/s41586-021-03187-x
Google Scholar
クロスフィルム
パブコメ
ムシダ
,
D.
,
M.M.
フセイン
S.
室井
,
F.
van Wijk
,
R.
Shinnakasu
,
Y.
直江
,
B.S.
ライズ
,
Y.
ホアン
,
F.
ランボレズ
,
M.
ドカティ
をはじめとする。
2013
.
成熟CD4⁺ヘルパーT細胞の転写リプログラミングは、異なるMHCクラスII制限の細胞傷害性Tリンパ球を生成する
.
Nat. Immunol.
14
:
281
289
.
https://doi.org/10.1038/ni.2523
Google Scholar
クロスフィルム
パブコメ
ムシダ
,
D.
,
N.
クチュクチュヒッツェ
,
A.
エラゾ
,
M.
ルッソ
,
J.J.
ラファイユ
と
M.A.
キュロット・ドゥ・ラファイユ
.
2005
.
自然界に存在するTregが存在しない場合の経口耐性
.
J. Clin. Invest.
115
:
1923
1933
.
https://doi.org/10.1172/JCI24487
グーグル・スカラー
クロスフィルム
パブコメ
ノウォサド
,
C.R.
,
L.
メシン
,
T.B.R.
カストロ
,
C.
ヴィヒマン
,
G.P.
Donaldson
,
T.
荒木
,
A.
Schiepers
,
A.A.K.
ロックハート
,
A.M.
ビレイト
,
D.
ムシダ
を、そして
G.D.
ビクトラ
.
2020
.
腸管胚中心におけるB細胞選択の調整可能なダイナミクス
.
自然界
.
588
:
321
326
.
https://doi.org/10.1038/s41586-020-2865-9
Google Scholar
クロスフィルム
PubMed
パブスト
,
O.
(パブスト) 、および
A.M.
モワット
.
2012
.
食品タンパク質に対する経口耐性
.
Mucosal Immunol.
5
:
232
239
.
https://doi.org/10.1038/mi.2012.4
Google Scholar
クロスフィルム
パブコメ
パルサ
,
R.
,
M.
ロンドン
,
T.B.
レゼンデ・デ・カストロ
,
B.
レイズ
,
J.
ビュサン・デ・アモリー
,
J.G.
スミス
を、そして
D.
ムシダ
.
2022
.
新規に採用された上皮内Ly6A+CCR9+CD4+T細胞は腸管ウイルス感染から守る
.
免疫力
.
55
:
1234
1249.e6
.
https://doi.org/10.1016/j.immuni.2022.05.001
Google Scholar
クロスフィルム
パブコメ
ライズ
,
B.S.
,
A.
ロゴス
,
F.A.
コスタ=ピント
,
I.
谷内
を、そして
D.
ムシダ
.
2013
.
転写因子Runx3とThPOKの相互発現が腸管CD4⁺T細胞免疫を制御する
.
Nat. Immunol.
14
:
271
280
.
https://doi.org/10.1038/ni.2518
Google Scholar
クロスフィルム
パブコメ
ロバートソン
,
J.M.
,
P.E.
Jensen
と
B.D.
エバヴォルド
.
2000
.
DO11.10およびOT-II T細胞は、C末端のオバルブミン323-339エピトープを認識する
.
J. Immunol.
164
:
4706
4712
.
https://doi.org/10.4049/jimmunol.164.9.4706
Google Scholar
クロスフィルム
パブコメ
セフィク
,
E.
,
N.
ゲバ-ザトルスキー
,
S.
オー
,
L.
Konnikova
,
D.
ゼンムール
,
A.M.
マクガイア
,
D.
バージン
,
A.
オルティス・ロペス
,
M.
Lobera
,
J.
Yang
, et al.
2015
.
個々の腸内共生生物は、RORγ⁺制御性T細胞の異なる集団を誘導する
.
サイエンス
.
349
:
993
997
.
https://doi.org/10.1126/science.aaa9420
Google Scholar
クロスフィルム
PubMed
ソネンブルグ
,
J.L.
(ソネンブルグ) 、および
F.
Bäckhed
.
2016
.
ヒトの代謝のモデレーターとしての食事-微生物叢の相互作用
.
ネイチャー
.
535
:
56
64
.
https://doi.org/10.1038/nature18846
Google Scholar
クロスフィルム
パブコメ
スチュアート
,
T.
,
A.
バトラー
,
P.
ホフマン
,
C.
ハーフェマイスター
,
E.
パパレキシ
,
W.M.
Mauck
を、3rd、
Y.
ハオ
,
M.
ストエキウス
,
P.
スミベール
を、そして
R.
サティヤ
.
2019
.
シングルセルデータの包括的な統合
.
セル
.
177
:
1888
1902 e1821
.
https://doi.org/10.1016/j.cell.2019.05.031
Google Scholar
クロスフィルム
パブコメ
スジノ
,
T.
,
M.
ロンドン
,
D.P.
ホイテマ・ファン・コーニネンブルク
,
T.
レンドン
,
T.
Buch
,
H.M.
シルヴァ
,
J.J.
ラファイユ
,
B.S.
ライズ
を、そして
D.
ムシダ
.
2016
.
制御性・上皮内CD4⁺ T細胞の組織適応は腸の炎症を制御する
.
サイエンス
.
352
:
1581
1586
.
https://doi.org/10.1126/science.aaf3892
Google Scholar
クロスフィルム
パブコメ
竹内
,
A.
と
T.
斉藤
.
2017
.
CD4+T細胞の細胞傷害性サブセットであるCD4 CTL、その分化と機能
.
Front. Immunol.
8
:
194
.
https://doi.org/10.3389/fimmu.2017.00194
Google Scholar
クロスフィルム
パブコメ
トーガソン
,
T.R.
,
A.
リネーン
,
N.
モーズ
,
S.
アノーバー
,
V.
マテオ
,
F.
リュー・ローカット
,
O.
Hermine
,
S.
ヴィジャイ
,
E.
Gambineri
,
N.
セルフ=ベンスサン
他
2007
.
FOXP3遺伝子のノンコーディング領域の欠失によるIPEX症候群の変種としての重篤な食物アレルギーの発生
.
胃腸病学
.
132
:
1705
1717
.
https://doi.org/10.1053/j.gastro.2007.02.044
Google Scholar
クロスフィルム
パブコメ
ワイナ
,
H.L.
,
A.P.
ダ・クーニャ
,
F.
クインタナ
を、そして
H.
ウー
.
2011
.
経口耐性
.
Immunol. Rev.
241
:
241
259
.
https://doi.org/10.1111/j.1600-065X.2011.01017.x
Google Scholar
クロスフィルム
パブコメ
ウィッカム
,
H.
2016
.
ggplot2: データ解析のためのエレガントなグラフィックス
.
シュプリンガー・ヴァーラグ
.
ニューヨーク
グーグルスカラー
クロスレフ
ヤン
,
B.H.
,
S.
Hagemann
,
P.
ママレリ
,
U.
ラウアー
,
U.
ホフマン
,
M.
ベックシュテット
,
L.
フェーセ
,
I.
プリンツ
,
J.
ペゾルト
,
S.
スエルバウム
, et al.
2016
.
RORγtを発現するFoxp3+ T細胞は、腸の炎症時に抑制能力を高める安定した制御性T細胞エフェクター系譜を表す
.
Mucosal Immunol.
9
:
444
457
.
https://doi.org/10.1038/mi.2015.74
Google Scholar
クロスフィルム
パブコメ
趙
,
Q.
,
S.N.
ハーバー
,
R.
コルデ
,
I.J.
ラトーレ
,
H.M.
Tun
,
T.R.
シューブ
,
H.
ターナー
,
J.J.
ムーン
,
E.
カフィプール
,
R.J.
ザヴィエ
, et al.
2017
.
微生物叢と相互作用するコレラ毒素によるマウス腸内の恒常性Th17細胞の選択的誘導
.
J. Immunol.
199
:
312
322
.
https://doi.org/10.4049/jimmunol.1700171
Google Scholar
クロスフィルム
PubMed
著者ノート
情報開示 著者は競合する利害関係が存在しないことを宣言している。
© 2023 Lockhart et al.
この記事はクリエイティブ・コモンズ・ライセンス(Attribution 4.0 International, as described at https://creativecommons.org/licenses/by/4.0/) の下で利用可能です。
補足データ
表S1
は、タンパク質抗原除去固形飼料(AA)の組成を示す。
docxファイル
表S2
AAまたはチョウ飼料を与えたSPFマウスの16S rRNAシーケンスの結果を示す。xlsxファイル
表S3
AA、AA + OVA、またはチャウ食を与えたGFおよびOligo-MM12マウスの全CD4+ T細胞UMAPクラスターで発現した遺伝子の違いを示す。xlsxファイル
表S4
AA、AA + OVA、またはチャウ食マウスからのIE CD4+T細胞間の三元差動遺伝子発現試験の結果を示す。xlsxファイル
表S5
AA、AA + OVA、またはチャウ食を与えたGFおよびOligo-MM12マウスからのTreg UMAPクラスター間で差次的に発現した遺伝子を示す。xlsxファイル
表S6
は、AA、AA+OVA、またはチャウ食マウスのTreg間の三元差分遺伝子発現試験の結果を示す。xlsxファイル
表S7
OVA摂食、耐性、およびアレルギーにおいて、iSellTomatoマウスからの全CD4+ T細胞UMAPクラスター間で差次的に発現した遺伝子を示しています。xlsxファイル
表S8
OVA摂食、耐性、およびアレルギーにおけるiSellTomatoマウス由来のTomato-IE CD4+T細胞間の三元差動遺伝子発現試験の結果を示す。xlsxファイル
表S9
は、OVA 摂食、耐性、およびアレルギーにおける、iSellTomato マウスからの Tomato+ IE CD4 T 細胞間の三元差分遺伝子発現試験の結果を示す。xlsxファイル
表S10
は、OVA摂食、耐性、およびアレルギーにおける、iSellTomatoマウスのTomato+ LP CD4+T細胞間の3ウェイ差分遺伝子発現試験の結果を示す。xlsxファイル
表S11
OVA摂食、耐性、およびアレルギーにおけるiSellTomatoマウスからのTreg UMAPクラスター間の差次的発現遺伝子を示す。xlsxファイル
表S12
OVA特異性について試験したscRNAseqデータセットで同定されたTCRに関する補足情報を示す。xlsxファイル
最新記事
最新号
アーカイブ
アラート
原稿を投稿する
著者向け説明
図書館員の方へ
JEMについて
編集部・スタッフ紹介
ポリシー&パーミッション
広告掲載について
お問い合わせ
ニュースルーム
個人情報保護方針
フェイスブック
ツイッター
インスタグラム
YouTube
RSSフィード
フェイスブック
ツイッター
インスタグラム
YouTube
オンライン ISSN 1540-9538
プリント ISSN 0022-1007
本サイトはクッキーを使用しています。当サイトの利用を継続することで、プライバシーポリシーに同意したものとみなされます。
同意する
この記事が気に入ったらサポートをしてみませんか?
