
初心者の菌叢解析 Qiime2で解析(9) Taxonomic analysis
前回に引き続き、Qiime2解析を進めていきます。
今回はひたすらコマンドを入れていく流れになります。
今回の解析はQiime2 docsの「”Moving Picture" tutorial」を参考に進めておりますので、そちらもご参照ください。
それではいつも通りQiimeを起動してください。
1.table.qzaの確認
まずは以下のコマンドを入れます。「mv」は名前の変更(場所の移動)を指示するコマンドです。
mv rep-seqs-dada2.qza rep-seqs.qza
mv table-dada2.qza table.qza以下のコマンドでtable.qzaの可視化を行います。
qiime feature-table summarize \
--i-table table.qza \
--o-visualization table.qzv \
--m-sample-metadata-file sample-metadata.txt生成された「table.qzv」をQiime2 Viewで確認してみます。

「Overview」のタブではデータのサマリーが見られます。ポイントとして、Qiime2で出てくる「Feature」は確認できた配列を表しています。1つの配列は1つの菌種を定義しており、同じFeatureがたくさん見られればその配列を有する菌がたくさん存在するということになります。
「Interactive Sample Detail」のタブでは各サンプルでどれくらいのFeatureが検出されたか確認できます。Featureがたくさん検出出来ればそれだけレアな菌が見つかる可能性が出てきます。つまり、より深くサンプリングして解析が可能ということになります(Sample Depth)。
「Feature Detail」のタブでは各Featureの頻度やいくつのサンプルで見られたかなどの情報が載っています。各FeatureにはそれぞれIDが振られており、30文字程度の英数字が並んでいます。これらのIDは今回の解析でランダムに振られたIDですので、他の解析とは共通していません。このIDを見ることで、次の「rep-seqs.qza」で該当する配列を見ることが出来ます。
2.rep-seqs.qzaの確認
以下のコマンドでrep-seqs.qzaの可視化を行います。
qiime feature-table tabulate-seqs \
--i-data rep-seqs.qza \
--o-visualization rep-seqs.qzv生成された「rep-seqs.qzv」をQiime2 Viewで確認してみます。
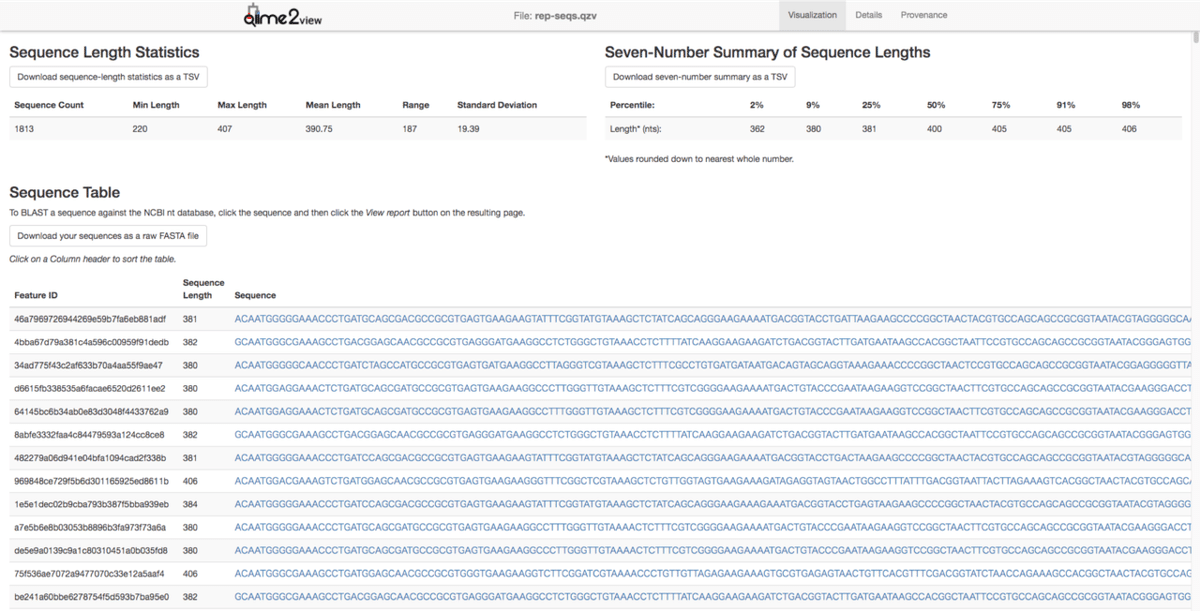
それぞれのFeature IDが対応している16S rRNAの配列を確認することが出来ます。こちらの配列をBLASTで検索すれば菌種同定も出来ますし、
EzBioCloud databaseでの解析も可能です。
3.Taxonomic analysis
次に菌叢の組成を可視化していきます。このタイミングで、classifireファイルが必要になってきます。今回は、Qiime2のチュートリアルからダウンロードしたものを使用します。
以下のコマンドを入れていきます。
qiime feature-classifier classify-sklearn \
--i-classifier gg-13-8-99-515-806-nb-classifier.qza \
--i-reads rep-seqs.qza \
--o-classification taxonomy.qzaコマンドの通り、classifierファイルと「rep-seqs.qza」ファイルを使用して「taxonomy.qza」ファイルを生成しています。
次に、「taxonomy.qza」ファイルを可視化します。
qiime metadata tabulate \
--m-input-file taxonomy.qza \
--o-visualization taxonomy.qzv出来上がった「taxonomy.qzv」をQiime2 Viewで確認してみます。
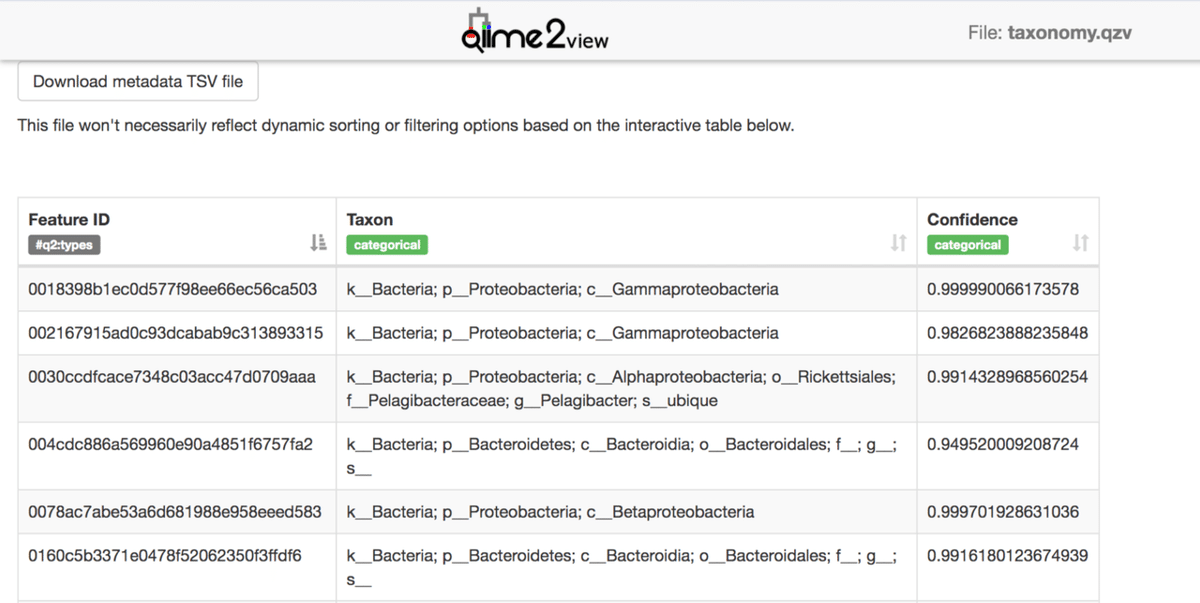
Feature IDに菌の名前が割り振られました。今回は画面外ですが、右上にある「Search:」を使えば特定の菌を探すことも出来ます。ただ、完璧に菌種を同定できるわけではないので注意が必要です。また、「rep-seqs.qzv」を参照すれば配列が分かりますので、より詳しい菌株まで追いかけることも出来ます。
最後に以下のコマンドでグラフを作成します。
qiime taxa barplot \
--i-table table.qza \
--i-taxonomy taxonomy.qza \
--m-metadata-file sample-metadata.txt \
--o-visualization taxa-bar-plots.qzv生成された「taxa-bar-plots.qzv」をQiime2 Viewで確認します。

赤枠の「Taxonomic Level」で階層を変更できます。Level 2で見るとマウスの腸内には「Firmicutes」が一番多いが、大阪湾には生息しておらず「Proteobacteria」が最も高い占有率だと言うことが分かります。
Firmicutesが腸内にしかいないとは知りませんでした。
この記事が気に入ったらサポートをしてみませんか?
