
初心者の菌叢解析 Qiime2で解析(5) ファイルの準備編 ~sample-metadataファイル~
Qiime2での解析に関する投稿を行っています。是非ご確認ください。
1.Qiime2を動かすのに必要なファイル
以前の記事でも記載したのですが、必要なファイルが4つあります。
1. Sequenceファイル(.fastqもしくは.fastq.gz)
2. manifestファイル(.csv)
3. sample-metadataファイル(.txt)
4. classifierファイル(.qza)
今回は3つ目のsample-metadataファイルの準備を行います。
2.sample-metadataファイルとは
sample-metadataファイルは解析するファイルに
様々な情報を付加する為のファイルです。
前回作成したmanifestファイルでは各サンプル名を設定できますが、
グループの設定等の情報は入っていません。
他にも時間や高さ、深さ等の数字の情報も追加可能です。
3.sample-metadataファイルの作成
まずはエクセルを開きます。
1行目の最初に「sample-id」と入れ、その下に「#q2:types」と入れます。

sample-idの列にはmanifestファイルで入れたサンプル名を入れていきます。

次にこれらを「Osaka」と「Mouse」の2つのグループに分けたいので、
グループをBの列に入れていきます。2つのカテゴリーに分けるので2行目には「categorical」と入れます。数字の場合は「numeric」と入れます。

グループの名前は何でもかまいません。「Origin」でも大丈夫です。
また、グループ分けをより細かく行いたい場合もあると思います。
その時は右に好きなだけどんどん追加してください。

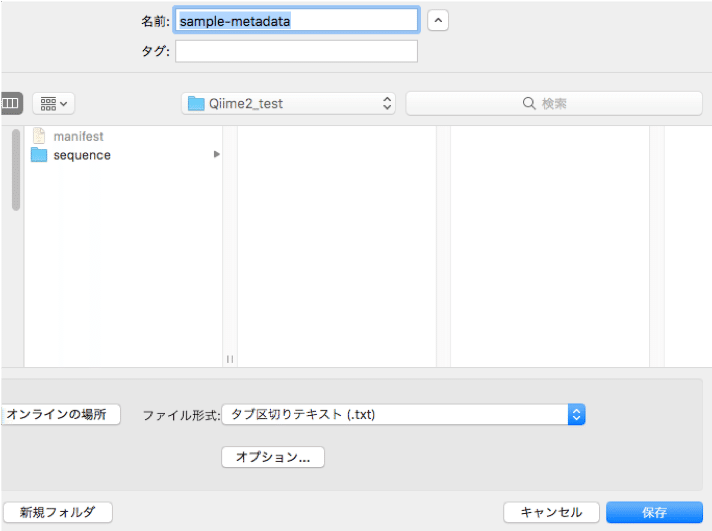
作成したら「sample-metadata」と名前をつけて、
「タブ区切りテキスト(.txt)」でQiime2_testのフォルダに保存します。
これで解析時のグループ分けが出来ました。
今回は以上になります。次回はclassifierを作成します。
こちらは以前も投稿しているので、そちらを参考にしていただければ問題無いと思います。
この記事が気に入ったらサポートをしてみませんか?
