
初心者の菌叢解析 Qiime2で解析(13.1) PICRUSt2の結果をKEGG pathwayにマッピングしてみる
前回の投稿でPICRUSt2解析を行いました。
せっかくなので、KEGG pathwayにマッピングしてみたいと思います。
クリティカルな解析にはならないかもしれませんが、映えるのでやってみました。
プレゼンの際などでは視覚的な説得力が増すかもしれません。
また、前回投稿もあわせてご確認ください。
1.はじめに
KEGGはKyoto Encyclopedia of Genes and Genomesのことで、京都大学で作成された代謝経路を中心としたあらゆる情報が統合されたデータベースです。
今回はKEGGにPICRUSt2で明らかとなった情報をマッピングし、代謝経路の中での変化を見てみたいと思います。
Qiime2の解析はあまり関係ないですが、もちろんQiime2以外の解析でも使用可能です。
2.変化のあった酵素の選別と色づけ
前回の解析で得た「ko-feature-table.biom.tsv」をエクセルで開きます。

一番左の項目はKEGGのEntry番号(#OTU ID)になっており、一番上の「K00001」を検索すると以下のように「alcohol dehydrogenase」がヒットします。

この情報を元に、マッピングしてみます。
2-1. 平均値とpseudocountの追加
まずは、右に平均値を入れます。
さらに、平均値が「0」だとこの後の変化量を計算しにくいので、ここでは「0.0001」を各平均値に擬似的に追加します(pseudocount)。
この数字は適当に入れていますので、ちゃんと解析するときは調べてからやります(すいません)。
ただ、ここでのデータでは最小値が「1」ですので、「0.0001」の追加で結果が変化することは無いと思います(統計処理には使用しません)。

2-2.P値の算出(P value)
次にT-testで有意差検定を行います。
本当は多重比較の問題があるので、使用不可ですが、ここでは見逃してください。本番ではちゃんとFDRを計算します。
「P value」を右のカラムに追加します。
一番上の計算ならば「=TTEST(B3:D3,E3:G3,2,2)」ですね。

2-3.変化割合の算出(logFC)
次に変化割合(FC: fold change)を計算します。
ここでは「0.0001」を追加した「pseudocount」の値を使用して、変化割合を計算します。
Mouse÷Osakaを計算して、変化割合を算出します。
2倍の変化であれば「2」と出ますし、半分に減少していれば「0.5」と出ると思います。「2」と「0.5」は値としては全然違いますが、変化割合で見ると同じ変化量ということになります(2倍と半分なので)。
ですので、底が「2」のlogをとります。そうすれば「2」は「1」に、「0.5」は「-1」に変換され、直感的に理解しやすくなります。
そのため、表記はLogFCとなります。
RNA-seqデータでも良く目にすると思います。

2-4.変化に合わせて色をつける
次に一番右にKEGG Entry番号を再度入れます。

次に、KEGG Entry番号を入れた右側に色を指定していきます。
Mouseで増加している物は「赤」でOsakaで増加している物は「青」です。
つまり、logFCが正の値であれば「赤」、負の値であれば「青」です。
エクセルの「if関数」を使用して、colorのカラムを追加します。
「K00001」の行だと以下の通りです。
色は小文字で指定します。
=IF(M3>0,"red","blue")
2-5.変化のあった酵素のフィルタリング
最後に上のカラムにフィルターを入れて、P value<0.01、logFCは「>1」もしくは「<-1」でフィルターをかけます。
正もしくは負で2倍以上の有意に変化したKEGG Entryの抽出ができます。

あとは右端のKEGG Entry番号と色のカラムをコピーしておきます。
「#OTU ID」とか「color」とかの情報はいらないので、その下をコピーしておきます。

3.KEGG Mapperへの入力
後はKEGG Mapperへ入力するだけです。
「Color」をクリックします。

後は先ほどコピーしたKEGG Entry番号と色のカラムをコピペします。
そして、下の実行ボタンを押します。

結果が出てくるので、興味のある物を確認してみます。

炭素固定経路(Carbon fixation pathway in prokaryotes)を確認してみます。

それぞれでより多く持っている遺伝子には差があることがよく分かります。
より多く持っているのであって、どちらかしか持っていないというわけでははないことに注意してください。
環境に合わせて菌叢の得意分野が違うということだと思います。
バンコマイシン耐性やβラクタム系抗生物質耐性を見ると腸内細菌の方が関連遺伝子を多く有しているようです。


耐性菌の発生が問題視される昨今、このような内容は少しショッキングです。
やはり、抗生物質に触れる機会が多いからこのような結果になるのでしょうか。それとも、生物学的・生態学的に妥当な結果なのでしょうか。
抗生物質は土壌の放線菌が産生菌として有名ですが、海ではどうなのでしょうか。マウスは陸上生物ですので、関連しているのでしょうか。
何か分かる人コメントください。
スターチとスクロースの代謝ですが、こちらも腸内細菌の得意分野のようです。
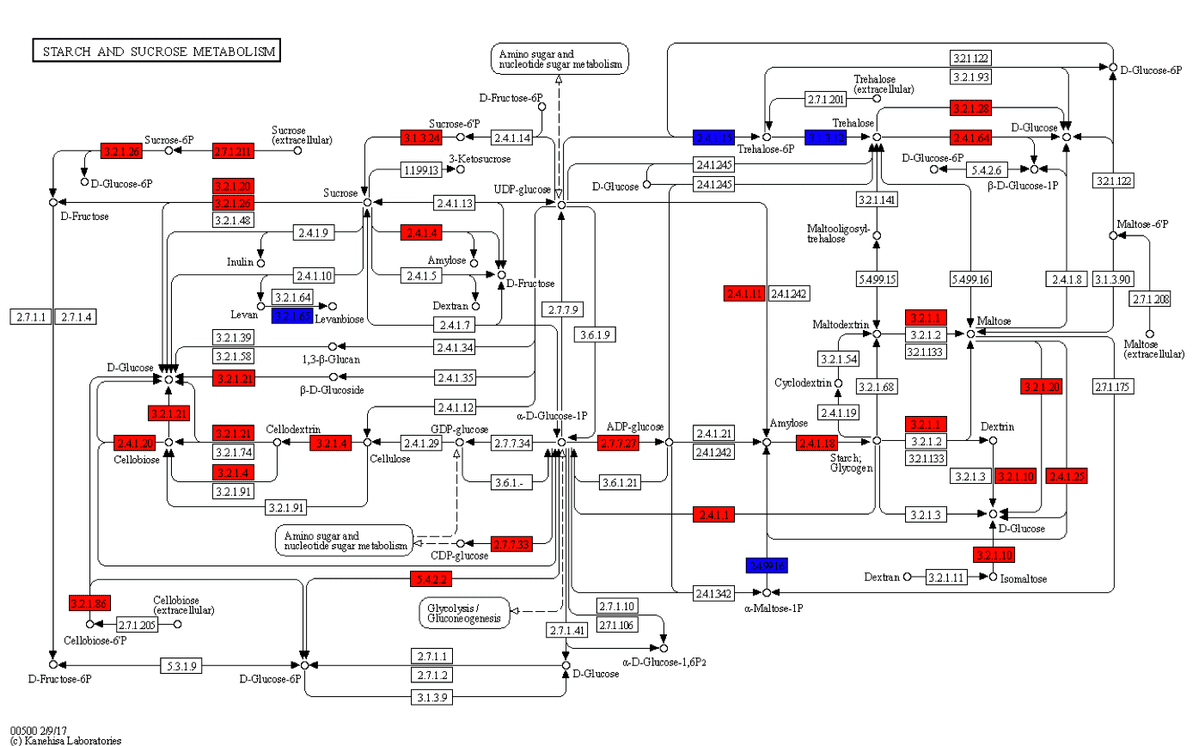
海洋微生物叢の方もそのうち追記できればと思います。
完全に趣味みたいな感じですが、今回は以上になります。
面白いので、是非やってみてください。
