
Rを使って複数の図を低労力で出力する
はじめに
こんなときありませんか・・・?
複数の変数から、変数の組み合わせを変えて関係をプロットしたい
種や調査地点ごとにプロットを生成し、一枚の図にまとめたい
調査地点ごとに多変量解析をおこない、特徴をプロットしたい
組み合わせパターンごとにコードを書いて出力したり、複数の図をパワポなどで組み合わせると大変ですよね
・・・そこでRの出番です
準備する
次のようなシナリオを考えます。
①魚の体長–体重の関係が、採取年や調査地点によって異なっているか
②採取年および魚種ごとに、魚の体長–体重の関係を確認する
③魚の胃内容物組成の特徴が、調査地点によって異なっているか
ただし、採取年は2年、魚種は5種、調査地点は3地点あるとします。
データファイル
今回は、サケ科魚類のデータセットを使って、コードを紹介していきます。NOAA Northwest Fisheries Science Center > Data Sets > Diet
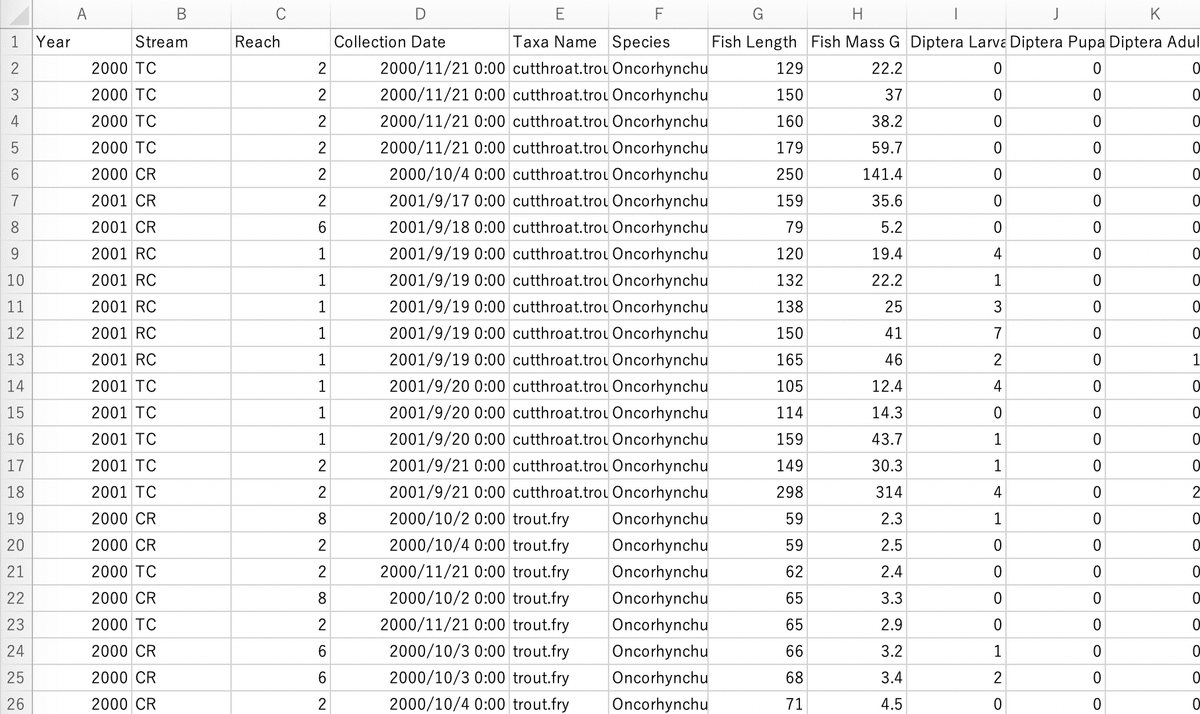
ポイントは、一枚のcsvファイルに多数の変数(捕獲年、調査地点、体長、体重、胃内容物組成など)が含まれている点です。各列に変数がおさまっています。
環境構築(Rパッケージ)
今回は、以下の3つを使用します。
library(dplyr)
library(ggplot2)
library(gridExtra)# library(dplyr) データ操作や集計で大活躍するライブラリーです
# library(ggplot2) プロット作成で大活躍するライブラリーです
# library(gridExtra) プロットのレイアウトのために使います
インストールが済んでいない場合は、install.packages("ライブラリー名")でダウンロードしましょう。
準備はここまで。
やってみよう
データセットの概観をつかむ
まずは、すべての変数の組み合わせで2次元プロットを作ってみます。
data <- read.csv("Diet - Cedar River Fish Diets 2000-2001 Data.csv", header = T)
plot(data[1:8])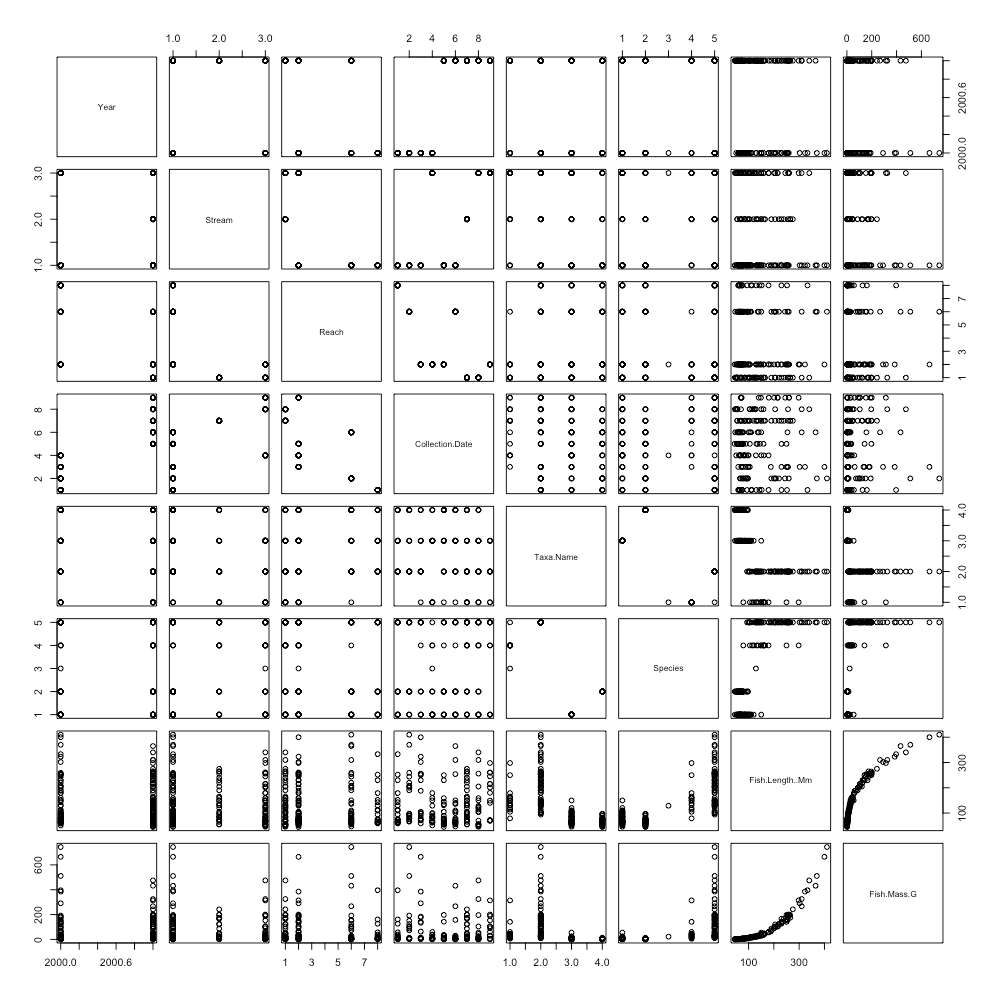
解説
#1 read.csv() でデータセットを読み込みます。
今回のデータセットでは、一行目に列名の情報が入っているため、"header = T" を指定します。
#2 plot("データセット") ですべての変数(列)の組み合わせで2次元プロットを作成します。
ただし、今回のデータセットは列数がかなり多いため、環境によっては描画に失敗するかもしれません。"figure margins too large"というエラーメッセージが出ていませんか?
このエラーを解消するため、plot(data[1:8]) として変数の数を減らしました。
data[1:8]は、データセットから1から8列目までのデータを抽出するということです。
さて、たった2行のコードで、すべての変数の組み合わせによる2次元プロットが生成されました
データの概観をつかむだけならこれで十分ですが、研究プレゼンや論文では着目する変数を絞って作図することでしょう。
ここからはデータ操作とプロットのレイアウトに役立つコードを紹介していきます。
dplyrを使ったデータ操作~列抽出~
冒頭のシナリオを考えてみます
①魚の体長–体重の関係が、採取年や調査地点によって異なっているか
②採取年および魚種ごとに、魚の体長–体重の関係を確認する
それでは、①や②を確認できるようなプロットを作ってみましょう。
まず、データセットを整理します。
データセット"data"から、"採取年"、"調査地点"、"種名"、"魚の体長"、および"体重"のデータを抽出してみましょう。
データセット"data"、つまり"Diet - Cedar River Fish Diets 2000-2001 Data.csv"では、それぞれの変数が
列名"Year", "Stream", "Species", "Fish.Length..Mm", "Fish.Mass.G" として格納されています。
colnames(data)
data_A <- dplyr::select(data,
Year, Stream, Species, Fish.Length..Mm, Fish.Mass.G)
plot(data_A)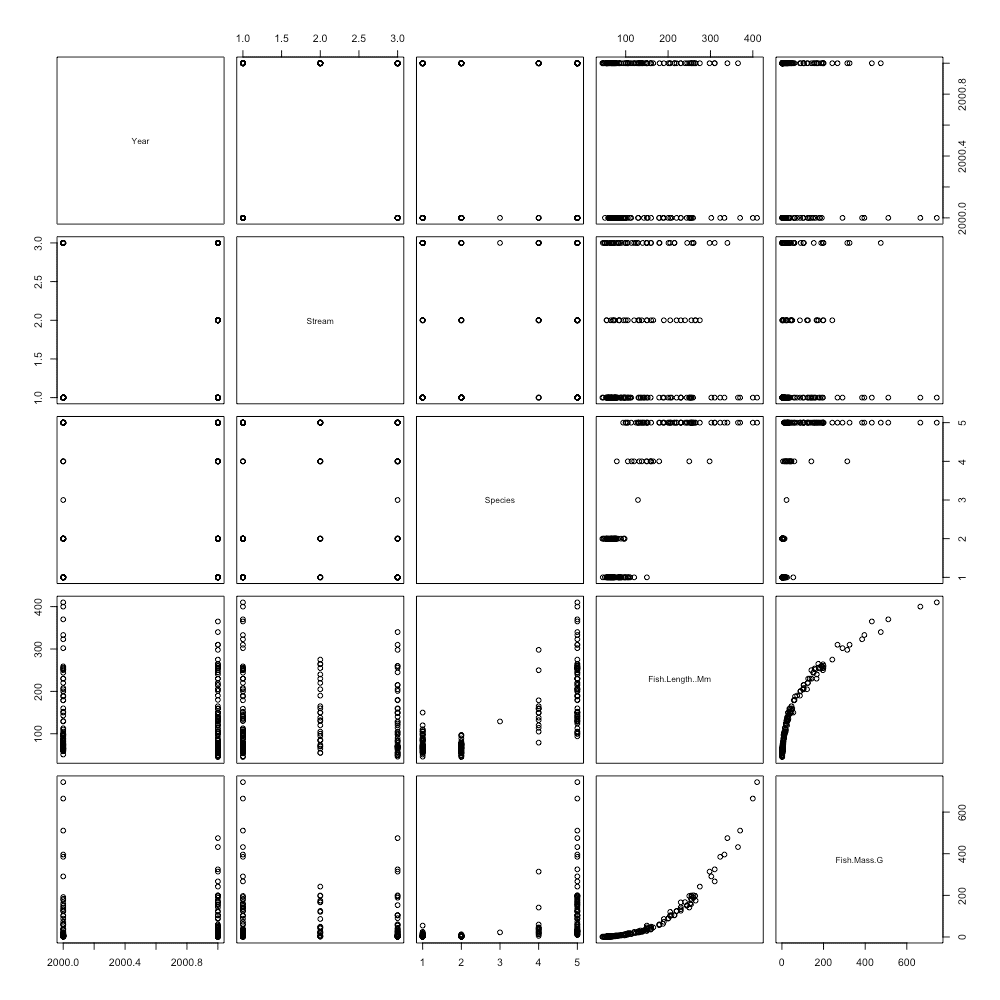
解説
#3 colnames() でデータセットの列名を確認します。この作業は必須ではありません。
次のコードで列名を指定する際、列名にスペルミスがあるとエラーとなってしまうため、
colnames() で出力される列名をコピペすることでエラーを防止する狙いがあります。
#4 dplyr::select() で、データセットから任意の列のデータを抽出します。関数のルールは、dplyr::select(データセット, 列名1, 列名2, ..) です。
列名のところに列番を指定する方法でも、任意の列のデータを抽出できます。
例)dplyr::select(data, 1, 2, 6, 7, 8)
抽出したデータを、data_A という箱に格納しておきます。
#5 plot() で、すべての変数(列)の組み合わせで2次元プロットを作成します。(コード#2と同じ)
dplyrを使ったデータ操作~行抽出~
次に、採取年別にデータセットを作成します。
data_00 <- dplyr::filter(data_A, Year == 2000)
data_01 <- dplyr::filter(data_A, Year == 2001)解説
#6,7 dplyr::filter() で、任意の列から条件が一致する行を抽出します。
関数のルールは、dplyr::filter(データセット, 列名 == 変数) です。
今回は、2000年と2001年に採取された個体のデータを分割して、別々のデータセットを作りたいので、
列"Year"で"2000"が入力されている行をdata_00に、"2001"が入力されている行をdata_01に格納しました。
データ抽出のポイント
i. データセットから、任意の列データを抽出するとき・・・dplyr::select()
ii. 任意の列データから、条件が一致する行のみを抽出するとき・・・dplyr::filter()
複数の図を一枚の図にレイアウトする
最後に、各採取年のデータセットに対して、体長–体重の関係をあらわす散布図を作成し、一枚の図にまとめてみましょう。
p_00 <- ggplot(data_00, aes(x = Fish.Length..Mm, y = Fish.Mass.G)) +
geom_point(size=3)
p_01 <- ggplot(data_01, aes(x = Fish.Length..Mm, y = Fish.Mass.G)) +
geom_point(size=3)
gridExtra::grid.arrange(p_00, p_01, ncol = 1)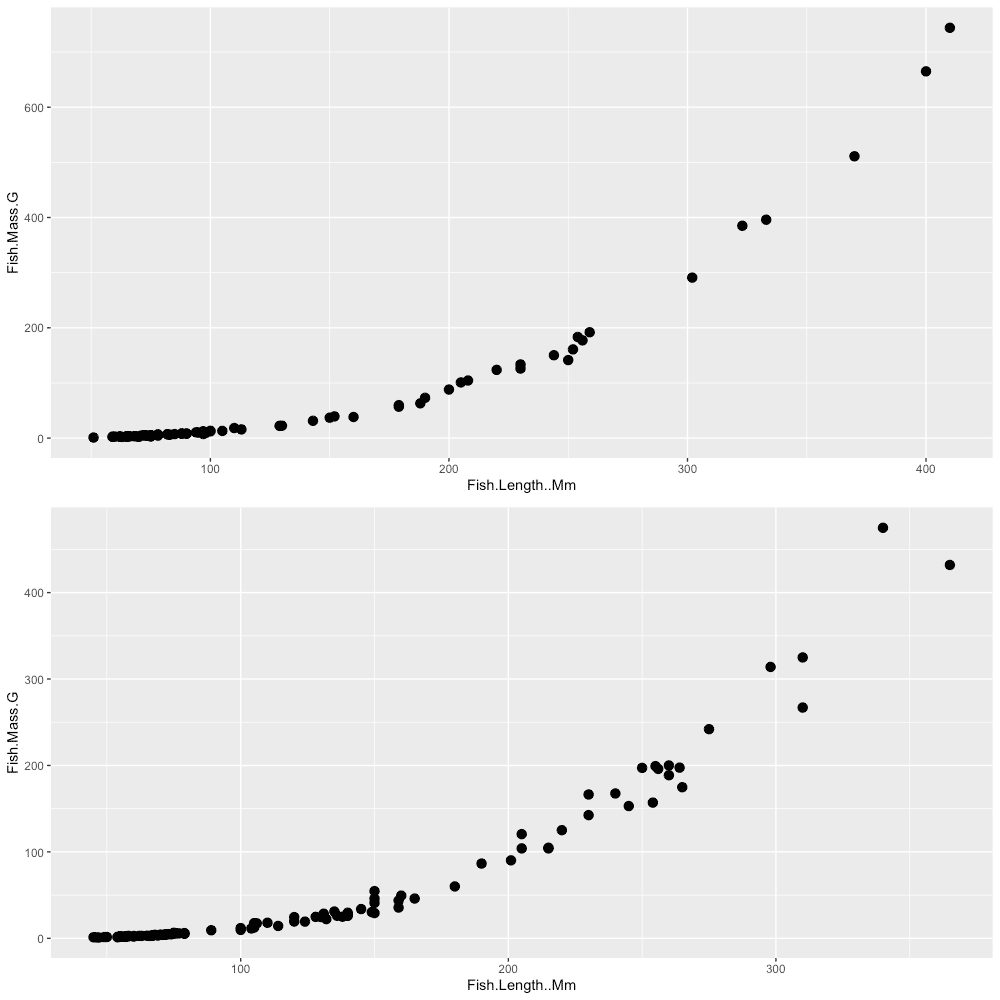
解説
#8 ggplot(data_00, aes(x = Fish.Length..Mm, y = Fish.Mass.G)) + geom_point(size=3) で、体長–体重の関係を示す散布図を作ります。
プロットのデータは、p_00 という箱に格納しておきます。
#9 では、#8と同様のルールで、データセットdata_01に対して散布図を作成し、p_01 という箱に格納しておきます。
もはや説明不要かもしれませんが、念のためggplot関数のルールを。
ggplot(データセット, aes(x = X軸データ, y = Y軸データ)) で、キャンバスを作ります。
そして、+ geom_point() を付け加えることで、散布図を描画します。
…棒グラフを作りたい場合は + geom_bar() を、箱ひげ図を作りたい場合は+ geom_boxplot() を書くといった具合です。
#10 gridExtra::grid.arrange() で、複数のプロットを一枚の図に配置します。
関数の基本的なルールは、gridExtra::grid.arrange(プロットデータ1, プロットデータ2) です。今回は、p_00 と p_01 の2枚の散布図を指定しました。
縦方向に図を積みたい場合は"ncol = 1"を指定しますが、横方向に図を並べたい場合は"nrow = 1"とします。
gridExtra::grid.arrange() を使う利点は、ggplot2で作成した図であれば、どんな形態の図でも自由に組み合わせて一枚の図として出力できる点です。
たとえば、「体長–体重の関係を示す散布図」だけでなく、「箱ひげ図」や「棒グラフ」も組み合わせることができます。
以下にサンプルコードを示します。
p_00 <- ggplot(data_00, aes(x = Fish.Length..Mm, y = Fish.Mass.G)) +
geom_point(size=3)
p_01 <- ggplot(data_01, aes(x = Fish.Length..Mm, y = Fish.Mass.G)) +
geom_point(size=3)
p_box <- ggplot(data, aes(x = Stream, y = Fish.Mass.G)) +
geom_boxplot()
p_bar <- ggplot(data, aes(x = Stream, y = Diptera.Larvae, fill = Species)) +
geom_bar(stat = "identity")
gridExtra::grid.arrange(p_00, p_01, p_box, p_bar,
ncol = 2, nrow = 2)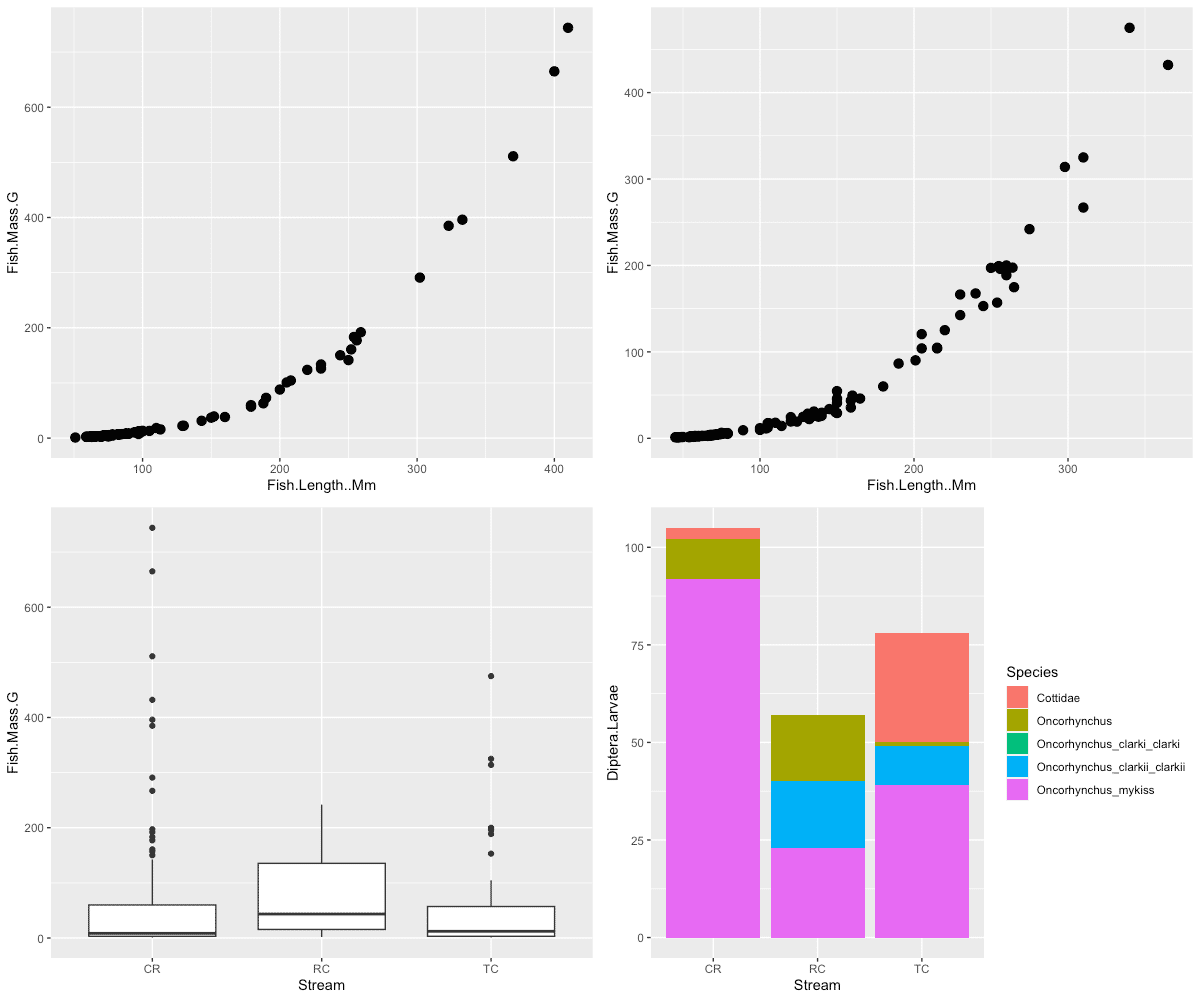
複数の図を一括で生成する
さて、ggplot() と gridExtra::grid.arrange() を活用することで、見栄えの良いプロットを
簡単に生成し、一枚の図としてまとめられるようになりました。
今回のデータセットでは、採取年が"2000"と"2001"の2つしかなかったため、比較的短いコードで済みました。
しかし、変数内の ID の数が多い場合には、先ほど紹介した方法ではかなり労力がかかります。
そこで、ggplot() を使いながら、もっと簡単に複数の図を生成するコードを紹介します。
#data_A <- dplyr::select(data, Year, Stream, Species, Fish.Length..Mm, Fish.Mass.G)
p <- ggplot(data_A, aes(x = Fish.Length..Mm, y = Fish.Mass.G)) +
geom_point(size = 3)
p + facet_grid(~Year)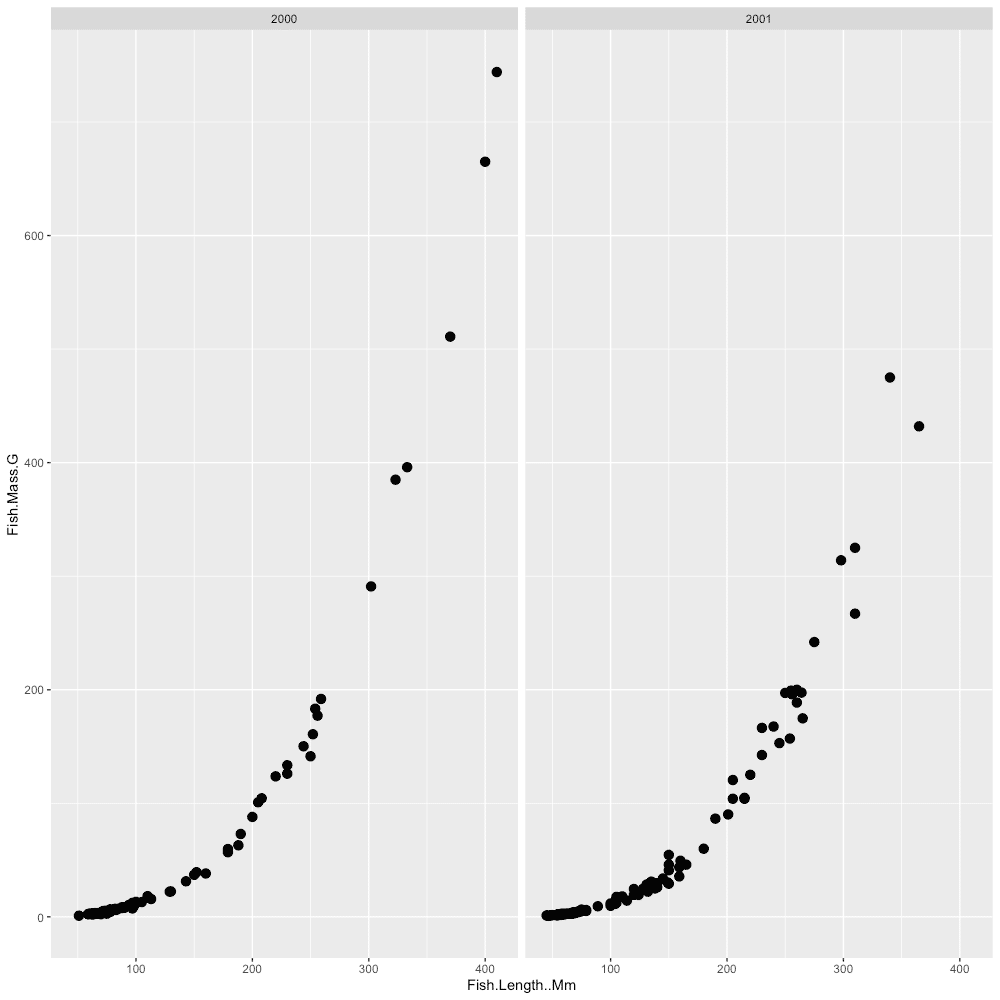
解説
#.# ここではコード#4 で作成したデータセット"data_A"を使います。
"data_A"は、生データセット"data"から、"採取年"、"調査地点"、"種名"、"魚の体長"、および"体重"のデータを抽出したものでした。
つまり、採取年ごとにデータセットを作成する作業はおこなっていません。
#11 ggplot(data_A, aes(x = Fish.Length..Mm, y = Fish.Mass.G)) + geom_point(size = 3) で、体長–体重の関係をあらわす散布図を作ります。
関数のルールは、コード#9で紹介したとおりです。
プロットデータを p という箱に保管しておきます。
#12 facet_grid() で、任意の変数カテゴリごとにデータセットを分けて散布図を作成します。
関数のルールは、プロットデータ + facet_grid(~ 列名) です。
今回は、採取年ごとに散布図を作成したいので、変数(列名)として"Year"を指定しました。
こうすることによって、"2000"と"2001"のデータを分けて散布図を作ってくれます。
調査地点ごとに散布図を作成する場合は、p + facet_grid(~Stream) とすればOKです。
また、facet_grid() 関数のオプションとして、scales="" があります。
scales="" では、プロットごとに軸の範囲をいい感じに自動設定してくれます。
設定の詳細については、以下のサイトが参考になります。
facet_grid() を使う利点は、2変数までの組み合わせのプロットを瞬時に生成してくれる点です。
たとえば、「②採取年および魚種ごとに、魚の体長–体重の関係を確認したい」とき、
たった2行のコードを実行するだけで、②を一覧できます。
p <- ggplot(data_A, aes(x = Fish.Length..Mm, y = Fish.Mass.G)) +
geom_point(size = 3)
p + facet_grid(Species~Year)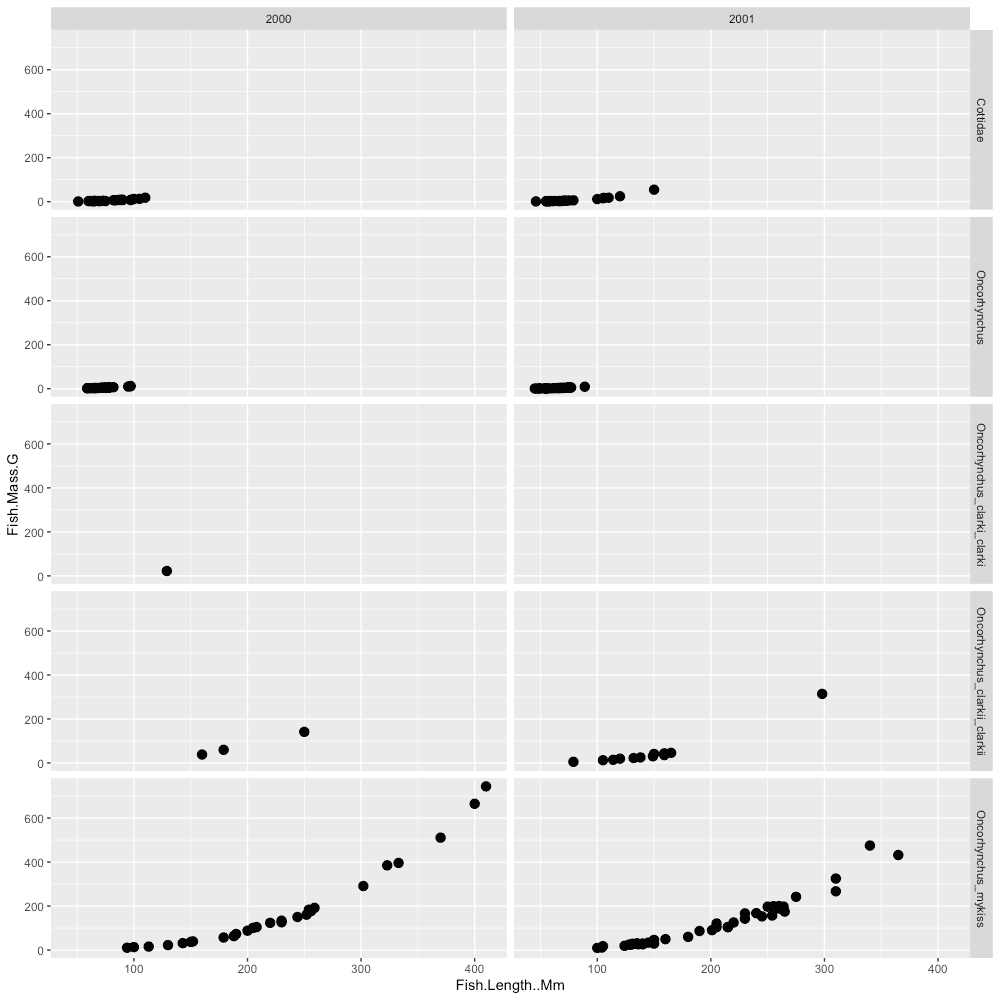
総括
gridExtra::grid.arrange() と facet_grid、どちらを使うべきか?
facet_grid() は、プロットの基本構造が同じでないと使えません。
先に紹介した gridExtra::grid.arrange() のように、散布図+箱ひげ図+棒グラフといった図の構造やタイプが異なる複数の図を組み合わせることはできません。
様々なタイプの図を自由に組み合わせたい場合には、 gridExtra::grid.arrange() を使うことを意識してコードを書くと良いでしょう。
「体長–体重の関係をあらわす散布図」という基本構造は同じで、変数(魚種や採取年)のみを変えてその関係を見たい場合には、facet_grid() が適しているでしょう。
基本は以上です。
なお、プロットの体裁(軸ラベル、プロットサイズ、凡例など)を操作する方法は、基本的に ggplot2 の操作と同様です。
今回の記事では詳細を紹介しませんが、気になる点などがあればコメントをいただければと思います。
応用編
ここからは、様々な図を自由に・大量に生成して、一枚の図におさめたいときに役立つアイデアとコードを紹介します。
この先の話はほぼ自己満足の可能性もありますが、
以前書いた「多変量解析、nMDS」の記事へのアクセスが多いことから(ありがとうございます!)、
nMDSプロットを大量生成できると嬉しい層もいると信じて、nMDSのプロットをまとめるコードを紹介したいと思います。
〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜〜
「やってみよう」編で紹介した図は、比較的シンプルなものでした。
たとえば、
体長–体重の関係のような、2変数の2次元プロットを大量生成する
体長データの分布を年ごとに箱ひげ図で示す
胃内容物組成を種ごとに棒グラフで示す
などです。
これらは最初に読み込んだデータセットを ggplot の基本構造
[ggplot(データセット, aes(x = 列1, y = 列2)) + geom_ホニャララ ] に
当てはめるだけで良いため、数行のコードで解決します。
一方で、データセットを直接、ggplotの基本構造に当てはめることができない場合もあります。
例えば、「③魚の胃内容物組成の特徴が、調査地点によって異なっているか」を示す図を作成する場合はどうでしょうか。
プロセスを細かく分割すると、(1) データセットから胃内容物組成に該当する列のみを抽出し、(2) 類似度行列の算出およびnMDSをおこない、(3) 2次元にプロットする 作業が必要です。
(2) のプロセスがあるため、作図または解析用コードがやや長くなります。
Rで複数のnMDSプロットを生成する
それでは、「③魚の胃内容物組成の特徴が、調査地点によって異なっているか」を手短に図示する方法を伝授しましょう。
ズバリ、for文の活用です。
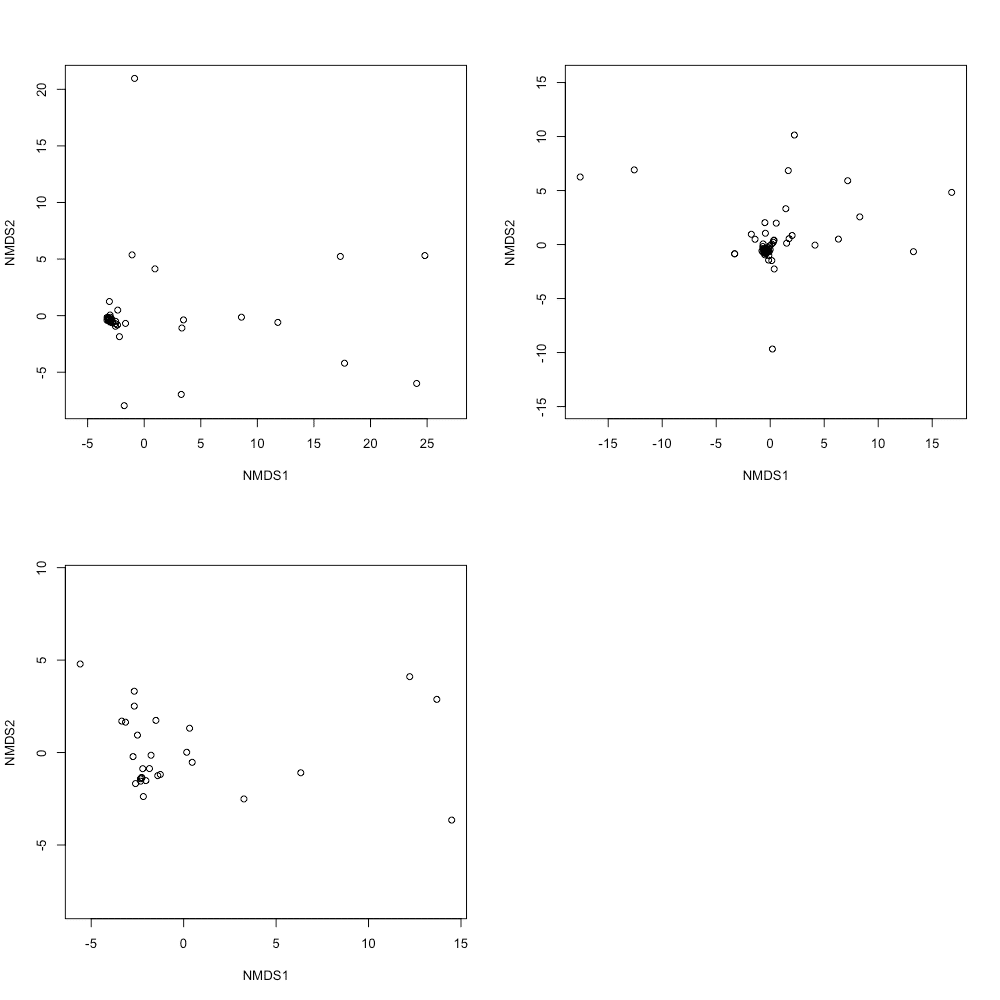
まずは、Rのデフォルト・plot関数で図を(大量)生成する方法を紹介します。
library(vegan)
str <- unique(data$Stream)
par(mfrow = c(2, 2))
for (i in 1:length(str)) {
site <- str[i]
data_str <- filter(data, Stream == site)
data_comm <- select(data_str, 9:28)
eudist <- vegdist(data_comm, "euclidean")
eumds <- metaMDS(eudist, k=2)
ordiplot(eumds,type="n")
points(eumds)
}
解説
#13 library(vegan) で、veganパッケージを呼び出します。行列の計算およびnMDSをおこなうためです。
#14 unique() で、調査地点"Stream"に含まれるIDを抽出します。
まず、data$Stream で、"data"データセットの"Stream"列にアクセスします。
data$Stream を実行すると、Stream列には、 地点TC が 51個、地点CR が99個、地点RC が 29個、入っていることがわかります。
> data$Stream
[1] "TC" "TC" "TC" "TC" "CR" "CR" "CR" "RC" "RC" "RC" "RC" "RC"
[13] "TC" "TC" "TC" "TC" "TC" "CR" "CR" "TC" "CR" "TC" "CR" "CR"
[25] "CR" "CR" "CR" "CR" "CR" "CR" "CR" "TC" "CR" "CR" "CR" "CR"
[37] "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR"
[49] "CR" "CR" "CR" "CR" "CR" "RC" "RC" "RC" "RC" "RC" "TC" "TC"
[61] "TC" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR"
[73] "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR"
[85] "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR"
[97] "CR" "CR" "RC" "RC" "RC" "RC" "RC" "RC" "RC" "RC" "RC" "RC"
[109] "RC" "RC" "RC" "RC" "TC" "TC" "TC" "TC" "TC" "TC" "TC" "TC"
[121] "TC" "TC" "TC" "TC" "TC" "TC" "TC" "CR" "CR" "CR" "CR" "CR"
[133] "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "TC" "TC" "TC" "TC"
[145] "TC" "TC" "TC" "TC" "TC" "TC" "TC" "CR" "CR" "CR" "CR" "CR"
[157] "CR" "CR" "CR" "CR" "CR" "CR" "CR" "CR" "RC" "RC" "RC" "RC"
[169] "RC" "TC" "TC" "TC" "TC" "TC" "TC" "TC" "TC" "TC" "TC"
> table(data$Stream)
CR RC TC
99 29 51 unique()で、任意の列に含まれるデータから、重複なしでIDを抽出します。
unique(data$Stream) とすることで、Stream列 [地点TC x 51個、地点CR x 99個、地点RC x 29個] から、調査地点 TC、CR、RCの3つの情報のみを抽出できます。
> unique(data$Stream)
[1] "TC" "CR" "RC"ひとまず、調査地点の情報を str という箱に情報を格納しておきます。
#15 par(mfrow = c(2, 2)) でキャンバスのレイアウトを決めます。
c(行数,列数) というルールです。今回は3枚の図を一枚の図にまとめるので、
par(mfrow = c(2, 2))でもいいし、par(mfrow = c(1, 3))でもOKです。
#16 for (i in number1:number2){ ..
…必殺 for 文です!
これから地点ごとにnMDSをおこなおうとしているのですが、地点名を変える以外は、すべて同じ作業の繰り返しです。
こういうとき、for文が役に立ちます。ルールは簡単です。
(1) "i" は変数です。とりあえずここでは i を使っていますが、どんなアルファベットを使っても良いです。大事なのは、ここで宣言したアルファベットの中身が変化するということです。
(2) number1:number2で、変数 "i" がとる範囲を指定します。3回同じ作業を繰り返す場合は、1:3とします。
今回、1:length(str) としているのは、地点の数だけ同じ作業を繰り返したいからです。
length(データ列)とすることで、データ列の長さ(格納されているデータ数)を計算してくれます。
str には調査地点 TC、CR、RCの3つのデータが入っているので、length(str) = 3となります。
(3) {} の中に、作業内容(コード)を書きます。
以下、作業コードの解説です。
#17 str[i] で、str列のうち、i番目のデータのみを抽出します。
まず、基本的な説明をすると、
データ列[数字] を実行することで、データ列の"数字"番目のデータにアクセスできます。
(ちなみに、行列の形をしているデータフレームであれば、データフレーム[数字1,数字2] を実行することで、データフレームの"数字1"行目・"数字2"列目のデータにアクセスできます)
for文中において、i = 1 のときは str列1番目のデータである "TC" にアクセス、
i = 2 のときは str列2番目のデータである "CR" にアクセスします。
この情報をsiteという箱に格納しておきます。
#18 filter(data, Stream == site) で、データセット"data" の列"Stream" から、site [i = 1 のときは "TC" 、i = 2 のときは "CR" ..] に一致する行を抽出します。(コード#6,7と同じ)
このデータセットを data_str という箱に格納しておきます。
#19 select(data_str, 9:28) で、データセット"data_str" から、9~28列目を抽出します。
説明が遅くなりましたが、生データセット"data" の9~28列目にサケ科魚類の胃内容物データが入っています。このデータを使って、nMDSをおこないます。
このデータセットを data_comm という箱に格納しておきます。
#20 vegdist(data_comm, "euclidean") で、胃内容物データからユークリッド距離を計算します。(*1)
計算結果を eudist という箱に格納しておきます。
#21 metaMDS(eudist, k=2) で、ユークリッド距離データに対してnMDSをおこない、2次元のデータに要約します。
計算結果を eumds という箱に格納しておきます。
#22,23 ordiplot(eumds, type="n") と points(eumds) で、nMDSのプロットを作成します。
#24 } for文を閉じます。カッコを忘れずに。
1回目(i=1のとき)の } までたどり着いた時点で、地点"TC" におけるユークリッド距離→nMDS→2次元プロットの作業が完了しています。
そしてfor文の冒頭に戻り、カウンターが i = 2 に変更されたあと、地点"CR" に対して同じ作業がおこなわれます。
*1 補足
vegdist() では、Bray–Curtis index や Jaccard indexなど、様々なタイプの類似度行列を計算してくれます。
ただし、今回のデータセットように「0しか入っていない列が存在する」場合は、類似度を計算することができません(エラーが出ます)。
ということで、ここではユークリッド距離を用いました。
なお、コード#20-23はveganパッケージで類似度行列やnMDSの解析をおこなうための操作です。
詳細な解説が必要な方は、こちらの記事をご参照ください。
ggplot2で複数のnMDSプロットを生成する
やはりggplotで作図したい!という人のために、コードを紹介します。
library(vegan)
str <- unique(data$Stream)
for (i in 1:length(str)) {
site <- str[i]
data_str <- filter(data, Stream == site)
data_comm <- select(data_str, 9:28)
eudist <- vegdist(data_comm, "euclidean")
eumds <- metaMDS(eudist, k=2)
data_ggp <- as.data.frame(scores(eumds))
p <- ggplot(data_ggp, aes(x = NMDS1, y = NMDS2)) +
geom_point(size = 3) +
theme_light() +
theme(panel.grid=element_blank())
assign(paste0("p", i), p)
}
gridExtra::grid.arrange(p1, p2, p3,
ncol = 2, nrow = 2)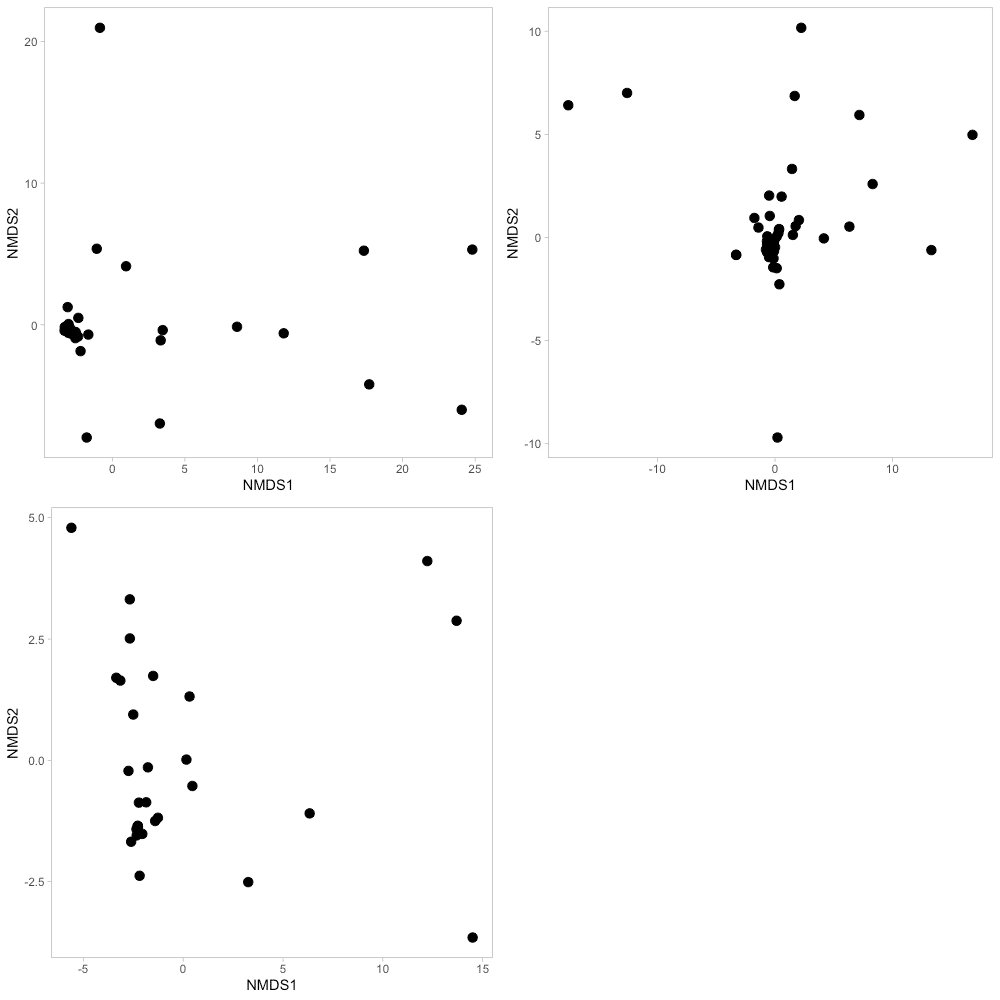
解説
#25 library(vegan) で、veganパッケージを呼び出します。(コード#13と同じ)
#26 unique() で、調査地点"Stream"に含まれるIDを抽出します。
ひとまず、str という箱に情報を格納しておきます。
(コード#14と同じ)
#27 for文の条件を指定します(コード#16と同じ)
#28-32 データセットの整形、ユークリッド距離、およびnMDSの計算をおこないます(コード#17-21と同じ)
#33 as.data.frame(scores(eumds))
まず、scores() で、nMDSのスコアを抽出します。これはveganパッケージの関数です。
また、as.data.frame() を実行してスコアデータを一つのデータフレームとして保管しておきます。この作業はggplotで描画するために必要です。
このデータセットを data_ggp という箱に格納しておきます。
#34 ggplot(data_ggp, aes(x = NMDS1, y = NMDS2)) + geom_point(size = 3) + theme_light() + theme(panel.grid=element_blank()) で、
データセット"data_ggp" に格納されているnMDSの結果をもとに、散布図を作成します。
theme_light() や theme(panel.grid=element_blank()) は図の体裁を整えるためのコードなので、あってもなくてもOKです。
最低限、ggplot(data_ggp, aes(x = NMDS1, y = NMDS2)) + geom_point(size = 3) があれば大丈夫です。
プロットデータを p に保管しておきます。
#35 assign(paste0("p", i), p) で、プロットデータを保管するための「箱」の名前を変更します。
assign() は、データを代入するための関数です。
たとえば、次のように使います。
assign("data",1:10)
data
[1] 1 2 3 4 5 6 7 8 9 10assign("data",1:10) とすると、 data のなかに 1, 2, 3, … , 10 が代入されます。つまり data <- 1:10 と同じです。
paste0() は、独立したデータを連結するための関数です。
たとえば、次のように使います。
paste0("C", 3, "PO")
[1] "C3PO"assign(paste0("p", i), p) とすることで、i = 1のときには p1、i = 2のときには p2 という名前の箱が作成され、
それぞれの箱にプロットデータを格納しておくことが可能です。
なぜこのような作業が必要かというと、
p <- ggplot(data_ggp, aes(x = NMDS1, y = NMDS2)) + geom_point(size = 3) のままだと、
i回目に生成したプロットデータが i+1回目のコードを実行した際に上書きされます。
for文が完結したときには、最後に生成したプロットデータのみが p に残されていることでしょう。
試行ごとに、独立した箱にプロットデータを格納しておけば、gridExtra::grid.arrange(p1, p2, p3, ncol = 2, nrow = 2) で図を並べることができます。
※注意
paste0() と paste() は似て非なるものなので注意!
詳細については、以下のサイトが参考になります。
おまけ(その1)
ここまで書いて、カバーイラストで示したプロットの作成方法を紹介していないことに気づきました。
詐欺はダメなので一応コードを貼っておきます。
spe <- unique(data$Species)
for (j in 1:length(spe)) {
species <- spe[j]
data_spe <- filter(data, Species == species)
p_point <- ggplot(data_spe, aes(x = Fish.Length..Mm, y = Fish.Mass.G)) +
geom_point(size = 3) +
theme_light() +
theme(panel.grid=element_blank())
assign(paste0("pp", j), p_point)
p_box <- ggplot(data_spe, aes(x = Stream, y = Fish.Mass.G)) +
geom_boxplot()
assign(paste0("pb", j), p_box)
}
gridExtra::grid.arrange(pp1, pp2, pp3, pp4, pp5,
pb1, pb2, pb3, pb4, pb5,
ncol = 5, nrow = 2)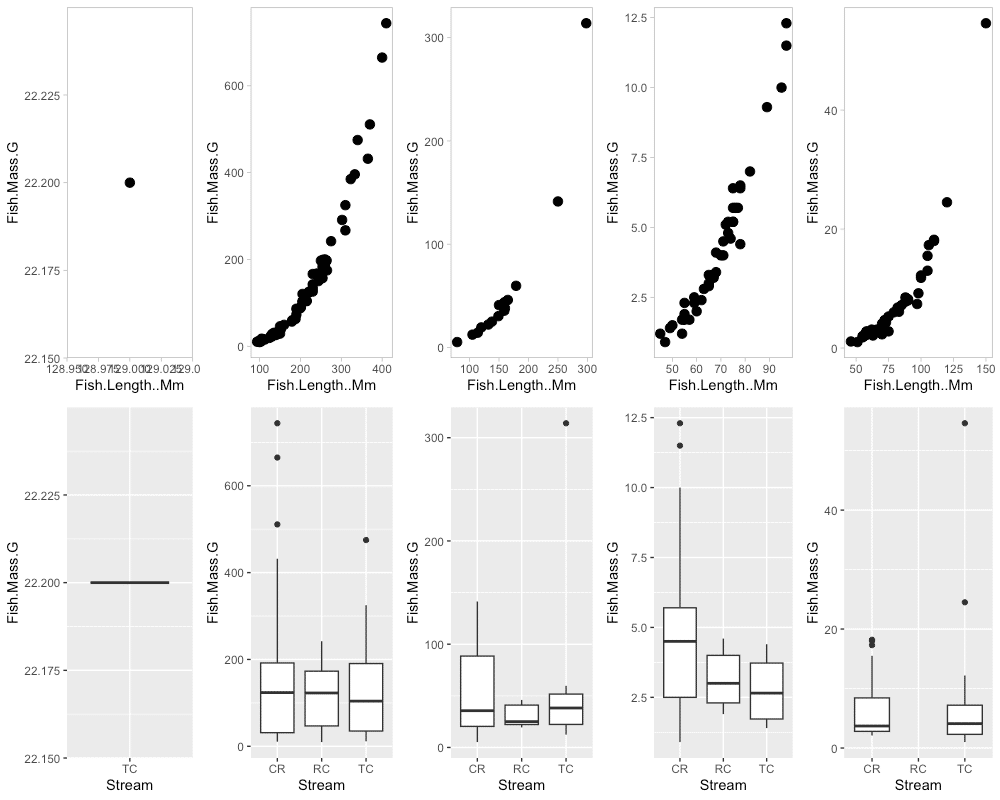
おまけ(その2)
魚種ごと・調査地点ごとに胃内容物組成の特徴をnMDSする方法も紹介します。
str <- unique(data$Stream)
spe <- unique(data$Species)
plots <- list()
n <- 1
for (i in 1:length(str)) {
site <- str[i]
data_str <- filter(data, Stream == site)
for (j in 2:length(spe)) {
species <- spe[j]
data_spe <- filter(data_str, Species == species)
data_comm <- select(data_spe, 9:28)
eudist <- vegdist(data_comm, "euclidean")
eumds <- metaMDS(eudist, k=2)
data_ggp <- as.data.frame(scores(eumds))
plots[[n]] <- ggplot(data_ggp, aes(x = NMDS1, y = NMDS2)) +
geom_point(size = 3) +
theme_light() +
theme(panel.grid=element_blank()) +
ggtitle(paste0("site", i, "species", j))
assign(paste0("site", i, "species", j), p)
n <- n + 1
}
}
all <- c(plots, list(ncol=4, nrow=3))
do.call(grid.arrange, all)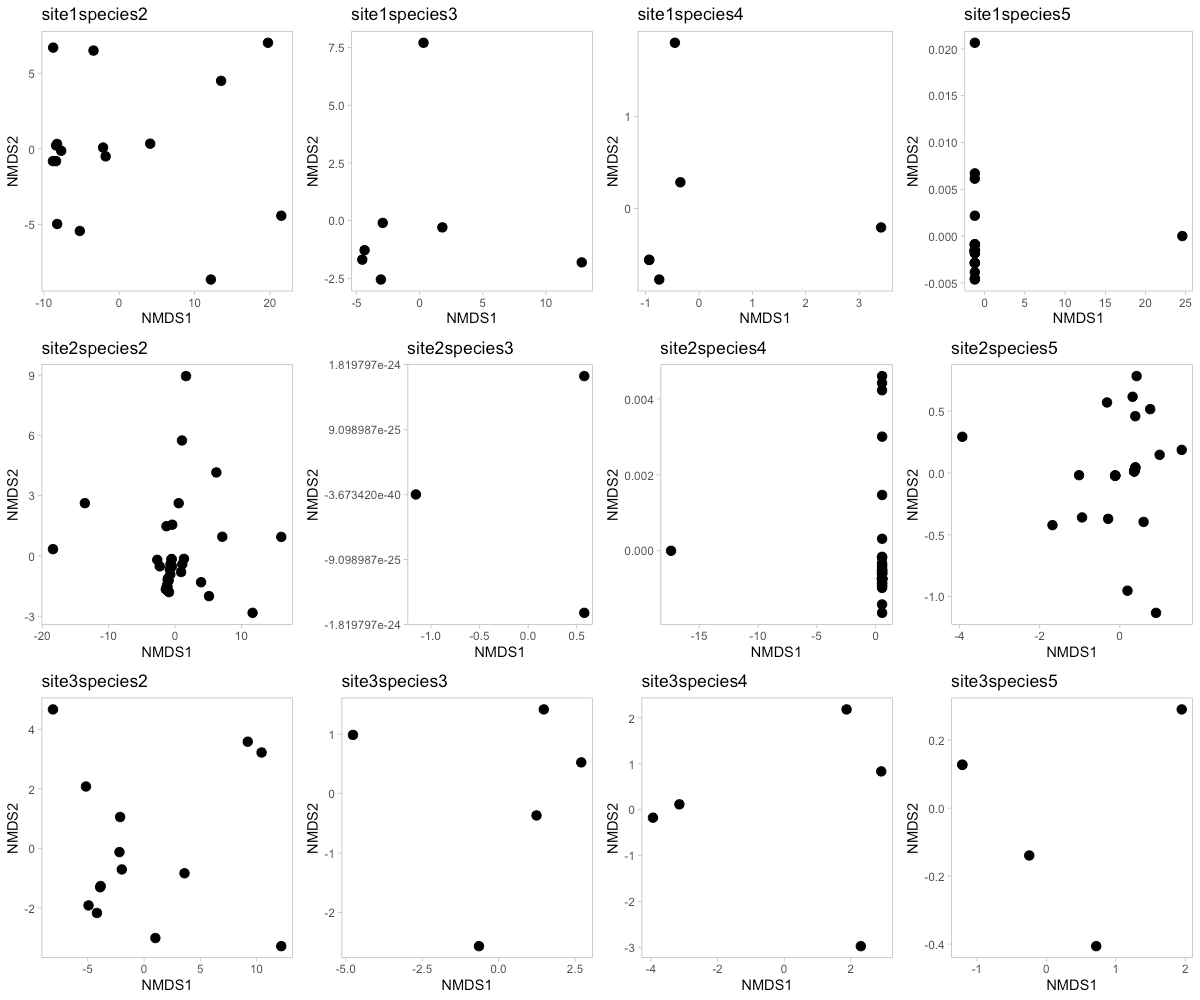
もはや gridExtra::grid.arrange() で各プロットのデータ(オブジェクト)を手入力することすらめんどくさくなってしまったので、
プロットデータを保管するためのlistを作成しました。
※生データではSpeciesが5種類含まれていましたが、Species1はデータ数が少なくユークリッド距離の計算をおこなえなかったため、除外しました。
そういうわけで、for (j in 2:length(spe)) としています。
※ユークリッド距離やnMDS解析の妥当性は確認していません。
※細かい体裁は整えていません。
例のごとく長い記事になってしまいましたが、今回はここまでです。
リクエストやお困りのことがあれば、コメント欄にてお待ちしております。
この記事が気に入ったらサポートをしてみませんか?
