
Pythonを使ってCOVID-19感染者データを加工してみる-part1
2020年冬から世界に多大な影響を与え続けているCOVID-19ですが、日本でも緊急事態宣言を始め、多くの人々に行動の自粛が求められる状況は依然として続いています。日本のCOVID-19感染者情報はいくつかのサイトでわかりやすくまとめられていて、気軽に私達がアクセスして状況の確認をすることができます。ですが、私個人としては生データを自分でグラフ化して傾向を確認したい、という欲求があり、Pythonで感染者データを自分のほしい形に加工しようと思い至りました。今回は、私自身初めてまともにpandasを使用してデータ加工を行ったこともあり、私と同じようにCOVID-19感染者データを加工したいと望む方々に向けて、スクリプトの詳細を発信してみたいと思います。
1.目標
今回は、色々なサイトでよく見かける、各都道府県別に一日の感染者数をプロットするために必要なデータを作ってみたいと思います。下記のようなグラフが作成できるようにしていきます。

2.環境
python 3 . 7 . 1、jupyter notebook (jupyter lab)
私は基本的にターミナルからスクリプトを実行しますが、pythonに初めて触れる方はAnacondaをインストールして、jupyter notebookから実行するのがわかりやすいと思います。またpython2以前では、若干仕様が違うので、コピペでは動作しないかもしれません。
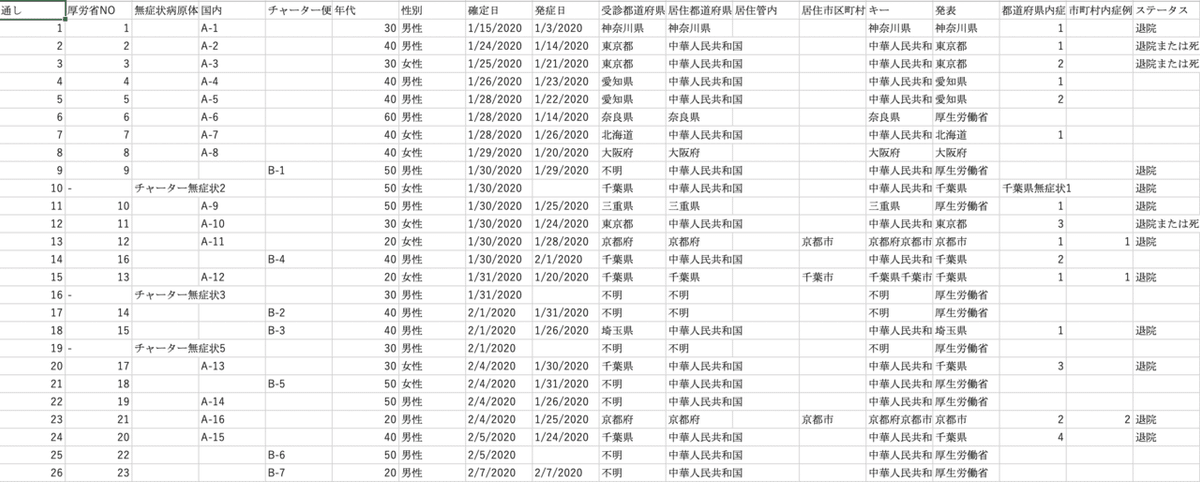
3.生データ
生データは「都道府県別新型コロナウィルス感染者数マップ」で公開されているCSVファイルを使用させていただきたいと思います。こちらのサイトでは、2020年1月から今までの日本におけるCOVID-19感染者に関する全てのデータがまとめられたCSVファイルを公開されています。
このCSVファイルでは、下記のように1行ごとに一人の感染者情報がまとめられているため、一日あたりの都道府県別の感染者データを得るためには少しデータの加工が必要です。
4.スクリプトの詳細〜データの読み込みと感染者数合計の計算〜
ここでは埋め込んであるコードを上から順にコピペすれば動作するようにしていくつもりです。まずライブラリを読み込みます。いずれのライブラリもあとで使用します。
import pandas as pd
import datetimeまずはcsvファイルを読み込み、DataFrame型として扱える状態にしましょう。csvファイルは「COVID-19.csv」という名前でスクリプトファイルと同じフォルダに入れます。データから「確定日」と「Hospital Pref」を抽出し、それを日付、都道府県ごとにソートを掛けて、「特定の日に特定の都道府県で出た感染者数の合計」を計算します。ソートを掛けた項目ごとの合計は、groupby().size()とすることで簡単に計算できます。
csv_data = pd.read_csv('./COVID-19.csv', encoding="utf_8")
series = csv_data[['確定日','Hospital Pref']]
series_freq = series.groupby(['確定日', 'Hospital Pref']).size()series_freqの出力結果は下記のようになります。一番右端は、groupby().size()で計算した項目ごとの感染者数の合計値になります。

ここから、抽出したデータをさらに加工していきます。データ加工の際、DataFrame型でデータを扱いたいのでデータ型を変更します。pd.read_csv()を使用してデータを読み込んだ場合、series型でデータが格納されてしまいます。dataframe型としてデータを扱いたいので、series型 → list型(tolist())、list型 → dataframe型(dataframe())と2段階でデータ型を変更しています。(もう少しスマートに上記処理を実行する方法をご存じの方がいらっしゃればぜひ教えて下さい。)
#seriesをlistに変更
l_2d = series_freq.reset_index().values.tolist()
#listをdataframeに変更、加えてcolumn indexも任意の名前に変更
df = pd.DataFrame(l_2d, columns=['Date','Region','infected'])あまり多くの都道府県のデータがあっても扱いづらいので、最新の日付で見たときに感染者数が多い都道府県の内、上位5位までを抽出しようと思います。また「最新の日付でのデータ」と記載していますが、このCSVファイル中ではデータの更新日が都道府県ごとに差異があるので本当に最新のデータで比較してしまうと、うまくデータが比較できないことがあります。そこで数日遡った日付で比較できるようにdate_deltaを設定しています。この際に、最新の日付から何日遡るかを計算する必要があるため、datetimeを使用して直感的に日付を引き算できるようにします。
from_d = datetime.datetime(2020,1,1)
tday = datetime.datetime.now()
tdelta = datetime.timedelta(days=1)
date_delta = 10 #どのくらい前のデータを参考にして上位5つの都道府県を決めるかのファクター
#Dateの大小比較をするために、データ型を変更
df['Date'] = pd.to_datetime(df['Date'])
#最新の日付を取得し、そこからdete_deltaだけ日付を遡る
date_max = df['Date'].max()
date_max_delta = date_max - tdelta * date_deltaquery()を使用して、特定の日付のデータのみを抽出します。さらにそのデータを感染者数の多い順に並べ替え、上位5つの都道府県を抽出し、その都道府県名をl_2d_regionに格納していきます。このとき、list型で格納しておくと後の処理がやりやすくなるのでtolist()でlist型に変更しています。
#queryを用いて最も新しい日付からdate_deltaだけ遡った日付のデータのみを抽出
df_query_date = df.query('Date == @date_max_delta')
#以下では、最新の日付(- date_delta)で感染者数の多い、上位5つの都道府県名を抽出
df_sort = df_query_date.sort_values('infected', ascending=False)
df_reindex = df_sort.reset_index()
df_slice = df_reindex.loc[0:4]
df_slice2 = df_slice['Region']
l_2d_region = df_slice2.reset_index().values.tolist()df_slice2とl_2d_regionの出力結果は下記のようになります。これで感染者数の多い上位5つの都道府県の名前を格納したリストができました。

今回はここまでにしたいと思います。近日中に続きをアップロードする予定ですが、次回は出力用のリストを作成し、実際にデータを出力してみます。
この記事が気に入ったらサポートをしてみませんか?
