OLS回帰 ? LAD回帰?
よく統計学の本で回帰分析を勉強する際に,「最小二乗法(OLS)」とともに,最小絶対偏差法(最小絶対値法?, LAD?)というものも同時に紹介されているかと思います.正直,統計レベル弱々の私には違いがなんとなくしかわかりません.
ということで,最小絶対偏差法を導入するコードを書き,OLSとLAD? の違いを整理してみました.
(もしかしたら,最小絶対偏差法のことをLADと呼ばないかもしれません.数学的定義は下に書いておきますが用語の使い方はガバガバなので,この記事のみを鵜呑みにしないようにお願いします)
1. 最小二乗法
定義は以下の通り.

残差二乗(誤差の予測値の二乗)を最小にする方法.それ以上の説明は統計学の教科書や計量経済学の教科書を見ていただけるといいかもしれません.
1.1 シミュレーション
回帰分析をするモチベーションとして,本当は回帰係数を知るために行うと思いますが,今回はOLSとLADの方法の違いによってどのような結果の違いが生まれるかを確認していきたいです.
よって,先にパラメタを決めてしまいましょう !

上に従うと仮定して,データをランダムに生成しましょう.
set.seed(1024)
N <- 100
X <- runif(N, min = -5, max = 5)
e <- rnorm(N, mean = 0, sd = 0.1)
Y <- 1 + 2 * X + e
sim_abs <- tibble(Y, X)これで,ランダムに1サンプル分のデータを生成することができました.
このデータを元に,回帰分析を行ってみましょう.
lm()を使っても良いのですが,optim()という関数を使ってみます.
( (Sho Kuroda)「Rで推定する回帰モデル入門」を元に書いています)
ols <- function(y_ols, x_ols){ ## 引数を決める
min_rss <- function(param){ ## 最小化問題を設定
sum((y_ols - x_ols %*% param)^2)
}
k <- ncol(x_ols)
return(optim(par = rep(0, k), fn = min_rss))
}
y <- sim_abs$Y
x <- cbind(1, sim_abs$X)
ols(y_ols = y, x_ols = x)結果を確認しましょう.
$par
[1] 0.9867376 1.9979531
$value
[1] 1.024894
$counts
function gradient
59 NA
$convergence
[1] 0
$message
NULL上の$par[1]がαの値で,$par[2]がβの値のように見えます.
ここで一応,lm関数を使って推定しておきましょう.
res_ols1 <- lm(data = sim_abs, Y ~ X)
#knitr::kable(broom::tidy(res_ols1)) ##PDFでknitするためのコード(無視でお願いします)
summary(res_ols1)結果を確認します.
Call:
lm(formula = Y ~ X, data = sim_abs)
Residuals:
Min 1Q Median 3Q Max
-0.31680 -0.05753 -0.00840 0.06266 0.27432
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.986561 0.010227 96.47 <2e-16
X 1.998001 0.003566 560.25 <2e-16
Residual standard error: 0.1023 on 98 degrees of freedom
Multiple R-squared: 0.9997, Adjusted R-squared: 0.9997
F-statistic: 3.139e+05 on 1 and 98 DF, p-value: < 2.2e-16optim()の結果とlm()の結果が限りなく近い値を取っているので,推定に成功していると言えそうです.
これだけでは何もわからないので,今度は1000回ほど回帰分析をしてみましょう.function()で関数を作っちゃいます.
(矢内勇生先生のサイトを参考に書いています.ill-identified氏からもTokyo.Rのslack上で意見をいただきながら,コードを書いております.お二方には本当に脱帽です..)
sim_normal_ols <- function(N = 100, b = 2, alpha = 1, sd1 = 0.1, mean1 = 0){
X <- runif(N, min = -5, max = 5) ##説明変数
e1 <- rnorm(N, mean = mean1, sd = sd1) ##誤差(sdとmeanを自分で設定)
Y <- alpha + b * X + e1 ##真のモデル
df <- tibble(Y, X) ##データフレーム化
models <- tibble(model = "normal", formula = list(Y ~ X)) %>%
mutate(fit = map(formula, ~lm(.x, data = df))) ##モデルの決定
models %>%
mutate(coefs = map(fit, ~tidy(.x) %>% filter(term == "X"))) %>%
unnest(coefs) %>% dplyr::select(model, estimate, std.error)
}
##コードのインデントが汚いです.エラーになるかもしれません.上のコードによって,ランダムにデータを発生させ回帰分析を行い,その時の回帰係数を数字として保存してくれるfunctionが書けました !
同じようなデータを1000個発生させ,回帰分析を1000回行ってみましょう!
n_trials <- 1000 ##回帰分析を行う回数
sim_normal_beta <- map_dfr(1:n_trials, ~ sim_normal_ols() %>% mutate(sim_id = .x))
g1 <- ggplot(sim_normal_beta, aes(x = estimate, y = after_stat(density))) +
geom_histogram(color = "black", fill = "dodgerblue")
print(g1)結果は以下のようなグラフになります.

少々みづらいですが,縦軸が密度・横軸が推定値となっています.
やはり回帰係数の分布はt分布(もしくは標準正規分布)となっています.
係数の平均値を取ってみましょう.
mean(sim_normal_beta$estimate)[1] 1.999958母数(母集団が持つ真の特徴)に限りなく近似していることがわかります.
OLSのシミュレーションはこんなところでしょうか ?
2. 絶対最小偏差法
数学的な定義は以下の通りです.

数式の解釈としては,「応答変数と(回帰係数 × 説明変数 + 切片)の差を残差(誤差の推定値)と定義し,個体iからnまでの残差の絶対値の和をパラメータβ, αを動かすことで最小にできると仮定した時,最小になった時のパラメタの値を求めよ」という問題というところでしょうか?
OLSで使った

を母数として,ランダムに発生させたデータを使って一回分析を行ってみましょう.
lad <- function(y_lad, x_lad){
#y_ladは非説明変数ベクトル
#x_ladは説明変数ベクトル
min_lad <- function(param){
sum(abs(y_lad - x_lad %*% param))
}
k <- ncol(x_lad)
return(optim(par = rep( 0, k), fn = min_lad))
}
lad(y_lad = y, x_lad = x)結果は以下の通りになります.
$par
[1] 0.9791139 1.9966752
$value
[1] 7.722072
$counts
function gradient
149 NA
$convergence
[1] 0
$message
NULLまた,違うパッケージを用いて分析を行ってみましょう.
quantreg::rq(formula = Y ~ X, data = sim_abs) ##最小絶対偏差法による回帰Call:
quantreg::rq(formula = Y ~ X, data = sim_abs)
Coefficients:
(Intercept) X
0.9791163 1.9966765
Degrees of freedom: 100 total; 98 residualこちらもoptim()の結果に限りなく近似しています.
OLSとの違いは,まだはっきりと見えてきません.
ということで,1000回ほどLAD回帰を行ってみましょう.
set.seed(1024)
sim_normal_lad <- function(N = 100, b = 2, alpha = 1, sd1 = 0.1, mean1 = 0){
X <- runif(N, min = -5, max = 5) ##説明変数
e1 <- rnorm(N, mean = mean1, sd = sd1) ##誤差(sdとmeanを自分で設定)
Y <- alpha + b * X + e1 ##真のモデル
df <- tibble(Y, X) ##データフレーム化
models <- tibble(model = "normal", formula = list(Y ~ X)) %>%
mutate(fit = map(formula, ~quantreg::rq(.x, data = df)))##モデルの決定
models %>% mutate(coefs = map(fit, ~tidy(.x) %>% filter(term == "X"))) %>%
unnest(coefs) %>% dplyr::select(model, estimate)
}
n_trials <- 1000
sim_normal_beta_lad <- map_dfr(1:n_trials, ~ sim_normal_lad() %>% mutate(sim_id = .x))
g3 <- ggplot(sim_normal_beta_lad, aes(x = estimate, y = after_stat(density))) +
geom_histogram(color = "black", fill = "dodgerblue")
plot(g3)回帰係数の確立密度の分布は以下のようなものになります.

数字が相当見えづらいですが,標準誤差はOLSよりも大きくなっていそうです.(ここからもOLSはBLUEであるというのが,なんとなく掴めるかと思います)
ですが,期待値は2.00の付近を取っているようにも見えます.
今一度,両者の分布を比較してみましょう.
g4 <- ggplot() +
geom_density(data = sim_normal_beta_lad, aes(x = estimate, y = after_stat(density))) +
geom_density(data = sim_normal_beta, aes(x = estimate, y = after_stat(density)), color = "purple")
plot(g4)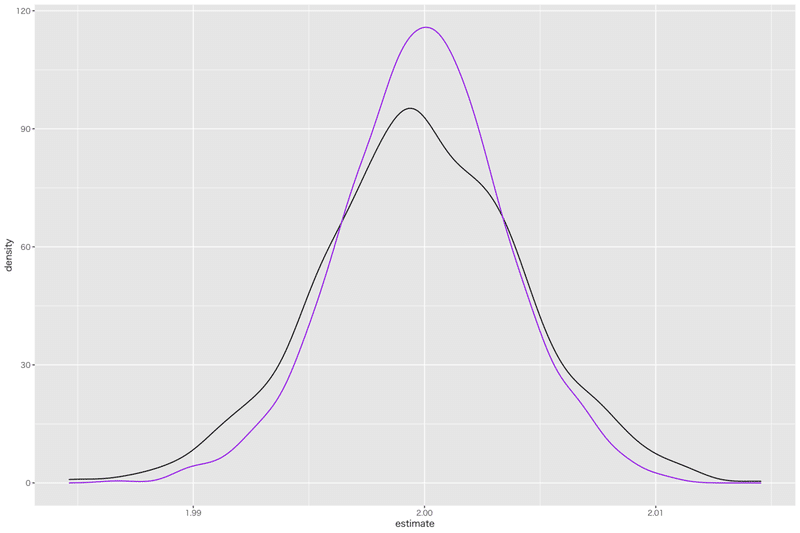
紫色の線がOLS,黒色の線がLADの線です.
分布の分散はLADの方が大きいものとなっていることは明白でしょう.
(あれ,ではLADはどういう時に使われるのでしょうか?)
3. 外れ値の影響を分析
次に,一つのデータセットを用いて外れ値の分析を行っていきます.
まずは97個のデータをランダムに発生させ,さらに3個ほど異常な値を発生させ,計100個のサンプルサイズのデータを作ります.
このデータの一つであるi番目の行のデータをわざと欠損させ,残りの99個のデータセットで回帰分析を行い,回帰係数を記録します.
これを任意のiが1 ~ 100をとるまで100回繰り返し,回帰係数のブレを確認します.
早速データを作ってしまいます.
以下の式を母集団の特徴として,97個データセットを作り,残り3個のデータは恣意的に異常値となるようにデータを作成します.

set.seed(1024)
N <- 97
X <- runif(N, min = -5, max = 5)
e <- rnorm(N, mean = 0, sd = 0.1)
Y <- 1 + 2 * X + e
sim_2 <- tibble(Y, X)
X <- c(5, 4, 5) ##外れ値のXの値
Y <- c(20, 21, 30)##外れ値のYの値
out <- tibble(X, Y)##外れ値のデータセット
sim_3 <- rbind(sim_2, out)##ランダムデータと外れ値データを結合(一様分布を明示するときって,Uniform(min, Max)っていう書き方でよかったんでしたっけ ? 数式で明示する機会があまりないので,うろ覚えです..)
どんな散布図になるのでしょうか ? 回帰直線と一緒に散布図もplotしてみます.
g5 <- ggplot(sim_3, aes(x = X, y = Y)) +
geom_smooth(method = "lm") +
geom_point()
plot(g5)
横軸xが5のときにあからさまに変な値が3つ取られているのが確認でき,回帰直線も,それに引っ張られています.
係数の値も確認してみましょう.
res_ols2 <- lm(Y ~ X, data = sim_3)
#gr1 <- knitr::kable(tidy(res_ols2))
summary(res_ols2)Call:
lm(formula = Y ~ X, data = sim_3)
Residuals:
Min 1Q Median 3Q Max
-1.4289 -0.8661 -0.2682 0.1720 17.5786
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.36876 0.23329 5.867 6.02e-08
X 2.21053 0.07886 28.031 < 2e-16
Residual standard error: 2.33 on 98 degrees of freedom
Multiple R-squared: 0.8891, Adjusted R-squared: 0.888
F-statistic: 785.8 on 1 and 98 DF, p-value: < 2.2e-16Estimateを確認するとわかると思いますが,説明変数にかかる回帰係数は約2.21と母集団よりも大きな値を取っています.
それでは,LAD回帰をしてみるとどうなのでしょうか ?
res_lad <- quantreg::rq(Y ~ X, data = sim_3)
#knitr::kable(tidy(res_lad))
summary(res_lad)Call: quantreg::rq(formula = Y ~ X, data = sim_3)
tau: [1] 0.5
Coefficients:
coefficients lower bd upper bd
(Intercept) 1.00493 0.98877 1.01777
X 2.00134 1.99883 2.00688 OLSの結果と比較すると,素晴らしいくらいに母数である2に近似しています.これが,ロバスト回帰とも言われている由縁でしょうか?
それでは,本題の「観測値の任意の値を一つ欠損にし,残り99個で回帰分析を行ったときの係数の分布」を確認してみましょう.
まずはOLSから
na_vec <- tibble(rep(0, 100)) ##0のベクトルを作成
for (i in 1:100) {
k <- sim_3[i,2] ##kにi行2列目の値を代入
sim_3[i, 2] <- NA ##i行2列目を欠損させる
sim_res <- lm(Y ~ X, data = sim_3, na.omit = TRUE) ##欠損したまま回帰
na_vec[i,1] <- sim_res$coefficients[[2]] ##0のベクトルに回帰係数を代入
sim_3[i, 2] <- k ##欠損部分にもとの値を代入
}
na_vec <- na_vec %>%
mutate(beta = na_vec$`rep(0, 100)`)#名前が面倒くさいので変更結果を確認していきます.
g7 <- ggplot(na_vec, aes(x = beta, y = after_stat(density))) +
geom_histogram(color = "black", fill = "dodgerblue", binwidth = 0.0001) +
labs(x ="サンプルサイズが99のときの回帰係数", y = "確率密度")
print(g7)
g8 <- ggplot(na_vec, aes(x = beta, y = after_stat(density))) +
geom_density()
print(g8)
思った通り,係数は母数の2.0付近を全く通っておらず,基本的には2.2よりも少し大きな値を取っているようです.
異常値(外れ値)が欠損になると,2.16くらいの値を取っていますが,基本的には異常値に引っ張られていることが伺えます.
次にLAD回帰を分析してみましょう.
na_vec_2 <- tibble(rep(0, 100)) ##0のベクトルを作成
for (i in 1:100) {
k <- sim_3[i,2] ##kにi行2列目の値を代入
sim_3[i, 2] <- NA ##i行2列目を欠損させる
sim_res2 <- quantreg::rq(Y ~ X, data = sim_3, na.action = na.omit) ##欠損したまま回帰
na_vec_2[i,1] <- sim_res$coefficients[[2]] ##0のベクトルに回帰係数を代入
sim_3[i, 2] <- k ##欠損部分にもとの値を代入
}
na_vec_2 <- na_vec_2 %>%
mutate(beta_lad = na_vec_2$`rep(0, 100)`)#名前が面倒くさいので変更結果を確認してみます.
g9 <- ggplot(na_vec_2, aes(x = beta_lad, y = after_stat(density))) +
geom_histogram(color = "black", fill = "dodgerblue", binwidth = 0.0000000000001) +
labs(x ="サンプルサイズが99のときの回帰係数", y = "確率密度")
print(g9)
どうやら,サンプルの一つを欠損値に変えて分析しても,係数の値に変化はないようです.(これ,コードミスくさい感じもしますが...)
しかしこれも,「OLSよりは異常値にひきづられていない」というだけで,真の値である2.0は取っていません.
ここから,外れ値の影響こそ受けないが,同様に「真の値」を補足することもなさそうです.
単にOLSの値のみを確認するのではなく,外れ値に影響されづらい分析方法で分析してみたいという時にLAD回帰の価値が出てくるというふうに考えることができそうです.
終わりに
ここまで時間をかけて記事を作っておいてあれですが,「99.9%の確率で示唆のどこかしらに間違いがある」と思っていただけたらと思います.
数学的にOLSとLADの違いを解き明かすのではなく,シミュレーションによって違いをみているため,「コードが間違っていたら,妥当な結論を得ることはできません」が,私のR LevelはLv. 5で,まだまだひよっこです.
もし,間違いを見つけていただけたらTwitterやnoteでツッコミをしていただけたらと思います.
参考文献
(Sho Kuroda)「Rで推定する回帰モデル入門」(2021/ 1/1 最終閲覧)
(矢内勇生)「KUT 計量経済学応用 Topic 4 回帰分析」(2021/ 1/1 最終閲覧)
この記事が気に入ったらサポートをしてみませんか?
