
機械学習〜パラメータのチューニングなどなど。
タイトルで区切ってますので、みたいところだけ見れるようにしてます。(つもり)
(見たいものが見れないなと感じたら、3分割とか、5分割とかにしようと思います)
ーーーーーーーーーーーーーーーーーーーー
・イントロ
今回のテーマは
モデルの評価とハイパーパラメータのチューニング
です。
その中でも機械学習のアルゴリズムをチューニングしていき、モデルを評価していくことで、より適したモデルを構築することができます
つまりつまりつまり、、、
技のコンボによってより強力な必殺技ができるから、技の連携とかみていくといいよね(例えば、スマブラのコンボ技みたいな?)って感じです。
・今回のテーマ
今回用いるのはsklearnのpipelineにあるmake_pipelineについてみていきます。
このmake_pipelineはなにをしているのかといえば、複数のモデルを結合して一連の処理を行うことが可能になる関数です。(詳細な使い方は後ほど)
・おおまかな準備
・今回使うデータとちょっと探索
今回は参考書籍がありますため、それに準ずる形で進めていきます
データも同様で、わりかし定番であるBreast Cancer Wisconsinデータセットを用います。(めっちゃ古いデータ使うのやめてほしい。。)
このデータは悪性と良性の細胞の合計569個のデータセットで構成されていますが、先にデータをみておきます。
- Pythonのコード
import pandas as pd
df = pd.read_csv('https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/wdbc.data', header=None)今回はなるべく詳細な解説はしますが、一定の部分(import とは?みたいな説明やread_csvとは?みたいな説明)に関しては省略させていただきます。。
実際にデータを上から少しみてみます。
df.head()
(Columnの名前が連番なの、なんで?ってなりますが、sklearn.datasetsにあるものを参照すると、以下。)
feature_names = np.array(['mean radius', 'mean texture',
'mean perimeter', 'mean area',
'mean smoothness', 'mean compactness',
'mean concavity', 'mean concave points',
'mean symmetry', 'mean fractal dimension',
'radius error', 'texture error',
'perimeter error', 'area error',
'smoothness error', 'compactness error',
'concavity error', 'concave points error',
'symmetry error', 'fractal dimension error',
'worst radius', 'worst texture',
'worst perimeter', 'worst area',
'worst smoothness', 'worst compactness',
'worst concavity', 'worst concave points',
'worst symmetry', 'worst fractal dimension'])
今回は1列目がtarget(予測したいもの。目的変数)だと思われるので、とりあえず種類とその数を確認しておきましょう。
df[1].value_counts()
ふむふむ。。
M(maligant: 悪性)とB(benign:良性)は半々ではなくB多め、ってことがわかりました。
(ちなみに、数はいらないよ!って場合はdf[1].unique()でおけです)
また、0列目に関してはIDなので、今回は無視して他の列を利用します。
・sklearnのLabelEncoderを使ってエンコーディング
少し詳細な説明を加えながら本題に入っていきます。
現在のデータフレームを目的変数(=df[1])と説明変数に分けて、目的変数のB, Mをコンピュータが判断できるように0, 1にラベルをつけていきます(これをエンコーディングと言います。代表的なものはOne-Hot Encoding)
今回はsklearnのpreprocessingにあるLabelEncodingを利用します。
(ちなみに、今回は0,1なのでOne-hotと区別しにくいですが、One-hotとLabelEncodingには違いがあります。
こちらの記事が参考になりました。)
from sklearn.preprocessing import LabelEncoder
# 説明変数を定義
X = df.iloc[:, 2:].values
# 目的変数
y = df[1]
# modelの定義
le = LabelEncoder()
# fit_transformで学習と最適な形に変形する処理を同時に行う
y = le.fit_transform(y)ちなみに、sklearnはarray(いわゆるベクトル)の形で処理するのが好ましく、基本的にはDataFrame→array(.valuesなどで処理)するのですが、わりとDataFrameのまま処理してくれることが多いため、エラーが出てから判断するとかでも個人的にはいいと思っています(※素人考えです)
・----ちょっと脱線-----
ちなみに、エンコーディングにはpandasのget_dummiesもありますが、get_dummiesはデータフレームになって返ってきます。
自分もこの辺が怪しかったので、
①df[1]のまま
②df[1].valuesにしておいた
③pd.get_dummiesを利用した
という3種類の方法をそれぞれ試してみてどう違うのかをみてみました。
# modelnの定義
le = LabelEncoder()
# fit_transformで学習と最適な形に変形する処理を同時に行う
y_le = le.fit_transform(y)
y_arr = y.values
y_arr_le = le.fit_transform(y_arr)
y_get_dummies = pd.get_dummies(y)
print(y_le)
print('--')
print(y_arr_le)
print('--')
# 何かしらに違いがあるかを確認(ちょっと可読性低め)
print((y_le == y_arr_le).sum() - len(y_le))
print('--')
print(y_get_dummies)
めちゃくちゃ見えにくいと思うので、簡潔に説明しておくと、
df[1]とdf[1].valuesに差はない(今回のように1列で処理した場合)
pd.get_dummiesを利用するとカラムが作成されてそれぞれの列に0, 1が入る
って感じでした。
・準備(train_test_splitで分割)
エンコーディング処理が完了したため、先にモデルを訓練用とテスト用に分類しておきます。
(最初は簡単にmake_pipelineを利用したいため、これを使いますが、この後k分割検証法にてちゃんと訓練します。k分割検証法については後ほど解説)
訓練用とテスト用に分類するのは定番のtrain_test_splitを利用します。
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, stratify=y, random_state=1)ここで、train_test_splitの引数を解説します。(公式ドキュメントを見るに越したことはありませんが、英語苦手な方もいると思いますので、、)
sklearn.model_selection.train_test_split(*arrays, test_size=None, train_size=None, random_state=None, shuffle=True, stratify=None)
*arrays: 基本的には分割したいデータフレームや配列を入れます。(今回で言えば、X, yが該当)
test_size: テストサイズと訓練サイズをそれぞれ何:何に分けるかを定義します(小数で指定。今回は0.2なので、2:8に分割。一般的には0.3や0.2くらい)
random_state: 数値を指定することにより、決められたrandom_seed(いわゆる決め打ち。ランダムとか言いながら八百長してる感じ)で振り分けてくれます
(教科書などでは誰でも同じ結果になるように何かしらの数値を指定しています。)
shuffle: 分類する前にデータをシャッフルして分割させます。ただし、stratifyの引数を指定する場合は必ずNone(デフォルト)のままにしておく必要があります。
stratify: データを分割した際、偏りが生じることもあります。
(今回の例で言えば、B(良性)の数が少ないため、訓練用データにばっかりBが入って、テストデータで検証した際に全然だめ、みたいなことが起こりかねません。
逆に、テストデータにだけBが入っちゃって、訓練データにはMしかない、みたいなことにもなりかねません)
これを防ぐために、
stratify =(分割するデータのどれか)
を指定することで、そのデータの中身の比率に従ってうまく分割してくれます。
今回の例で言えば、(B:M = 357: 212なので、trainデータとtestデータの中身の比率も357:212になるようにうまく分類してくれます。)
・本題1
・make_pipelineを使う!
それでは、今回の最初の本題であるmake_pipelineを使ってみます
(ちなみに、自分も最近初めて使ってみただけなので、一緒に馴染んでいけたらなと思います。
また、基本はmake_pipelineではなく、sklearn.pipelin.pipelineを使う方がメジャーぽい)
まずは、現時点でdfの尺度はバラバラで単位が全く違うため標準化を行う必要があります。
さらに、今回は次元圧縮もしたいと想定してみます。(主成分分析を利用)
・次元圧縮: d次元のあるものをk次元(d >> k)に削減する方法
この背景には次元の呪いと言われるものがあり、たくさんの次元数(今回はcolumnの数)が多くなればなるほど、データの表現できる幅が非常に広くなりすぎて有限なデータでは訓練が追いつかないということが起こります。
そのため、次元を削減することにより学習の精度向上につながります。
・主成分分析: 次元削減の方法の一つ。説明変数をベクトルと見なしたとき、そのベクトルには固有値と固有ベクトルの大きい順にk個だけとってくる手法
(この辺はかなり数学理論の話になるので、詳細な説明は割愛しますが、
行列にはあるベクトルxと定数のλによってAx = λxと表現できるようなxとλがあり、これらを固有ベクトル、固有値と言います。
つまり、アイス好きのタカシ君を誰かに紹介する時、
「あいつはアイス(←固有ベクトル)でできてるんだよ!」みたいなことwww)
さて、毎度訓練用データやテストデータ、実際の取得したデータで毎度
標準化→主成分分析→ロジスティック回帰って行うのがめんどくさいので、make_pipelineの出番となります。
まずは、コードをみてみましょう。
from sklearn.decomposition import PCA
from sklearn.linear_model import LogisticRegression
from sklearn.preprocessing import StandardScaler
from sklearn.pipeline import make_pipeline
# 連結を行う。
# 処理したい順番に定義して、最後は必ず推定器を記載
# 最近LogisLogisticRegressionのsolver='lbfgs'がデフォルトになったため、実際には明示する必要はない
pipe_lr = make_pipeline(StandardScaler(),
PCA(n_components=2),
LogisticRegression(random_state=1, solver='lbfgs'))割と明快に記載されている気もしますが、解説します。
(公式ドキュメント)
sklearn.pipeline.make_pipeline(*steps, memory=None, verbose=False)
*steps: sklearnの変換器と推定器を取ることができます。変換器に関しては数の限界はなく、いくらでもOKです。また、fitメソッド、transformメソッドを実装していることが条件であり、推定器に関してはfit、predictメソッドがあることが前提となります。
memory: (使っている例をあまり知らないw)
joblibという並列処理が得意なライブラリが背景にあり、数値(もしくはstr)を指定することでfitしたtransformのpipelineのキャッシュを利用することが可能。
verbose: 引数に入れた処理にかかった時間が表示されるようになる(デフォルトはFalse)。重すぎる処理などをした際に、どこに時間がかかっているのかをみれるが、経験が浅いため利用しているコードをまだみたことがない
つまり、基本的にはパラメータは気にせず、変換器と推定器を入れちゃおう!って感じでOK(だと思います。)
このmake_pipelineの返り値はpipelineオブジェクトであり、.fit(学習)やpredict(予測)、.score(性能評価)などが使えます
pipe_lr.fit(X_train, y_train)
y_pred = pipe_lr.predict(X_test)
print(f'Accuracy score is {pipe_lr.score(X_test, y_test)}')
って感じで、精度的には95.6%でした
・make_pipelineの外観
さて、ざっと駆け足でみてきましたが、具体的にどのようなことが行われているのかといえば、
① スケーリング(StandardScaler)にてfit & transform
↓
②次元削減(PCA)にてfit & transform
↓
③学習と予測(LogisticRegression)のfit
テストデータにおいては
①、②でtransform
↓
③でpredict
って感じです。(以下が簡単なイメージ)

・本題2~ k分割交差検証によるモデル性能評価 ~
・イントロ
ここまではたくさんの工程を一括でまとめ上げて処理してくれるmake_pipelineによる威力やその内容を見てきました。
次に、k分割交差検証という手法についてみていきます。
この時点でかなり長文になっていますが、もう少しお付き合いください。。
さて、機械学習ではモデルの訓練時に「過学習」・「過小学習」という問題が起きることがあります。
過学習
訓練データに過剰に適合しすぎてしまい、テストデータや未知のデータ予測を大きく外してしまうことです。これはパラメータが多すぎて複雑なモデル構築をしてしまい、本番にうまく適応できない状況などが代表的です
(例えば、入試で赤本を丸暗記しただけで、本番では全く通用しない、みたいなw)
過小学習
過学習の反対で、モデル性能が簡単すぎるために、未知データをうまく予測できない状態
(学校の小テストだけやって、入試に挑む感じw)
この過学習と過小学習は別の側面からそれぞれ
「Varianceが高い」、「Biasが高い」とも言われます。
つまり、
過学習→分散(散らばり, variance)がありすぎてうまく学習できない
過小学習→偏見(bias)が偏りすぎてうまく学習できない
と言われてるわけですね。
こうした問題を起こさないために、モデル評価というものは入念にする必要があります。
そこでこれらを解決すべく
・ホールドアウト法
・k分割交差検証(KFold)
という手法について解説します。
これによって、過学習や過小学習を抑えてよりよいモデル性能を発揮できるようになります。
・ホールドアウト検証
先ほどの腫瘍の例でtrain_test_splitを使い、データを訓練データとテストデータに区切り、訓練データを学習し、テストデータでどのくらい予測できているか見てきました。
一般的に訓練データで学習して調整していきますが、その中でも学習前にあらかじめ決めるパラメータを変えてみることも大切となります。(これをモデル選択(※)と言います。)
※ 具体的にいえば、最適なハイパーパラメータ(学習前に人が決めるパラメータ)の最適な値を探すこと。今回の例ではPCAの中にあるn_components、決定木などのmax_depthなどが一例
しかし、一般的にこのtrain_test_splitのみに頼りきっていると、予期せぬ問題が起こります。
例えば、ある程度訓練データで学習が終わり、さぁ、テストデータだ!と意気込んで結果を見てみると過学習気味だったのか精度が良くなかったことが起こったとします。
そこで、「あ、過学習なのか!」と理解し、もう少しバラメータを調整し、
再度訓練データの学習
↓
テストデータで検証
↓
いやいや、まだ足りないな〜
↓
もっと訓練データの学習
↓
テストデータで検証
って感じで繰り返しちゃうケースがあります。
さて、ここで大きな問題が起きていることに気づきましたでしょうか??
「テストデータも訓練用のモデルに入っている」
ということです。
つまり、ハイパーパラメータのチューニングは本来訓練データのみで完結する必要があり、
そこにテストデータの結果を参照してしまうと、
「そのモデルは訓練データとテストデータに最適なモデル」といえ、
肝心の「未知データに対してうまく予測できているのか?」という課題から離れてしまっています。
モデル選択にテストデータを用いることは悪手であり、非常に危険です。
ここで、うまくホールドアウト法を使う方法として、
データをtrain, test, validation(訓練、テスト、検証)の3データに分割しておく方法があります。
訓練用で学習
↓
モデル選択のために検証用データでチューニング
↓
最後の最後にテストデータで性能判定を行う
という流れです。
簡単に図を用いると以下のようになります。
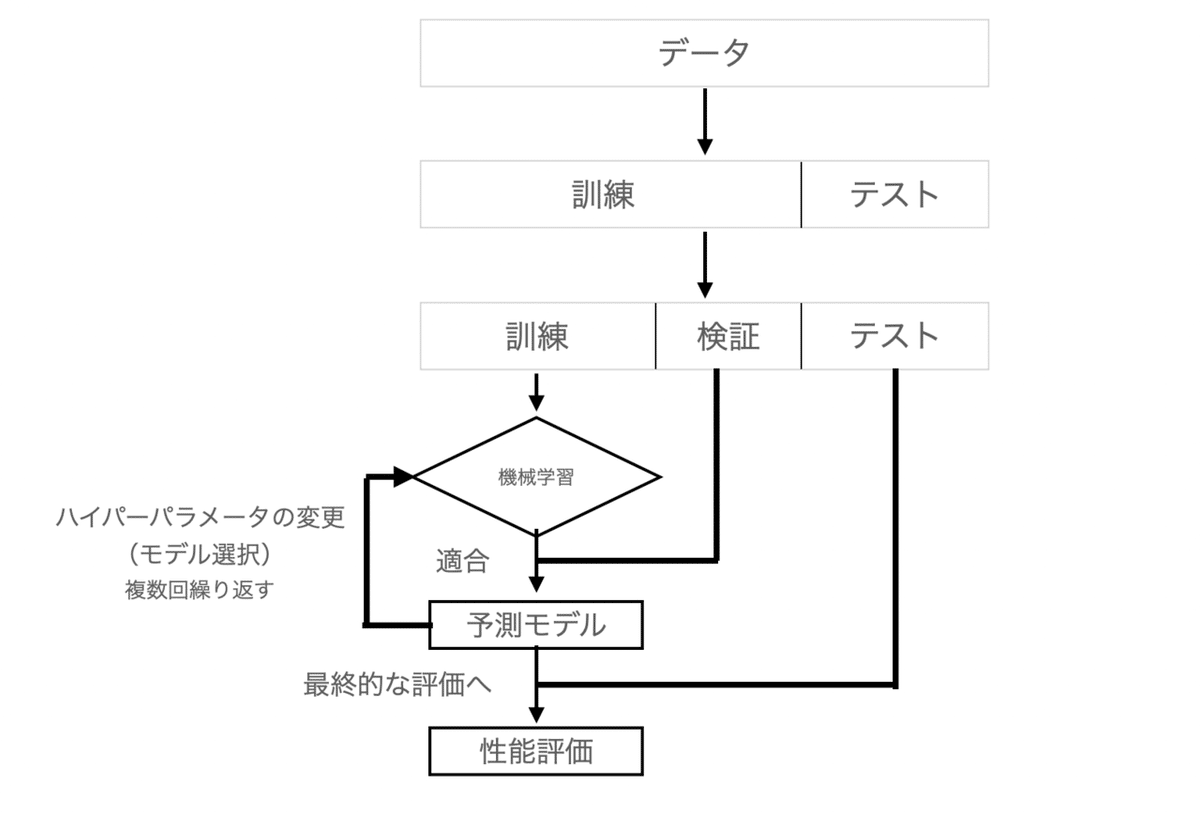
・ホールドアウト法の問題点
検証用の分割により問題は無くなったかと思いきや、ホールドアウト法には拭えない問題があります。
訓練データと検証データの分け方をどう分割するのかにより、モデル性能に影響が発生する場合場合があります。
どのように抽出するか?によって訓練用データや検証用データに偏りが生じる問題が残ってしまいます。
それらを解消した方法が本題のk分割交差検証(K-fold cross validation)という手法になります。
・k分割交差検証 (K-fold cross validation)
k分割交差検証はデータセットをk(これは自分で指定します)個に分割したのち、k-1個を訓練用、残りの1つをテスト用データに使用します。
これをk回繰り返すことで、モデルを選択して性能を評価します。
・k分割交差検証の何が良いのか?
先に、イメージ図を載せると以下のようになります。
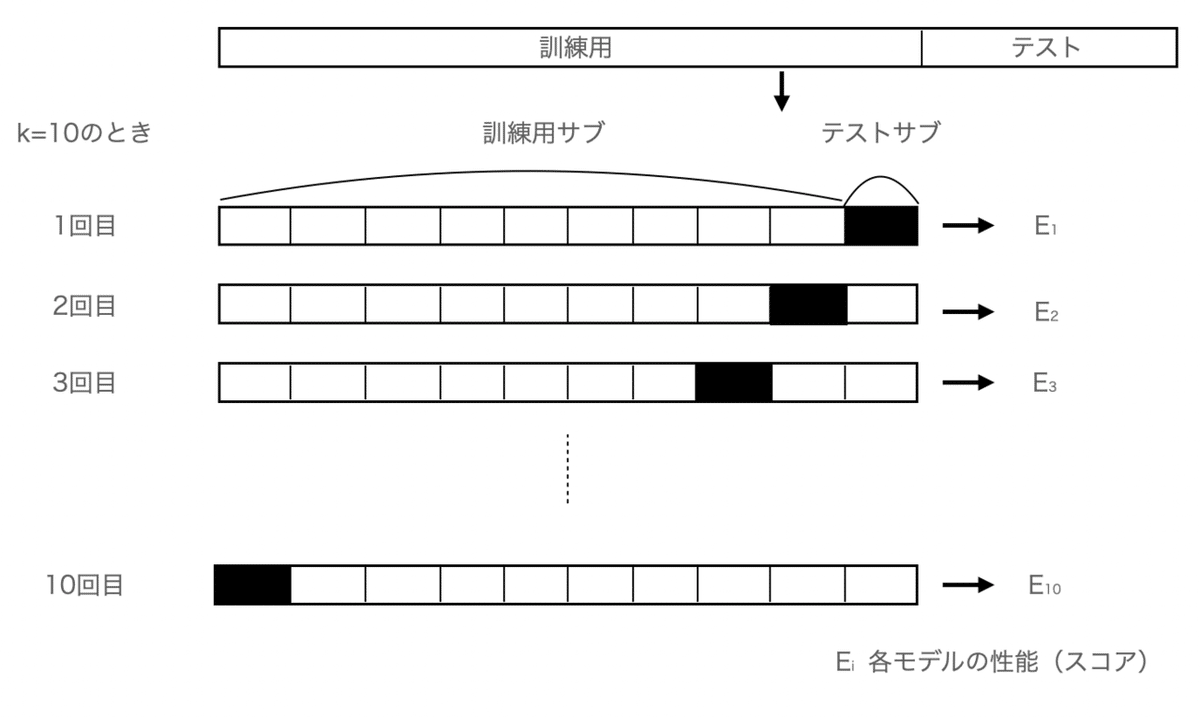
だいたいこれでイメージはつくのかなと思います。
この方法のメリットとしては
・分割して一通り訓練するためデータの偏りを気にする必要がない
・少ないデータセットでもある程度訓練をすることができる
これにより汎化的な(つまり未知データにもうまく適合しやすい)ハイパーパラメータを見つけやすく、
最終的に訓練した後は、未知データ(最初に分割したテストデータ)をどのくらい予測できているのか?を知ることができます。
ちなみに、kの値は一般的には10回だそうですが、データ量があまりにも大きい場合は1回の訓練に時間がかかるため、k=5などに調整することもあります。
何回も訓練できるのだからkをめちゃくちゃ大きくして分割したらいいのにと考えている方もいるかもしれませんが、その場合は各訓練データが似通ってしまう危険性や計算コスト(何回も繰り返して計算する)があり、性能評価のVariance(分散。散らばり具合)が高くなってしまいます。
正解はないため、状況に応じて見てみることが一番です。
・k分割交差検証の実装と注意点~Stratified k-fold cross validation~
さて、実際にpythonでk交差検証を実装していきたいところですが、実はk分割交差検証にもまだ問題点があります。。
気づいた方、いらっしゃいますでしょうか??
すぐ前にkの数値を大きくすると、「各訓練データが似通ってしまう危険性」があるということを示しました。
しかし、これってkの数値が10の時でも起こり得ます。
つまり、10分割したうちの1つに今回ならB(良性)のデータが偏る(Bのサンプル数の方がMより多いため)という危険性があります。
そこでtrain_test_splitのパラメーターのstratifiyを指定したように、k分割する際に、できるだけ元データの比率に合わせて分割したk分割の進化バージョンが層化k分割交差検証(stratified k-fold cross validation)です!
以上を踏まえて、先程のデータを用いてk分割交差検証を行いましょう
まずは、コードから。
import numpy as np
from sklearn.model_selection import StratifiedKFold
# n_splitsで何分割にするのか指定し、.splitのメソッドに trainデータを渡して分割させる
kfold = StratifiedKFold(n_splits=10).split(X_train, y_train)
scores = []
for k, (train, test) in enumerate(kfold):
# まずはk分割したうちの kー1個を取り出し、学習
pipe_lr.fit(X_train[train], y_train[train])
# 残りの 1つの性能の評価
score = pipe_lr.score(X_train[test], y_train[test])
# scoresのリストに格納
scores.append(score)
# k回目にてそれぞれの要素数(0と1の個数カウント)、正解率を表示
print(f'Fold: {k+1} Class distribution: {np.bincount(y_train[train])}, Accuracy: {score:.3f}')
# 最終的なscoreをscoresの要素を平均して算出
print(f'Cross Validation Accuracy is {np.mean(scores) * 100:.2f} +/- {np.std(scores) * 100:.2f} %')
ちなみに、このcodeを書いていて思ったのですが、やはりsklearnはarrayにしておいた方が、楽なんだなと思いましたw(X_train[train]とかの部分。)
では、中身を見てみましょう。
まずは、StratifiedKFoldのドキュメントから。
class sklearn.model_selection.StratifiedKFold(n_splits=5, *, shuffle=False, random_state=None)
実際の引数として、
n_splits: (デフォルトは5)ここで分割したい数値を指定。(最小可能指定数は2)
shuffle: 各クラスを分割前にシャッフルしたいか(デフォルトはFalse)
random_state: shuffle=Trueの時にrandom_seedの指定が可能。
また、Stratifiedの持つメソッドは主に2つ。
get_n_splits([X, y, groups]): n_splitsに指定した数値が返ってくる
split(X, y[, groups]): X, yをk-1:1に分割した時のそれぞれのインデックスが返ってくる
さて、kfoldにより、Stratified KFoldのインスタンスが終われば、あとはforループで回していきます。(ちなみに、kfoldはiteratorのため、forループで回す必要があります。)
for分の中身に関しては、make_pipelineでのコードとあまり変わりませんので割愛します。
ちなみに、具体的な仕組みとして上記のコードは役に立ちますが、もはやsklearnにはワンライナーでさっきの結果を出すことができます(そりゃ、世界のエンジニアはそれくらい一瞬で実装しちゃいます。)
ちなみに、現在調べるとcross_val_scoreよりもcross_validateの方が好まれていたりするそうです。
from sklearn.model_selection import cross_val_score
scores = cross_val_score(estimator=pipe_lr,
X=X_train, y=y_train,
cv=10, n_jobs=1)
print(scores)
print(f'Cross Validation Accuracy is {np.mean(scores) * 100:.2f} +/- {np.std(scores) * 100:.2f} %')
では、見ていきましょう。公式ドキュメントより
sklearn.model_selection.cross_val_score(estimator, X, y=None, *, groups=None, scoring=None, cv=None, n_jobs=None, verbose=0, fit_params=None, pre_dispatch='2*n_jobs', error_score=nan)
estimator: 推定器を指定。指定した推定器でfitメソッドを実行
X: array形式の訓練用データ(説明変数)
y: array形式の訓練用データ(目的変数)
groups: train/testデータの分割の際にラベルを指定
scoring: strを指定することで評価指標を選択できる
cv: 何分割したいか?
n_jobs: いくつの並列処理を行うか?(例えばn_jobs=2のとき、マシンCPUを2つ使って処理を実行できる)
verbose: 数値で指定。何ステップで学習状況を出力するか
fit_params: estimatorのfitメソッドに渡すパラメータ(辞書で指定)
pre_dispatch: 並列処理実行時の起動ジョブ数(デフォルトはn_jobsの2倍)
error_score :‘raise’ or numericで指定。エラー発生時にraiseによりエラー出力したいかどうか。
・学習曲線と検証曲線によるアルゴリズム診断
・学習曲線とは? なぜ使うのか?
機械学習をする際に、懸念事項として過学習があるよね、という話を前回しました。
モデルの自由度や、パラメータが多すぎたりすると、モデルが複雑になりやすく訓練データに過剰に適合しちゃうわけです。
これの対処法として、「データをもっと集める」というアプローチがありますが、収集にかかるコストなども含めるとあまり現実的な対処とはいえません。
ここで、学習曲線を使います。
学習曲線は訓練と検証データのそれぞれの正解率をプロットした線のことですが、これにより、
・モデルのBias やVariance が高いかどうか
・データを追加回収することが良いアプローチといえるかどうか
を判断できます。
2つ目に関して説明すると、学習曲線をみて、データの個数不足により学習ができてないのか、そもそも頭打ちなのか?を判断できます。
実際に一般的な学習曲線の例を3つほど載せます。

真ん中のバイアスが高いということは過小学習が起こっています。つまりパラメータの個数が不足していたりするため、特徴量の生成・追加などが対処法としてあります。
右はバリアンスが高く、過学習をしています。特徴量の個数削減や、正則化による対処などがあります。
・学習曲線をpythonでPlot!
では、今回のデータ(Breast Cancer)で学習曲線の状況を見てみましょう
まずはコードから。
import matplotlib.pyplot as plt
from sklearn.model_selection import learning_curve
%matplotlib inline
# 標準化→ロジスティック回帰を一括で行う
# L2ノルムで正則化する
pipe_lr = make_pipeline(StandardScaler(),
LogisticRegression(penalty='l2', random_state=1, max_iter=10000))
train_sizes, train_scores, test_scores = learning_curve(estimator=pipe_lr, X=X_train, y=y_train,
train_sizes=np.linspace(0.1, 1.0, 10),
cv=10, n_jobs=1)ちょっと長いので、一旦くぎります。
まずは必要なライブラリのインストールの後、今回は前回作ったpipe_lrと違って、PCAを除いて
標準化-> ロジスティック回帰で作ります。
では、みていきましょう。
公式ドキュメントより、
sklearn.model_selection.learning_curve(estimator, X, y, *, groups=None, train_sizes=array([0.1, 0.33, 0.55, 0.78, 1.0]), cv=None, scoring=None, exploit_incremental_learning=False, n_jobs=None, pre_dispatch='all', verbose=0, shuffle=False, random_state=None, error_score=nan, return_times=False, fit_params=None)
基本的にはcross_val_scoreのパラメータと被るため、ある程度割愛します。
exploit_incremental_learning: incremental learning(オンラインで未知データを学習する手法。ざっくりな説明。)を使う時に学習スピードをあげてくれるもの。
return_times: fitとscoreを計算するのにかかった時間を出力するかどうか。
返り値は、
・train_sizes_abs(学習曲線の作成に使ったサンプル数のリスト)
・train_scores & test_scores(それぞれの正解率)
- (return_times = Trueのとき追加で)
・fit_times(学習にかかった時間)
・score_times(スコアリングにかかった時間)
train_sizes_abs(ここではtrain_sizes)をprintしてみると、

それぞれの学習に利用したサンプル数がリスト形式で格納されています。
ちなみに、今回learning_curveの引数train_sizes=np.linspace(0.1, 1.0, 10)としているため、10分割となっています。
では、これらをプロットしてみます。
train_mean = np.mean(train_scores, axis=1)
train_std = np.std(train_scores, axis=1)
test_mean = np.mean(test_scores, axis=1)
test_std = np.std(test_scores, axis=1)
plt.plot(train_sizes, train_mean, color='blue', marker='o', markersize=5, label='Trainig accuracy')
# fill_between でmean - std ~ mean + stdの幅を塗る
plt.fill_between(train_sizes, train_mean + train_std, train_mean - train_std, color='blue', alpha=0.15)
# テストの学習曲線
plt.plot(train_sizes, test_mean, color='green', marker='s', linestyle='--', markersize=5, label='Validation accuracy')
plt.fill_between(train_sizes, test_mean + test_std, test_mean - test_std, color='green', alpha=0.15)
plt.grid()
plt.xlabel('Number of training examples')
plt.ylabel('Accuracy')
plt.legend(loc='lower right')
plt.ylim([0.8, 1.03])
plt.tight_layout()
plt.show()
コード量が多い気もしますが、プロットしているだけに近いので、結果のみ見てみましょう。
訓練データと検証データは250~286付近でお互い良い結果を出しています。
反対にそれ以前は訓練データは常に良い結果を出していますが、検証データとの乖離があり、過学習しているとみて取れます。
---脱線(ロジスティック回帰のパラメータ説明)---
ちなみに、前回割愛しましたが、ロジスティック回帰のパラメータも解説します。
参考サイト(https://data-science.gr.jp/implementation/iml_sklearn_logistic_regression.html)
class sklearn.linear_model.LogisticRegression(penalty='l2', *, dual=False, tol=0.0001, C=1.0, fit_intercept=True, intercept_scaling=1, class_weight=None, random_state=None, solver='lbfgs', max_iter=100, multi_class='auto', verbose=0, warm_start=False, n_jobs=None, l1_ratio=None)
penalty: 正則化の方法(penaltyとは罰則のことで、これにより過学習を防ぐ)を指定します。
種類としてはL2, L1, elasticnet, もしくはNoneを指定します。
デフォルトはL2。正則化などの詳細は一旦割愛します。
dual: 数学における双対問題もしくは主問題をTrue or Falseで定義(この「双対問題」とかは正直意味不明でした。。 かなり本格的な数学理論が必要ぽい)
念のため、興味ある方のためにすこし流し見して参考になりそうなサイトを載せます。
https://manabitimes.jp/math/1186
https://www.acceluniverse.com/blog/developers/2020/09/svm-dual-problem.html
tol: Tolerance(寛容の意味)、つまり、どのくらい小さい値(小数)まで許容するか
C: 正則化するときの逆数パラメータ(次の検証曲線ですこし関わります。)(小さいほど制約を大きくする)
fit_intercept: 切片(バイアス)を最適化するかどうか
intercept_scaling: liblinearという探索法で切片(バイアス)を探索。
class_weight: 辞書型 もしくは ‘balanced’と指定。指定したいクラスラベルを{class_label: weight}で指定することにより重みが調整される(指定なしの場合は1が指定される。)
random_state: solver == ‘sag’, ‘saga’ or ‘liblinear’指定した時のみ使用可能
solver: データセットの個数により、使い分けると効果的(らしい。)
penalty='l2'の場合はデフォルトのlbfgs(1,000以下の小さいデータセットの場合に高パフォーマンスで、高速にトレーニングが可能。)を推奨
max_iter: イテレーションの回数の指定
multi_class: 二クラスもしくは多クラスで分類させるかどうか
verbose: 出力の過程を出すかどうか
warm_start: Trueを選択した場合、すでに学習したモデルに追加して訓練させる
n_jobs: 使うCPUの指定。
l1_ratio:penalty='elasticnet'のとき使用可能。l1_ratio=0でL2正則化と同義。=1でL1正則化。0<l1_ratio<1でL1L2の組み合わせ 。
---脱線おわり---
・検証曲線で過学習・学習不足を検知する
学習曲線のx軸はサンプルサイズでしたが、検証曲線はパラメータの値を変化させることによる訓練データの正解率と検証データの正解率の乖離をみていきます。
コードから先に。
(ちょっと長いですが、後半はほとんど先程と変わらないので、一気に書きます。)
from sklearn.model_selection import validation_curve
# LogisticRegressionのパラメータ Cの変化させたい数値を指定
param_range = [0.001, 0.01, 0.1, 1.0, 10.0, 100]
# validation_curveのestimatorは先程のpipe_lrを使用、
# 返り値はtrain, testの正解率をリストで格納
train_scores, test_scores = validation_curve(estimator=pipe_lr,
X=X_train, y=y_train,
param_range=param_range,
param_name='logisticregression__C', # 「_」は2つ!
cv=10)
train_mean = np.mean(train_scores, axis=1)
train_std = np.std(train_scores, axis=1)
test_mean = np.mean(test_scores, axis=1)
test_std = np.std(test_scores, axis=1)
plt.plot(param_range, train_mean, color='blue', marker='o', markersize=5, label='Trainig accuracy')
# fill_between でmean - std ~ mean + stdの幅を塗る
plt.fill_between(param_range, train_mean + train_std, train_mean - train_std, color='blue', alpha=0.15)
# テストの学習曲線
plt.plot(param_range, test_mean, color='green', marker='s', linestyle='--', markersize=5, label='Validation accuracy')
plt.fill_between(param_range, test_mean + test_std, test_mean - test_std, color='green', alpha=0.15)
plt.grid()
# scaleをlogにすることで幅の調整をする
plt.xscale('log')
plt.xlabel('Parameter C')
plt.ylabel('Accuracy')
plt.legend(loc='lower right')
plt.ylim([0.8, 1.03])
plt.tight_layout()
plt.show()
コードから。
公式ドキュメントより
sklearn.model_selection.validation_curve(estimator, X, y, *, param_name, param_range, groups=None, cv=None, scoring=None, n_jobs=None, pre_dispatch='all', verbose=0, error_score=nan, fit_params=None)
先ほどと同じくある程度かぶっておりますので、特徴のある部分のみ。
・param_name: 変化させたいパラメータの指定
(指定方法は<パイプライン上の処理名>__<パラメータ>、[_(アンダースコアは2つ!)])
・param_range: 評価したいパラメータをarray-like(今回はリスト)で指定
今回は「logisticregression」の「C(正則化するときの係数)」を変化させます。
Cが小さい(制約が大きい)と学習不足になり、その反対は過学習が起きやすくなることが見て取れます。
図より、おおよそC=0.01~0.1くらいが適していそう、と判断できます(個人的には1くらいでもOKと感じます、、、
・グリッドサーチ(Grid Search)をつかってチューニング
グリッドサーチとは簡単にいえば、ハイパーパラメータをとにかく総当たり(しらみつぶし)で見つけていく手法です。
とにかく調べまくる方法ですw。
では、コードから。
from sklearn.model_selection import GridSearchCV
from sklearn.svm import SVC
pipe_svc = make_pipeline(StandardScaler(), SVC(random_state=1))
param_grid = [
{'svc__C': param_range, 'svc__kernel': ['linear']},
{'svc__C': param_range, 'svc__gamma': param_range, 'svc__kernel': ['rbf']}
]
# ハイパーパラメータの値のgridを指定して、
# グリッドサーチを行う
gs = GridSearchCV(estimator=pipe_svc,
param_grid=param_grid,
scoring='accuracy',
cv=10, n_jobs=1, refit=True)
gs = gs.fit(X_train, y_train)
# モデルのベストスコアとその時のパラメータを表示
print(f'Best Score is {gs.best_score_}')
print(f'Best Param is {gs.best_params_}')![]()
(今回、SVM(サポートベクターマシン)という分類器を使ってますが、今回のテーマの話題とはずれてしまうため、そういう分類器がある、くらいで進めます。実際、SVMに関しては別の記事とかで(自分のために)書こうかとも思っています。)
標準化→SVC という流れを一括で行いたいため、make_pipelineで定義
↓
しらみつぶしに見つけていきたいパラメータの名前(今回はSVCのCパラメータ、kernelパラメータ、gammaの組み合わせ)を辞書型で設定しておきます。(複数可)
↓
GridSearchCVでインスタンス化します
さて、公式ドキュメントより
class sklearn.model_selection.GridSearchCV(estimator, param_grid, *, scoring=None, n_jobs=None, refit=True, cv=None, verbose=0, pre_dispatch='2*n_jobs', error_score=nan, return_train_score=False)
ある程度のパラメータはすでに出てきているので、割愛。
param_grid: 調べたいパラメータを辞書で
{'<パイプライン上の処理名>__<パラメータ>: [パラメータ候補のリスト]'}
で定義
refit: ベストパラメータを見つけた後、訓練データを再学習してくれるreturn_train_score: Falseにすると、cv_results_という属性が出力されなくなる。これは各パラメータのスコアや順位などを表示してくれます。(上記コードはFalseのため、Trueにしたものを以下で記載し、cv_results_を見ておきます。)
(return_train_score=True)
from sklearn.model_selection import GridSearchCV
from sklearn.svm import SVC
pipe_svc = make_pipeline(StandardScaler(), SVC(random_state=1))
param_grid = [
{'svc__C': param_range, 'svc__kernel': ['linear']},
{'svc__C': param_range, 'svc__gamma': param_range, 'svc__kernel': ['rbf']}
]
# ハイパーパラメータの値のgridを指定して、
# グリッドサーチを行う
# return_train_score=Trueにしてみる
gs = GridSearchCV(estimator=pipe_svc,
param_grid=param_grid,
scoring='accuracy',
cv=10, n_jobs=1, refit=True,
return_train_score=True)
gs = gs.fit(X_train, y_train)
# cv_results_を表示
print(f'cv_results_ is \n {pd.DataFrame(gs.cv_results_).head()}')
辞書型で出力されますが、公式ドキュメントではDataFrameを推奨されていましたので、先頭5行だけかるく見てみました。
cv=10(10分割でのk分割交差検証)にてそれぞれのscoreが出力され、その時のパラメータなどが表示されています。
今回は使いませんが、より結果以外に他のパラメータの精度もみたい時はTrueにすると見ることができます。
・GridSearchCVのもつメソッド
cv_results_: 前述
best_estimator_: 最適なパラメータが入っている推定器(今回はpipe_svc)が返る
best_score_: 最適なパラメータでk分割交差検証を行った時の正解率の平均値
best_params_: 最適なパラメータの詳細
best_index_: cv_results_の中の最適なパラメータのインデックス番号(refit=Trueのみ利用可)
scorer_: データのスコア評価に使った関数を出力(scoreパラメータの値)
n_splits_: cvパラメータの値
refit_time_: (refit=Trueのときのみ)refitに要した時間
multimetric_: booleanで出力。複数のscorer(評価関数)を定義しているかどうか
では、GridSearchを用いて見つけたパラメータでテストデータのスコアを見てきましょう。
clf = gs.best_estimator_
print(f'Test Accuracy is {clf.score(X_test, y_test):.3f}')![]()
今回、refit=Trueとしているため、gs.fit(~)は不要です。
・GridSearchの欠点とその代用(ランダムサーチ)
グリッドサーチはある意味マシンパワーを使うことで素早くそして総当たり線でハイパーパラメータを見つけてくれますが、それでも計算コストが非常に高いことが欠点となります。
そこで、ランダムサーチという方法があります。
グリッドサーチと違って、交差検証の数を制限可能ですので、探したいパラメータが増えたとしてもある程度の時間の節約ができます。
今回の本題ではないのですが、GridSerchを使ったとき、RandomSearchを使ったとき、iteration=5に制限したとき、で少しだけ見てみましょう
1・GridSerchを使ったとき
%%timeit
# 実際にどのくらい時間が違うのかtimeitで検証
from sklearn.model_selection import GridSearchCV
from sklearn.svm import SVC
pipe_svc = make_pipeline(StandardScaler(), SVC(random_state=1))
param_grid = [
{'svc__C': param_range, 'svc__kernel': ['linear']},
{'svc__C': param_range, 'svc__gamma': param_range, 'svc__kernel': ['rbf']}
]
# ハイパーパラメータの値のgridを指定して、
# グリッドサーチを行う
gs = GridSearchCV(estimator=pipe_svc,
param_grid=param_grid,
scoring='accuracy',
cv=10, n_jobs=1, refit=True)
gs = gs.fit(X_train, y_train)
gs.best_score_![]()
2・RandomSearchを使ったとき
%%timeit
# 実際にどのくらい時間が違うのかtimeitで検証
from sklearn.model_selection import RandomizedSearchCV
from sklearn.svm import SVC
pipe_svc = make_pipeline(StandardScaler(), SVC(random_state=1))
param_distributions = [
{'svc__C': param_range, 'svc__kernel': ['linear']},
{'svc__C': param_range, 'svc__gamma': param_range, 'svc__kernel': ['rbf']}
]
# ハイパーパラメータの値のgridを指定して、
# グリッドサーチを行う
rs = RandomizedSearchCV(estimator=pipe_svc,
param_distributions=param_distributions,
scoring='accuracy', n_iter=10,
cv=10, n_jobs=1, refit=True)
rs = rs.fit(X_train, y_train)
rs.best_score_![]()
3・iteration=5に制限したとき
%%timeit
# 実際にどのくらい時間が違うのかtimeitで検証
from sklearn.model_selection import RandomizedSearchCV
from sklearn.svm import SVC
pipe_svc = make_pipeline(StandardScaler(), SVC(random_state=1))
param_distributions = [
{'svc__C': param_range, 'svc__kernel': ['linear']},
{'svc__C': param_range, 'svc__gamma': param_range, 'svc__kernel': ['rbf']}
]
# ハイパーパラメータの値のgridを指定して、
# グリッドサーチを行う
rs = RandomizedSearchCV(estimator=pipe_svc,
param_distributions=param_distributions,
scoring='accuracy', n_iter=5,
cv=10, n_jobs=1, refit=True)
rs = rs.fit(X_train, y_train)
rs.best_score_![]()
1〜3をみてみると、RandomizedSearchの方が処理速度は早くなり、また、n_iterの数値を制限するとさらに速くなりました。
一応、肝心の精度を見てみましょう。
from sklearn.model_selection import GridSearchCV
from sklearn.svm import SVC
pipe_svc = make_pipeline(StandardScaler(), SVC(random_state=1))
param_grid = [
{'svc__C': param_range, 'svc__kernel': ['linear']},
{'svc__C': param_range, 'svc__gamma': param_range, 'svc__kernel': ['rbf']}
]
# ハイパーパラメータの値のgridを指定して、
# グリッドサーチを行う
gs = GridSearchCV(estimator=pipe_svc,
param_grid=param_grid,
scoring='accuracy',
cv=10, n_jobs=1, refit=True)
gs = gs.fit(X_train, y_train)
print(f'Grid Search Score is {gs.best_score_}')
param_distributions = [
{'svc__C': param_range, 'svc__kernel': ['linear']},
{'svc__C': param_range, 'svc__gamma': param_range, 'svc__kernel': ['rbf']}
]
rs = RandomizedSearchCV(estimator=pipe_svc,
param_distributions=param_distributions,
scoring='accuracy', n_iter=10,
cv=10, n_jobs=1, refit=True)
rs = rs.fit(X_train, y_train)
print(f'Randomized Search Score (n_iter=10) is {rs.best_score_}')
rs = RandomizedSearchCV(estimator=pipe_svc,
param_distributions=param_distributions,
scoring='accuracy', n_iter=5,
cv=10, n_jobs=1, refit=True)
rs = rs.fit(X_train, y_train)
print(f'Randomized Search Score (n_iter=5) is {rs.best_score_}')
さすがに制限回数を減らすと精度は落ちてしまいましたが、n_iter=10では処理速度はより速く、Grid Searchと同精度が出てきました。
一応、Randomized~の方が効率よくパラメータやら精度を出してくれますが、パラメータをしているする際には分布の指定(param_distributions)の理解などが必要(らしいの)で、玄人向けだそうです。
・入れ子式交差検証〜アルゴリズム選択〜
ここまでk分割交差検証やら、グリッドサーチの内容を見てきました。
これらにより、ハイパーパラメータを細かくチューニングする時に組み合わせて使うと効果的でした。
ここでは、「モデル選択ではなく、アルゴリズムを比較したい」時の話をします。
今まではアルゴリズムは決めておいた状態で、ハイパーパラメータをチューニングしたい、という状況に対処してきましたが、アルゴリズムの比較をしたいときもあります。
そこで使えるのが入れ子式交差検証というものです。
簡単にいえば、「お互いが最強な状態でぶつかり合ってどちらが勝つのか?」を検証したい、ということです。
理論を噛み砕くと、
用いるデータセットを訓練データ、テストデータに分割
↓
訓練データをk分割交差検証するため、k分割する
↓
k-1 個のうちでk'分割交差検証を行い、比較したいアルゴリズムの最適なパラメータを選択した上で、残りの1つで検証を行う
具体的にk=5, k'=2の5x2交差検証を図で載せておきます。

・コードで入れ子式交差検証を見てみる
では、今回はSVMと決定木のどちらが良いか選択したい状況を想定して、コードを見ていきましょう。(話を簡単にするために、決定木はmax_depthのみを決めたいとします。)
# SVMで入れ子式交差検証
pipe_svc = make_pipeline(StandardScaler(), SVC(random_state=1))
param_grid = [
{'svc__C': param_range, 'svc__kernel': ['linear']},
{'svc__C': param_range, 'svc__gamma': param_range, 'svc__kernel': ['rbf']}
]
# グリッドサーチはk=2
gs = GridSearchCV(estimator=pipe_svc,
param_grid=param_grid,
scoring='accuracy',
cv=2)
# k=5
svm_scores = cross_val_score(estimator=gs,
X=X_train, y=y_train,
cv=5,
scoring='accuracy')
print(f'SVM Score is {np.mean(svm_scores):.3f} +/- {np.std(svm_scores):.3f}')
# 決定木
from sklearn.tree import DecisionTreeClassifier
# ハイパーパラメータを設定
max_depth = [1, 2, 3, 4, 5, 6, 7, None]
param_grid = {
'max_depth': max_depth
}
clf = DecisionTreeClassifier(random_state=0)
# グリッドサーチはk=2
gs = GridSearchCV(estimator=clf,
param_grid=param_grid,
scoring='accuracy',
cv=2)
# k=5
clf_scores = cross_val_score(estimator=gs,
X=X_train, y=y_train,
cv=5,
scoring='accuracy')
print(f'Decision Tree Classifier Score is {np.mean(clf_scores):.3f} +/- {np.std(clf_scores):.3f}')![]()
コードの中身自体は既に出てきているものなので、詳細は割愛しますが、流れとしては、
GridSearchでk=2でハイパーパラメータを見つける
Cross Validation Score で全体の評価を確認し比較。
今回はSVMの方が優れているという結果がわかりました!
次回は、さまざま性能指標などを使ってアルゴリズムの精度などを確認していきます。
この記事が気に入ったらサポートをしてみませんか?
