AlphaFold2の非専門家向け活用法 第4回後半「AlphaFoldが現状カバーできていない構造情報」(2021年11月4日記事末尾をアップデート)
前回の第4回記事の「AlphaFold2が現状カバーしていないこと、苦手なこと」に基づき、研究をどう進めていくかということについての後編記事です。
当初予定していた複合体構造予測に加え、「新規フォールド」の予測についても述べたいと思います。また、その関係で「After AlphaFoldの構造生物学研究」についての個人的初見については第五回記事にまわる形でと考えています。
ヘテロ複合体構造予測は当たったり外れたり
私が一か月ほどnote更新を放置している間に世の中は進化していまして、現状最強のGoogleColab版AlphaFold2はSergey OvchinnikovさんらによるColabFoldプロジェクトでして、その中でも複合体予測に対応しているのがAlphaFold2_advancedです。長リンカーでつなぐ必要もなしです。関連してプレプリントも出ています。
本家Colab版を超えてる点多数。超スゴイ。
AlphaFold2_advanced、AlphaFold2.ipynbでうまく予測できなかった対象も普通に複合体として出力する。AXXXXBXXXXCのようにPolyGやPolyXリンカーを入れずとも、A/B/Cで予測できるので3つ以上のヘテロ複合体の解析も容易になった。アミノ酸配列の最大値は2500aahttps://t.co/R1qMBPp8rx
— Hiroki Onoda (@onoda_hiroki) August 7, 2021
従来とは異なりjackhmmerを搭載しており、recycles数やturboモード、ensemble数指定(本家は1と8だけ)、そのうえ本家ではデフォルトで出力されない図まで作ってくれるので便利。GUIも見やすくなった。https://t.co/5OgLdntgIU
— Hiroki Onoda (@onoda_hiroki) August 7, 2021
ただ、使ってみるとわかるのですが、ホモオリゴマーについては既に量体数が既知かつ類似構造が既知の場合はかなりいい感じに予測ができる一方、ヘテロ複合体予測については「当たったり、外れたり」という感じです。モノマー構造単体、特にシングルドメインのときのような高い予測レベルには至っていません。元々AlphaFold2の開発者側も予期していなかった使い方ということで、改善の余地があると思われます。
うまくいった例
AlphaFold 2 prediction of the (TIM9:TIM10)3 complex and comparison to the crystal structure https://t.co/4A4HSf2qKQ. Too good to be true? https://t.co/Btmbplwuq5 pic.twitter.com/XGgYz2itaC
— Kresten Lindorff-Larsen (@LindorffLarsen) August 11, 2021
うまくいかなかった例
既知の複合体構造なので当たって然るべきだが、当たらなかった #AlphaFold 2 複合体疑似ドッキングの例。
— 叢雲くすり(創薬ちゃん) (@souyakuchan) July 20, 2021
マゼンタ: 正しい ACE2 - Spike RBD 複合体
黄色: AlphaFold 2 による Spike RBD 予測位置
シアン: (GGGS)₉ リンカー
リンカーが短かったか?
各モノマーの構造のアサイン自体は合っている pic.twitter.com/v77un5X41l
私自身が試してみた中でわかりやすい例はGFPと抗GFP抗体の複合体構造でしょうか。私の研究室で以前に構造解析を行ったGFPと抗GFPナノボディ抗体複合体の構造についてGoogleColab版AlphaFold2(AlphaFold2_advanced)で構造予測した結果です。

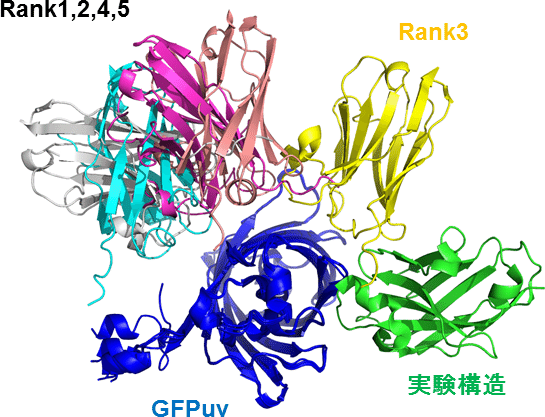
GFPuv-LaG16抗体の複合体構造重ね合わせ図
抗体-抗原相互作用に関しては進化情報がほとんど効いてこないはずで、外れるのが当たり前といえば当たり前なのですが、5つの予測構造すべて外れていました。怖い点としては、「外れている予測構造のほうが実験構造よりも抗原(GFP)により密に相互作用しており、一見すると正解のようにみえる」という点です。
また、興味深い点として、他の予測とは1つだけ傾向が異なり、Predicted aligned error(PAE)が明らかによくなっていたRank3の予測構造については、他の構造とは異なる位置、実験構造により近い位置に結合予測を示していました。将来的には抗体-抗原相互作用についてもAlphaFoldベースでより良い手法が出てくるかもしれません。いずれにせよ、AlphaFold2のヘテロ複合体予測について現状では、そこまで高い信頼度と言えず、かなり注意が必要でしょう。使用にあたっては、PAEの高低とにらめっこしつつ、結合界面の変異体解析を丁寧にやる等の慎重さが必須と思われます。
新規フォールドの予測は実はかなりいけてそう
「AlphaFoldが現状カバーできていない構造情報」という記事タイトルに反しますが、ヘテロ複合体の構造予測と異なり、新規フォールド、それも単一ドメインの構造予測については、AlphaFold2はかなり良いポテンシャルがあると私は考えています。実際、最近構造が報告されたTTYH(新規フォールド)については、ほぼドンピシャで当たってます。
side note: like many structural biologists recently, i've enjoyed played with AF2/ColabFold, and was very impressed by the AF2 prediction as well as the ColabFold dimer prediction! we also used the AF2 prediction to help model the N-terminus pic.twitter.com/vp2QclahrC
— Christopher Hoel (@chrismhoel) August 16, 2021
私のほうの手持ちの新規構造でも同様にかなり当たっているものがあり、新規フォールドかつ単一ドメインの場合、主鎖レベルではかなり高い質の高い構造予測を期待してもいいのではないかと考えています。新規フォールドの構造予測はこれまで非常に難しかったことを考えると、これは非常に大きなブレークスルーといえるでしょう。
微妙かつ重要な点の予測はまだまだ苦手?
その一方、構造の中の細かい部分、つまり側鎖の向きといったような話ですが、この部分についてはまだまだ気を付けたほうが良さそうです。東京大学総合文化研究科の加藤英明先生が最近プレプリント発表された微生物ロドプシンChRmine(オプトジェネティクスのツールとして有用)の構造では、 機能的に重要なall-trans 型レチナール(ATR)のシッフ塩基プロトンのカウンターイオンとして働く、機能的に重要なアスパラギン酸(D115)部位の構造が予測できていませんでした。この位置のカルボン酸(アスパラギン酸)は微生物型ロドプシンで高度に保存されており、ChRmine以外は全部同じ方向を向いているのですが、AF2の予測構造ではそれに引きずられたのか実際の構造とは異なり他の微生物型ロドプシンと同じ向きを向いていました。メカニズムを議論する上でこれはかなり大きな問題となります。また、このカルボン酸のpLDDT scoreは90以上と非常に高く、本来はかなり信頼して良い部位です。ですので、pLDDT scoreが高いからといって必ずしもAF2の予測構造を正しい前提で研究を進めるのは危ういと言うことも意味しています。
Notably, the unique structure of ECL1, including the putative proton acceptor D115, could not be correctly predicted by AlphaFold2. (8/n) pic.twitter.com/8VXEWYhQ6j
— Hideaki E. Kato (@emeKato) August 16, 2021
未知の新規フォールドの構造を主鎖レベルではかなり良好に予測でき、AlphaFold2が非常に有用であることは確かです。その一方、既に多数の類似構造がある微生物ロドプシンのケースでも、機能的に非常に重要な残基について適切な予測が出来なかったというのはAlphaFold2を使うにあたっての大切なメッセージと思われます。有用な点、注意が必要点、それぞれに留意して利用することが重要でしょう。
実際、上記の例以外にも、例えば第一回記事で扱ったbeta2アドレナリン受容体の場合、そのICL2領域がいわゆるinactive状態ではunfoldしているのですが、active状態ではヘリックス構造を形成することがが知られていますが、AlphaFold2の予測構造では、全体構造としてはinactive状態であるにも関わらず、ICL2だけactive状態というnon-physiologicalなキメラ構造になっています。
以上です。今回は自分自身がAlphaFold2を使ったケースというよりは、他の人がトライしたケースの紹介がメインになってしまいましたが、それほどAlphaFold2が広く使われているということでご容赦ください。次回は、「After AlphaFoldの構造生物学研究」についての個人的初見については最終回(第五回)記事として記したいと思います。
DeepMind公式も複合体構造予測をサポートするように(2021年11月4日記事更新分)
The #AlphaFold source code has been updated and now accounts for multi-chain protein complexes - providing a significant improvement in accuracy for predicting protein interactions: https://t.co/4gyZ0loLrd
— DeepMind (@DeepMind) November 2, 2021
Generate predictions from your browser via: https://t.co/2Vd4itjElu pic.twitter.com/Db66KcXCRD
DeepMind公式も複合体構造予測をサポートするようになったので、上述のGFP-抗体複合体の構造予測をこちらでも試してみました。

GFPuv-LaG16抗体の複合体構造重ね合わせ図
が、DeepMind公式のほうでもあまり思わしい結果ではありませんでした。抗体-抗原相互作用に関しては進化情報がほとんど効いてこないはずで、外れるのが当たり前ではあるのですが、複合体構造予測についてはこのあたりの得意不得意によく注意して使う必要が依然としてありそうです。
この記事が気に入ったらサポートをしてみませんか?
