
【R言語】irisのデータを使って正規性の検定をしてみた
こんにちは。プログラミング超初心者のえいこです。
前回の記事で、パラメトリックとノンパラメトリックの違いについて勉強しました。
パラメトリックとノンパラメトリックの検定を使い分けるためには、データの正規性を確認しておいた方が良い。
ということで、今回はRの"iris"データを使ってデータの正規性を確認してみようと思います。
Rを使ったデータの正規性の確認方法は、こちらのブログを参考にしました。
こちらのブログはめちゃくちゃニッチだけど、知っておきたいことがたくさん載っているので、これからたくさんお世話になりそうです。
正規性を確認する検定がある
さて、参照したブログによると、Rで正規性を確認するには、Shapiro-Wilk test (シャピロ・ウィルクの検定)を使うと書いてありました。
実は他にも正規性を確認する検定はあるようです。
Rでできる正規性の検定
・シャピロ・ウィルク検定
データが正規母集団に由来するという帰無仮説を検定する。
・コルモゴロフ・スミルノフ検定
得られた2つのデータ間の確率分布の相違の検定、1データにおける確率分布の正規性の検定。データが正規分布に従うという帰無仮説について検定する。
気をつけておきたいことは、どちらの検定も帰無仮説が「正規分布している」なので、棄却されなかったからといって正規分布を積極的に証明しているわけではないということです。
Rでシャピロ・ウィルク検定してみる
まずは、シャピロ・ウィルク検定をしてみます。
今回は例として、irisのsetosaのSepal.Lengthについて検定してみようと思います。
まずは、データセットを用意して、"shapiro.test()"で検定するデータを入れたらOK。
library(dplyr) #dplyrを呼び出す
data<-iris #dataにirisを入れる
data_setosa<- #irisのなかのsetosaのデータだけ抽出した表を作る
data %>%
group_by(Species) %>%
dplyr::filter(Species=="setosa")
shapiro.test(data_setosa$Sepal.Length) #シャピロ・ウィルク検定 果はこのように表示されます。
Shapiro-Wilk normality test
data: data_setosa$Sepal.Length
W = 0.9777, p-value = 0.4595p=0.4595と帰無仮説は棄却されなかったので、正規分布と言っても良いでしょう。
Rのコードでサクッとヒストグラムを描いてみます。
hist(data_setosa$Sepal.Length)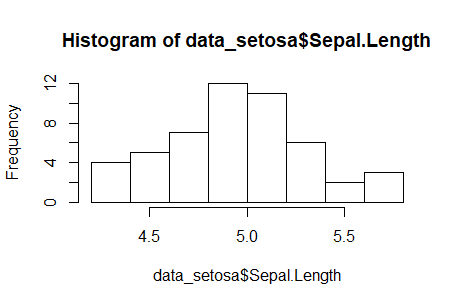
なんとなく、正規分布っぽいですね。
複数のデータを一気にシャピロ・ウィルク検定をする
でも複数サンプルについて一つ一つリストを作るのはめんどくさいですよね。
irisには3種類のアヤメのデータが入っているので、一度の検定したいものです。
複数のデータを一気に検定するときには、"tapply()"を使います。
tapply(data$Sepal.Length, data$Species, shapiro.test)実行すると...
$setosa
Shapiro-Wilk normality test
data: X[[i]]
W = 0.9777, p-value = 0.4595
$versicolor
Shapiro-Wilk normality test
data: X[[i]]
W = 0.97784, p-value = 0.4647
$virginica
Shapiro-Wilk normality test
data: X[[i]]
W = 0.97118, p-value = 0.2583setosa, versicolor, virginicaの3サンプルのSepal.Lengthについて正規性の検定ができました。
いずれも、p-value = 0.4595, p-value = 0.4647, p-value = 0.2583とどのサンプルでも帰無仮説が棄却されなかったので、正規性があると判断できます。
先ほどと同様にヒストグラムを描いてましょう。
この時もtapplyを使ってヒストグラムを描いて、"layout"で1行3列で並べます。
layout(t(1:3))
tapply(data$Sepal.Length, data$Species, hist)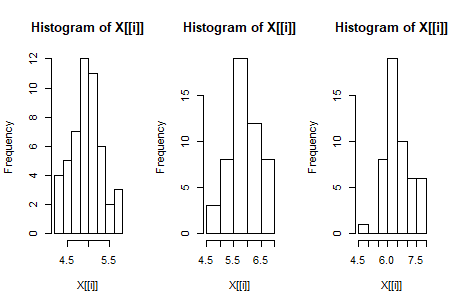
左側から、setosa, versicolor, virginicaのヒストグラムです。どれもなんとなーく正規分布っぽいですね。
ということは、パラメトリック検定を採用してよさそうです。
Rでコルモゴロフ・スミルノフ検定してみる
もう一つのコルモゴロフ・スミルノフ検定もしてみます。
コルモゴロフ・スミルノフ検定のコマンドは、"ks.test()"でyの引数をpnormを指定することで1標本の正規性を検定できるようです。平均値と不偏分散が必要なようです。
ks.test(x=data$Sepal.Length,y="pnorm",
mean=mean(data$Sepal.Length),
sd=(data$Sepal.Length))実行すると...
One-sample Kolmogorov-Smirnov test
data: data$Sepal.Length
D = 0.38109, p-value < 2.2e-16
alternative hypothesis: two-sided
警告メッセージ:
ks.test(x = data$Sepal.Length, y = "pnorm", mean = mean(data$Sepal.Length), で:
コルモゴロフ・スミノフ検定において、タイは現れるべきではありません 警告メッセージが出てきました。
「コルモゴロフ・スミノフ検定において、タイは現れるべきではありません 」
調べてみると、こちらのサイトでこんな解説がされていました。
こちらのエラーは同じ値が複数回出てくるため出たようです。コルモゴロフ・スミノフ検定は連続分布を仮定しているのため、離散分布のデータが与えられると正しくp値ができないことを示しています。
Q-Qプロットを描いてみる
最後に、Q-Qプロット(Quantile-quantile plot)を描いて正規性の確認をしてみようと思います。
Q-Qプロットは横軸に理論的な分位数(クォンタイル)、縦軸に実際の値としてプロットします。対角線上にプロットが並べば正規分布だと考えられます。
RでQ-Qプロットを描く関数はqqnorm。Q-Qプロットにもいくつか種類があるようですが、正規性をぱっと見で判断するにはnormalなQ-Qプロットでよさそうです。
また、"qqline"で理論的な分布に一致する場合の線を描いておくとより分布がわかりやすいとのことだったので線も追記しています。
qqnorm(data_setosa$Sepal.Length)
qqline(data_setosa$Sepal.Length)このコードを実行すると、
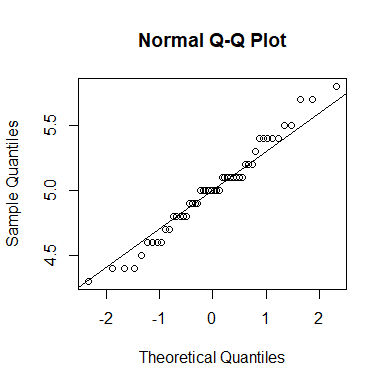
右上は線から離れてしまっている感じがしますが、なんとなく直線っぽくなっていますね。
複数のデータのQ-Qプロットも一気に描画できます。
layout(t(1:3))
tapply(data$Sepal.Length, data$Species, qqnorm)を実行すると、
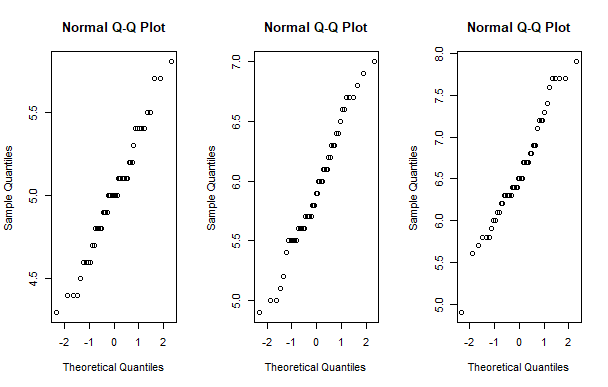
こんな感じで左からsetosa, versicolor, virginicaのヒストグラムが出てきます。
コンソール画面には、
> layout(t(1:3))
> tapply(data$Sepal.Length, data$Species, qqnorm)
$setosa
$setosa$x
[1] 0.17637416 -0.43991317 -0.87789630 -1.22652812 -0.22754498 0.87789630
[7] -1.12639113 -0.17637416 -1.88079361 -0.38532047 0.95416525 -0.73884685といった、数字がたくさん出てきます。
理論値の直線も引いてみようとしたのですが、あまりうまくいかず...
layout(t(1:3))
tapply(data$Sepal.Length, data$Species, qqnorm)
tapply(data$Sepal.Length, data$Species, qqline)というコードを書いてみたのですが、コンソール画面に
> tapply(data$Sepal.Length, data$Species, qqline)
$setosa
NULL
$versicolor
NULL
$virginica
NULLと出てきて、グラフはこんな感じに
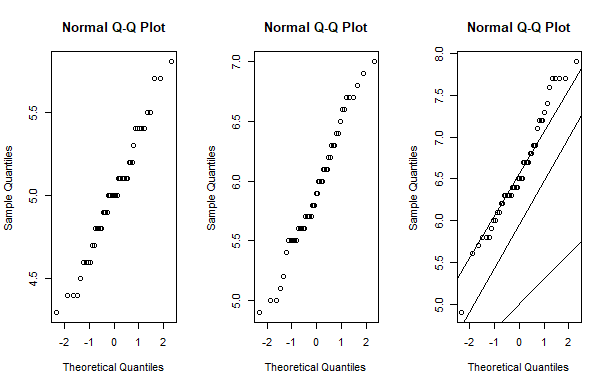
なんかもう少し工夫すればうまくいきそうな気がするんですが..
今回はRで正規性を確認することをしてきました。
具体的な検定をする前に、一応で良いのでデータの正規性を確認できると論文での統計処理で突っ込まれても対応できそうです。
次回は、Rでサンプルサイズを計算してみようと思います。
それでは、また!
最後までお読みいただきありがとうございます。よろしければ「スキ」していただけると嬉しいです。 いただいたサポートはNGS解析をするための個人用Macを買うのに使いたいと思います。これからもRの勉強過程やワーママ研究者目線のリアルな現実を発信していきます。
